서 언
재료 및 방법
보리 유전자원 보존현황
형태적 다양성 및 상관성 분석
결 과
종, 원산지에 따른 유전자원 다양성
종과 원산지에 따른 형태적 형질 다양성 관계
보리 유전자원의 대표성과 특이성
특성평가 항목 간의 상관관계
고 찰
적 요
서 언
보리는 왕겨가 쌀처럼 잘 벗겨져 식용으로 사용되는 쌀보리(naked barley), 왕겨가 붙어 사료로 많이 이용되는 겉보리(Hulled barley)로 나눌 수 있다. 쌀보리는 아밀로스와 아밀로펙틴의 함량에 따라 찰보리(glutinous barley/waxy barley)와 메보리(non-waxy barley)로 다시 구분되는데, 특히 찰보리는 높은 아밀로펙틴 함량으로 식용에 주로 이용되고 있다(Jo et al., 2013). 보리는 과거 쌀 다음으로 소비되는 곡실이었으나, 쌀 소비량의 증가로 인하여 점차적으로 소비량과 생산량이 감소하게 되었고, 2012년에는 보리감축대책으로 보리 수매제를 폐지하기도 하였다(Kim et al., 2012). 그런데 최근에는 보리의 건강 기능성이 부각되면서 베타글루칸, 폴리페놀, 안토시아닌 등을 이용한 건강식품의 소재로 이용되고 있고, 건강식 혼반, 새싹보리, 검정보리 음료, 수제 맥주용 등 다양한 먹거리로 활용됨으로써 수매제 폐지 이후 2019년에는 재배면적과 생산량이 2배 가까이 증가하였다(Kim et al., 2020b). 보리에 대한 소비량이 증가함에 따라 다양한 보리 신품종이 개발되고 있는데, 효소 역가가 높아 엿기름으로 이용되는 혜양, 혜강, 혜다와 기능성 안토시아닌 함량이 많은 겉보리 흑다향, 보리차용으로 이용되는 흑수정찰 등 기능성을 중심으로 꾸준히 개발되고 있다(Kim et al., 2020b). 또한 지구온난화에 따른 기후변화의 대응으로 파종 시기 및 재배방법 개발(Kim et al., 2013), 병 저항성 강화 등과 같이 보리의 생장능력과 생산성 향상을 위한 다양한 육종연구도 함께 진행되고 있다(Kim et al., 2020a, Park et al., 2006). 이러한 다양한 연구와 미래 식량안보를 위해서는 보리 특성의 다양성을 관리하고 보존해야 할 필요가 있기에, 국립농업과학원 농업유전자원센터(National Agrobiodiversity Center, National Institute of Agricultural Sciences, The Republic of Korea)에서는 국내 국적미상, 에티오피아, 중국, 터키 등을 포함한 해외 102개국에서 수집, 기탁받은 약 2만5천여 자원(21년 기준)의 보리 유전자원을 보존하고 있으며, 이 밖에 식량, 원예, 특용 작물에 대한 다수 자원을 관리하고 있다(Bae et al., 2021; Kim et al., 2022). 그리고 씨앗은행(https://genebank.rda.go. kr)을 통해 보존하고 있는 보리 유전자원의 특성평가 정보를 제공하고 있으며, 신품종, 육종기술 개발 등과 같은 농업연구를 목적으로 20~50립 정도의 종자를 무료로 분양하고 있다.
작물을 포함한 식물은 서식 환경과 유전적 요인에 따라서 변이가 나타나고, 환경에 대한 적응하는 속도가 같은 종 내에서도 차이가 있기 때문에 형태적으로 또는 유전적으로 다양한 형질이 나타난다. 그러므로 유전자원 형질에 대한 다양성 연구는 자원의 특성을 이해하고 유용한 육종소재를 발굴하고 보존하며, 장기적으로 식량안보를 위하여 자원을 활용하고 관리하기 위한 기초자료로 이용되는 중요한 연구 중 하나이다(Jarvis et al., 2010). 일 예로 형질에 대한 다양성 연구결과는 종, 원산지의 추정과 함께 생물의 생육환경의 이해와 계통분류, 진화 등의 연구, 교배를 통한 전통육종과 유전자, DNA 정보를 이용한 분자육종, 신품종 개발 등과 같은 다양한 농업연구분야로 확대할 수 있는 자료로 활용할 수 있다(Cho et al., 2009; Rommens, 2007; Tolbert et al., 1979). 따라서 보리 유전자원의 보존 및 활용을 위한 정보의 축적, 기능성 대사산물 생산을 위한 유용 소재발굴, 기후변화에 대응한 안정적 생산과 공급, 다양한 품종 개발, 작물 육종기술 연구를 추진하기 위해서는 자원의 형질에 대한 다양성 연구가 선행되어야 하며, 이를 위해서는 종과 원산지에 따른 형질의 차이를 확인하고 유사한 자원을 군집화(Clustering)하여 집단 간의 대표적, 특이적 형질을 분석하며 각 형질 간의 상호관계 분석과 같은 연구결과가 필요하다.
본 연구는 농업유전자원센터에서 보존하고 있는 보리 유전자원에 대하여 특성평가 정보를 바탕으로 형질의 다양성을 분석함으로써 내병성, 고기능성, 경관용 등과 같은 기능성 육종소재 발굴 및 신품종 개발 등 유전자원의 광범위한 활용의 근간을 마련하고자 하였다. 종과 원산지 수준에서 자원 개체에 대한 다양성을 분석하고자 작물 유효수(Effective number of crop species, ENCS), 섀넌 종 다양도 지수(Shannon-Wiener index), 종 풍부도(Species richness), 종 균등도(Species evenness) 등을 사용하였고, 자원의 형태적 형질의 차이가 종과 원산지의 다양성과 관련이 있는 점을 고려하여(Ehrlich and Wilson, 1991) 보리 유전자원의 각 특성평가 항목이 종과 원산지의 차이와의 관련성을 검정함으로써, 종과 원산지에 따라 형질의 다양성을 추론하였다. 그리고 특성평가 항목에 따라 자원을 분류하여 형질에 따른 자원의 유사성과 대표성을 분석하고 형질 간의 상관관계를 통계적으로 분석하여 보존된 자원의 특성에 대한 다양성을 고찰하였다. 본 연구에서 수행한 보리 유전자원의 형질 다양성 및 특성 분석에 대한 연구는 유전자원 다양성 확보를 위한 수집과 보존 목적의 근거를 마련하고, 자원의 대표적, 특이적 특성평가 정보를 통해 다양한 유용 형질 발굴 및 신품종 개발을 위한 육종연구의 기초정보로 활용할 수 있을 것으로 기대한다.
재료 및 방법
보리 유전자원 보존현황
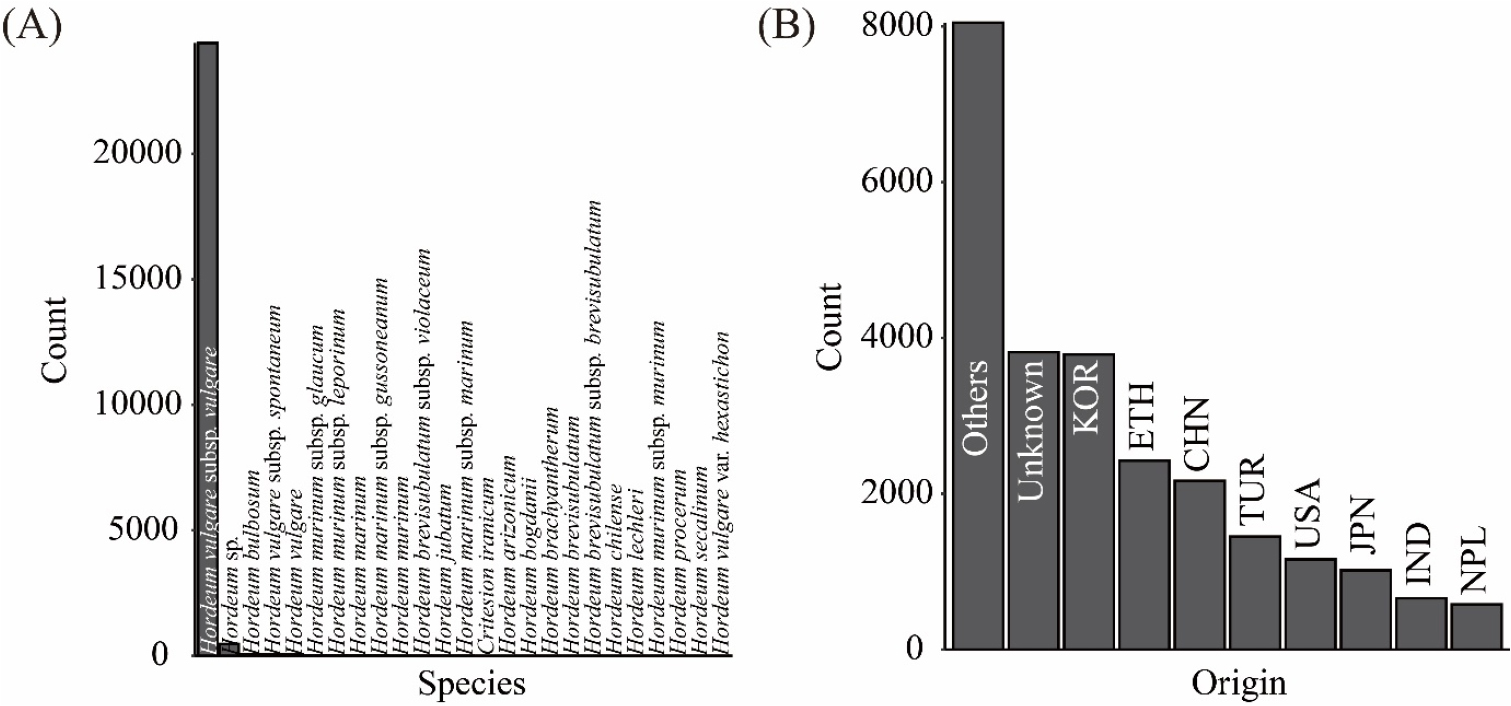
농업유전자원센터에서 보존 중인 보리 유전자원은 특성 분석이 필요한임시자원을 제외한 관리자원 가운데 1900년부터 2021년까지 수집 및 조사된 총 25 종(Species), 25,104 자원을 대상으로 분석하였다. 이 들 보리 유전자원은 야생종, 재배종, 육종자원을 다양하게 포함하고 있다. 이 들 유전자원은 종에 있어서 Hordeum vulgare subsp. vulgare가 97.2% (24,412 자원)로 대부분을 차지하고 있는데(Fig. 1A, Supplementary Table 1), 이는 식량으로써 보리의 대표적 종으로 재배되어 왔기 때문에 해당 종에 기반한 종자생산 및 보존, 육종기술의 발전 등으로 인하여 H. vulgare subsp. vulgare가 다른 종에 비해 많이 수집되었을 것으로 추정된다(Jakob et al., 2014). 반면에, 원산지(Origin)는 국내 뿐만 아니라 에티오피 (ETH), 중국(CHN), 터키(TUR) 등, 해외 100개국과 국적 미상의 자원(Unknown origin)도 포함되어 있는데, 특히 국적 미상의 자원을 제외하면 국내자원이 3,787자원(15.1%)로 가장 많고 그 외 원산지의 자원도 다수 보존되어 있다(Fig. 1B, Supplementary Table 2). 종과 원산지는 카이제곱 검정(Chi-square test)에서 종과 원산지의 차이가 서로 유의적 관련이 있음(P-value < 0.05)을 추정할 수 있다.
농업유전자원센터에서는 보리 유전자원을 장기적으로 보존함과 동시에, 신품종 개발, 육종연구 등 연구목적의 분양에 있어서 보리 유전자원의 식물체 생장습성(Growth habit), 지엽 직립성 정도(Flag leaf attitude), 출수기(Heading date), 간장(Culm length), 성숙기(Maturing date), 이삭조성(Spike type), 이삭길이(Spike length), 이삭 직립정도(Spike erectness), 등과 같은 기초형질조사 항목(Lee et al., 2019)과 함께 종실피과성(Grain type), 병와성(Normal and uzu type), 망활(Rough/ Smooth Awn), 이삭모양(Spike shape), 한해(Cold damage), 도복(Lodging), 파성(Vernalization degree), 천립중(1000 grain weight) 등의 형태적 형질과, 흰가루병(Powdery mildew), 줄무늬병(Leaf stripe disease), 보리누른모자이크병(Barley yellow mosaic virus), 깜부기병(Loose smut) 발병, 조단백함량(Crude protein)과 같은 생리적 형질(Physiological traits)에 대한 특성평가 정보를 추가하여 국립농업과학원 농업유전자원센터에서 운영하고 있는 씨앗은행 홈페이지 및 농업유전자원관리시스템(Genebank Management System)에서 서비스하고 있으며, 본 연구에서는 형태적 형질과 생리적 형질을 함께 다양성 분석에 이용하였다(Supplementary Table 3). 이러한 특성평가 정보는 간장, 이삭길이, 천립중 등과 같은 양적형질과 식물체 생장습성, 이삭조성, 이삭 직립정도 등과 같은 질적형질로서, 시계열, 연속형, 이산형과 같은 수치형 자료와 코드화로 구분된 범주형 자료로 제공한다. 본 연구에서는 작물의 증식과정에서 발생할 수 있는 인위적, 자연적 영향으로 인하여 시기의 차이가 나타날 수 있는 파종기(Sowing date), 정식기(Planting date), 출수기, 성숙기와 같은 시계열 정보와 정보가 부정확한 항목은 제외하고, 형태적 형질과 함께 생리적 형질을 포함하여 총 20항목을 대상으로 분석하였다. 또한 각 평가항목에서 세부적으로 분류된 기준인 ‘기타’와 ‘혼립’은 조사자가 자원의 특성을 구분하기가 모호한 경우에 기록된 사례가 많으므로 보다 명확한 정보 사용과 분석 결과의 신뢰성을 위하여 본 연구에서는 제외하였다.
형태적 다양성 및 상관성 분석
보리 유전자원의 종과 원산지에 대한 다양성 분석은 보유한 자원의 종 및 원산지 수(s)와 개체수(n)를 바탕으로 다양성 확보 및 증대를 목적으로 하는 자원 수집의 척도로 활용할 수 있다. 본 연구에서는 종과 원산지 다양성 분석을 위하여 생물, 생태계 등 다양한 분야에서 활용되고 있는 섀넌 종 다양도 지수(식 1) 종 풍부도(식 2), 종 균등도(식 3), 작물 유효수(식 4)를 사용하였다(Aguilar et al., 2015; Yu and Yoo, 2015). 참고로 작물 유효수는 작물 종 다양성에 대한 지수로 해석할 수 있기 때문에 본 연구의 다양성 분석에 활용하였다(Aguilar et al., 2015).
유전자원의 특성평가 항목에 기반한 자원의 대표성과 특이성은 자원을 특성의 유사성에 따라 세부 집단으로 분류함으로써 분석할 수 있는데(Kang and Kim, 2006), 분류된 군집이 나타내는 공통된 특성과 각 군집에 대한 특이적 특성을 추정할 수 있다. 그런데 자원을 그룹화함에 있어서 사용하는 특성평가 항목 가운데, 수치형 자료는 양적형질이 대부분으로 환경적 영향(기후, 토양, 인간 등의 요인)에 따라 조사된 값에 대한 오차가 크고 필요에 따라 결측치(Null value)를 보완해야 하는 문제가 있다(Rawal et al., 2017), 따라서 본 연구에서는 범주형 특성평가 항목을 대상으로 cacGMS (Clustering Analysis for Categorical genetic traits of germplasms in Genebank Management System) 알고리즘을 이용하여, 자원 와 간에 일치하는 특성평가 항목()에 대한 비율로 계산(식 5)한 유사도()를 거리행렬(, 식 6)로 변환하여 전체 자원을 군집화하고 각 군집의 특성과 다양성을 분석하였다(Yu et al., 2022).
변환된 거리행렬을 바탕으로 cacGMS는 Ward2 방법을 이용한 계층적 군집 알고리즘(Murtagh and Legendre, 2011)과 대표객체 기반 알고리즘의 K-medoids (Park and Jun, 2009)을 사용하고, 각 알고리즘에서 필요한 자원의 군집 수는 임의로 지정한 범위(최소 6개, 최대 핵심집단 수)에서 평균 실루엣 지수(Average Silhouette Index)가 가장 높은 군집 수를 선택하였다. 각 알고리즘에 의한 군집은 Newman’s modularity 방법(Newman, 2006)으로 통합하고, PowerCore (Kim et al., 2007)에 의해 결정된 핵심집단을 적용함으로써 자원의 군집을 최종적으로 결정하였다(Fig. 2). 참고로 PowerCore는 특성평가 항목의 자료와 SSR (Simple sequence repeat), SNP (Single nucleotide polymorphism)과 같은 유전자형 정보를 이용하여 핵심집단을 선발할 수 있는데, 사용자가 목적하는 연구와 자료에 따라서 선택적으로 사용할 수 있다. 본 연구에서는 형태적 다양성을 분석하기 위한 연구목적에 따라 범주형 특성평가 정보를 대상으로 핵심집단을 선발하였다.
자원의 세부적인 특성은 각각 독립적으로 작용하기도 하지만, 각 특성 간의 상호작용에 의해 결정되기도 한다. 따라서 각 자원의 특성에 대한 상관관계를 확인하기 위하여 수치형과 범주형을 포함한 모든 보리의 특성평가 항목의 상관관계를 분석하였다. 특성평가 항목의 자료형에 따라서 수치형-수치형 관계는 Pearson Correlation Coefficient, 수치형-범주형(혹은 범주형-수치형)은 Polyserial correlation coefficient, 그리고 범주형-범주형 관계는 Cramer’s V coefficient방법으로 R-project 라이브러리를 사용하였다(Benesty et al., 2009; Dessau and Pipper, 2008; Olsson et al., 1982; Prematunga, 2012). 그리고 모든 상관계수(correlation coefficient)는 절대값을 적용하여 계수의 범위를 0에서 1사이로 통일하였다.
결 과
종, 원산지에 따른 유전자원 다양성
보리 유전자원에 대한 종 다양성을 분석했을 때, 종 다양도 지수는 0.16, 종 풍부도는 2.37, 종 균등도는 0.05로 나타나 원산지와 비교했을 때 매우 낮게 나타났으며, 작물 다양성을 의미하는 작물 유효수도 1.17로 낮은 수준을 보였다(Table 1). 이는 Fig. 1에서 언급한 바와 같이 종에 있어서 H. vulgare subsp. vulgare가 전체 자원의 97 %를 차지하고 있고 있는 반면, 원산지는 특정 국가에 편중되지 않은 결과라 볼 수 있다.
Table 1.
Diversity of barley's germplasm
| Type | Species | Origin |
| Species/Origin | 25 | 102 |
| Shannon-Wiener index | 0.16 | 3.21 |
| Species richness (Margalef) | 2.37 | 9.97 |
| Species evenness | 0.05 | 0.69 |
| ENCS | 1.17 | 24.73 |
종과 원산지에 따른 형태적 형질 다양성 관계
종과 원산지의 차이와 형태적 형질 다양성 관계를 추정하기 위하여, 총 20개의 특성평가 항목에 대하여 분석하였다. 종에 대해서는 조단백함량, 병와성, 망활, 이삭모양, 파성, 흰가루병, 줄무늬병, 보리누른모자이크병, 깜부기병을 제외한 11개 항목, 원산지에 대해서는 조단백함량과 파성을 제외한 18개 항목이 유의적인 차이(P-value < 0.05)를 보임으로써, 종과 원산지에 대한 생물학적, 지리·환경적 분류가 다수의 특성에 대한 차이와 관련이 있음을 추정할 수 있다(Table 2). 검정되지 않은 조단백함량, 파성, 흰가루병, 줄무늬병, 깜부기병은 자원이 H. vulgare subsp. vulgare에서 집중되었거나 특정 원산지에서만 조사되었기 때문인 것으로 알 수 있었다. 종에 대하여 유의적 차이가 없는 것으로 나타난 망활과 이삭모양, 보리누른모자이크병 발병경률은 원산지에 대하여 차이가 있는 것과 같이 서로 다른 결과를 보이고 있는데, 종과 원산지와의 유의적 상관관계를 고려했을 때 종에 대하여 상관관계가 있을 것으로 추정할 수 있다.
Table 2.
Statistic test for genetic traits with species and origin
| Genetic traits | Germplasms | Method |
Species (P-valuez) |
Origin (P-valuez) |
| Culm length | 24,912 | Kruskal-Wallis | *** | *** |
| Spike length | 24,169 | Kruskal-Wallis | *** | *** |
| 1000 grain weight | 5,363 | Kruskal-Wallis | *** | *** |
| No. of grains per spike | 2,440 | Kruskal-Wallis | *** | *** |
| Crude protein | 101 | Kruskal-Wallis | N/A | + |
| Growth habit | 22,007 | Chi-square | *** | *** |
| Flag leaf attitude | 21,344 | Chi-square | *** | *** |
| Spike type | 23,624 | Chi-square | *** | *** |
| Spike erectness | 22,376 | Chi-square | *** | *** |
| Grain type | 19,769 | Chi-square | *** | *** |
| Normal and uzu type | 8,180 | Chi-square | * | *** |
| Rough/Smooth Awn | 9,600 | Chi-square | + | *** |
| Spike shape | 940 | Chi-square | + | *** |
| Cold damage | 11,787 | Chi-square | *** | *** |
| Lodging | 6,221 | Chi-square | *** | *** |
| Vernalization degree | 98 | Chi-square | N/A | N/A |
| Powdery mildew | 100 | Chi-square | N/A | *** |
| Leaf stripe disease | 100 | Chi-square | N/A | *** |
| Barley yellow mosaic virus | 4,688 | Chi-square | + | *** |
| Loose smut | 686 | Chi-square | N/A | *** |
보리 유전자원의 대표성과 특이성
수치형으로 표현가능한 양적형질은 기후, 토양, 인간 등을 포함하는 환경적 요인에 의하여 작물의 형질이 변화하는 기존의 연구(Hallsson and Björklund, 2012)에 따라 수치형 특성평가 항목에 대한 관측결과의 이상치(outlier)와 재배 당시의 환경을 고려하지 않는다면, 비록 Table 2와 같이 통계적 유의성이 확인된다 하더라도 자원을 종별, 원산지별 범위를 결정하기가 어려워 군집화가 쉽지 않다. 보리 유전자원의 특셩평가 항목 중에서 수치형으로 조사되는 간장, 이삭길이, 천립중, 1수립수, 조단백함량을 대상으로 종, 원산지별로 구분하여 분포를 조사했을 때, 통계적으로 유의적 차이가 확인된다고 하더라도 조사결과가 모두 비슷한 범위에서 분포함을 알 수 있었다(Supplementary Fig. 1). 따라서 자원의 대표성과 특이성 분석을 위한 군집화를 위하여, 수치형 특성평가 항목을 제외하고 범주형을 대상으로 자원을 분류하였다.
범주형 특성평가 항목을 대상으로 한 보리 유전자원의 군집은 식물체 생장습성, 지엽 직립성 정도 등과 같이 코드화 된 15개 범주형 특성평가 항목을 대상으로 PowerCore에 의해 결정된 핵심집단 23개 자원과 함께 cacGMS 알고리즘에 의해 총 15개로 결정되었다. 각 군집에 대한 자원의 분포를 확인했을 때, 전체 자원의 97%가 1번에서 7번 군집에 밀집되어 있었다(Table 3). 또한 15개 군집을 대상으로 종과 원산지에 대하여 카이제곱 검정을 수행한 결과, 유의적 차이(P-value < 0.05)가 확인되어, 분류된 군집이 종과 원산지에 대한 특성의 차이가 반영되었을 것으로 유추할 수 있었다.
Table 3.
The number of germplasms and relative genetic traits in each cluster
| Cluster no. | Germplasms | Genetic traits |
| 1 | 4,497 | 14 |
| 2 | 4,374 | 15 |
| 3 | 4,304 | 15 |
| 4 | 3,366 | 13 |
| 5 | 3,047 | 13 |
| 6 | 2,751 | 15 |
| 7 | 2,070 | 13 |
| 8 | 394 | 12 |
| 9 | 141 | 14 |
| 10 | 92 | 14 |
| 11 | 41 | 11 |
| 12 | 13 | 2 |
| 13 | 8 | 9 |
| 14 | 4 | 7 |
| 15 | 2 | 2 |
종과 원산지에 따른 차이는 재배, 증식되는 지리, 환경적 영향이 반영된 자원 특성의 차이와 관련이 있음을 바탕으로 분류된 군집과 특성평가 항목 단위로 각 군집의 유의적 차이를 검정하였다(Table 4). 흰가루병과 줄무늬병 발병경률을 제외하고 13개 항목에서 유의적 차이가 확인되었는데, 흰가루병과 줄무늬병은 H. vulgare subsp. vulgare에서만 확인되었을 뿐만 아니라 원산지도 2개국(미국과 독일)에서만 조사되었기 때문에 유의적 차이가 나타나지 않았으나, 품종에 따라서 병 저항성이 차이가 있음(Park et al., 2006)을 고려해 볼 때, 흰가루병과 줄무늬병이 군집에 따른 유의적 차이가 없다고 단정하기는 어렵다.
Table 4.
Chi-square test for clusters with genetic traits of barley
| Genetic traits | Statistic | DF | P-valuez |
| Growth habit | 16777.07 | 24 | *** |
| Flag leaf attitude | 6066.22 | 24 | *** |
| Spike type | 18381.10 | 36 | *** |
| Spike erectness | 11593.53 | 44 | *** |
| Grain type | 10572.42 | 13 | *** |
| Normal and uzu type | 280.23 | 12 | *** |
| Rough/Smooth Awn | 1296.42 | 22 | *** |
| Spike shape | 275.85 | 20 | *** |
| Cold damage | 6872.13 | 60 | *** |
| Lodging | 866.73 | 55 | *** |
| Vernalization degree | 42.78 | 28 | *** |
| Powdery mildew | 9.49 | 15 | + |
| Leaf stripe disease | 9.49 | 15 | + |
| Barley yellow mosaic virus | 463.21 | 55 | *** |
| Loose smut | 175.79 | 40 | *** |
자원의 97%에 해당하는 1번~7번 군집을 대상으로 다수의 자원이 대표하는 특성과 각 군집별로 특이적으로 나타나는 특성을 분석하였다(Table 5). 군집별로 분류된 특성 가운데, 식물체 생장습성에서 잎과 분얼의 각도가 중간(Immediate), 지엽 직립성 정도의 중간(Intermediate), 이삭조성의 6조(Six rows), 이삭 직립정도의 직(Erect), 종실피과성의 피성(Husked), 망활의 조망(Rough)이 다수의 자원에서 확인되었다(Table 5, Supplementary Fig. 2, Supplementary Table 3). 군집에 있어서 공통적으로는 병와성의 병성(Normal), 도복상태의 무도복(Nothing), 깜부기병의 무발병(Nothing)이 확인되었고, 각 군집에 대한 특이적 특성으로는 특성평가 정보 조합의 차이와 함께, 특히 1번과 3번 군집은 한해에서 각각 21~40%, 41~60%, 7번 군집은 생장습성에서 평행수준인 개(prostrate) 특성이 나타났다(Table 5). 그 중에서 특히 5번 군집은 망활(반활망, semi-smooth), 한해(61~80%), 파성(type 3, 동보리 1호), 그리고 보리누른모자이크병 발병경율(1~5%)에서 다른 군집과 차별되는 특이성을 보였다.
각 군집에서 나타난 종의 분포를 확인했을 때, 모든 군집에서 공통적으로 H. vulgare subsp. vulgare가 포함되어 있었는데, 특히 다수의 자원이 포함되어 있는 1번~7번 군집에서 H. bulbosum와 Hordeum sp., H.vulgare가 비록 빈도(Frequency)의 차이는 있지만 공통적으로 분포하였다(Fig. 3). 각 군집에서의 특이적 분포로는, 특히 1번 군집에서는 H. arizonicum, H. bogdanii, H. chilense, H. jubatum, H. procerum가 빈도(Frequency)가 가장 높았고, 2번 군집은 H, lechleri의 빈도가 가장 높은 등 군집마다 빈도가 높은 종이 확인되었다. 그 외에 8번~15번 군집에서는 H. vulgare subsp. vulgare가낮은 빈도로 분포하였는데 특이적으로 Critesioniranicum은 9번 군집에 포함되었다.
Table 5.
Representative Genetic traits of each cluster over 97% cumulatively
특성평가 항목 간의 상관관계
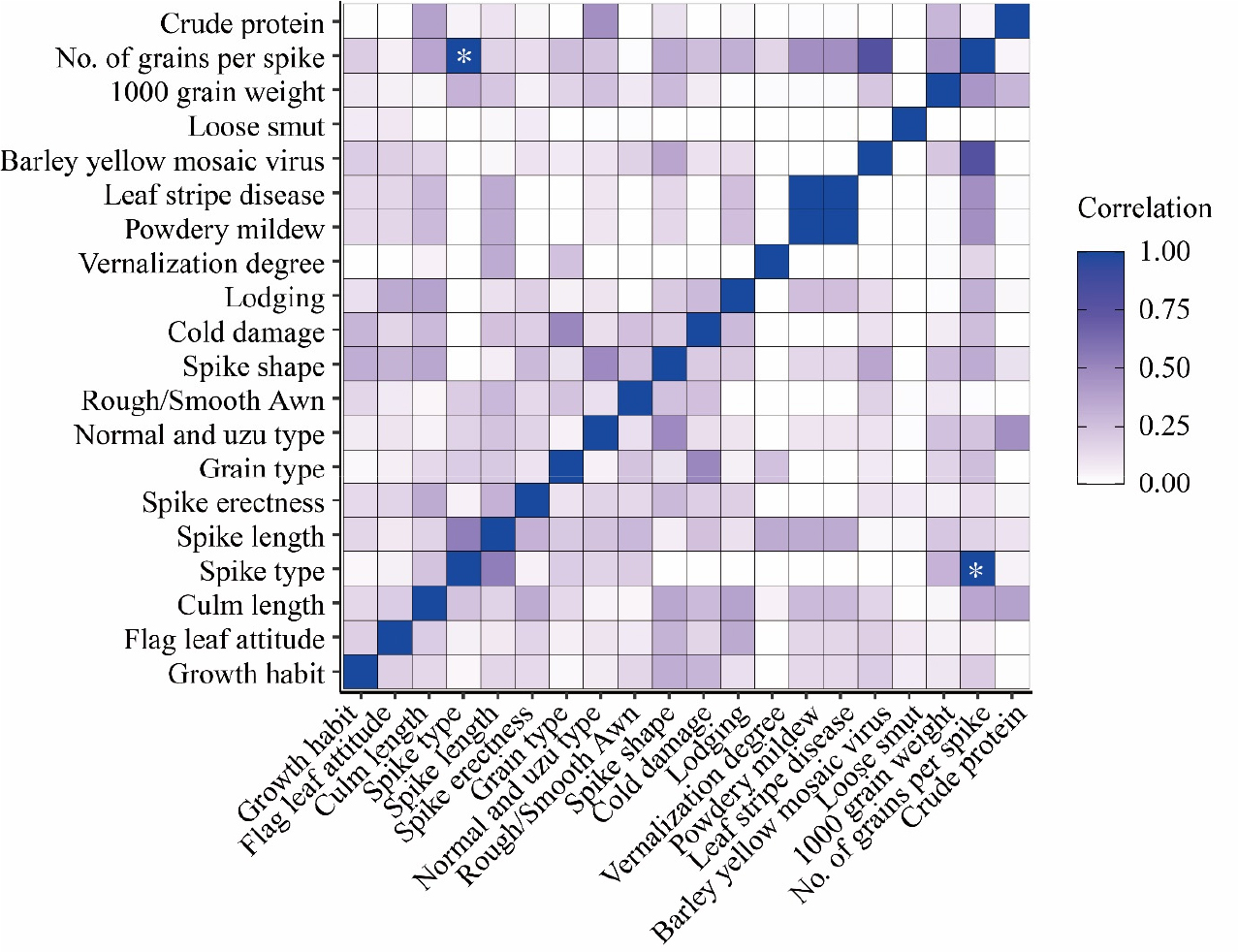
식물의 표현형질은 환경적 및 유전적 영향에 의해 결정되며 자손에게 유전되면서 다양한 형태로 진화하는데, 각각의 형질은 서로 독립적이거나 혹은 상호적인 관계가 있다(Jung et al., 2015; Lee et al., 2020). 또한 Table 5와 Fig. 3와 같이 보리 유전자원 군집은 다수의 형질의 조합으로 분류되고, 각 특성이 군집마다 차이가 있는 점, 자원의 군집이 종, 원산지에 따른 유의적 차이를 보인 점에서 각각의 형질이 상호적인 유연관계가 있을 것으로 추정할 수 있기 때문에, 보리 유전자원의 수치형, 범주형 특성평가 항목을 종합하여 각 형질 간의 상관관계를 분석하였다. 그 결과, 1수립수는 보리누른모자이크병과 상관성이 높은 것(상관계수 0.79)으로 확인되었는데(Fig. 4), 보리누른모자이크병은 Polymyxa gaminis 곰팡이균을 매개로 하는 토양전염성 바이러스에 의해 전염되어 작물의 수량 생산과 관련한 1수립수에 영향을 줄 것으로 예상할 수 있다(Park et al., 2004). 또한 종자의 특성에 있어서 중요하게 고려되는 천립중은 깜부기병, 줄무늬병, 흰가루병과는 관련이 없는 것으로 보이며, 비록 0.2에서 0.4사이의 낮은 상관계수이지만 이삭조성(0.31), 이삭길이(0.23), 병와성(0.24), 이삭모양(0.28), 보리누른모자이크병(0.23), 1수립수(0.43), 조단백함량(0.29)과 관련이 있는 것으로 추정된다. 흰가루병과 줄무늬병은 이삭길이(0.34)와 1수립수(0.46)와 상관관계가 있을 것으로 판단된다. 그런데 간장과 도복에 대해서는 각각 0.27과 0.26으로 낮은 상관성과 유의적 차이가 없는 것으로 나타났는데(P-value > 0.05), 특히 도복은 종실 생산량의 중요한 요소이고(Caierão, 2006), 흰가루병 저항성이 종실 생산량(Carver and Griffiths, 1981), 단백질함량과 상관관계가 있는 점(Öztürk et al., 2014)에서 서로 무관하다고 단정하기는 어렵다. 또한 줄무늬병과 흰가루병이 서로 매우 높은 비정상적 상관관계(≈ 1)를 보인 것은 1개 종(H. vulgare subsp. vulgare)에 대한 조사와 평가정보에 따른 통계적 오류로 판단되지만, 벼의 흰가루병 저항성 관련 유전자인 mlo 유전자가 QTL (Quantitative Traits Loci) 분석에서 줄무늬병 저항성에 관여하는 Rdg1a 부위와 서로 유사하다는 점(Biselli et al., 2010)을 고려할 때, 유전적 유사성에 따른 상관관계가 있을 것으로 유추해 볼 수 있다. 하지만 명확한 상관관계를 결정하기 위해서는 동일 환경에서 다양한 종과 원산지에 기반한 다수 자원을 대상으로 평가한 정보가 요구된다.
고 찰
본 연구는 국립농업과학원 농업유전자원센터에서 보존하고 있는 보리 유전자원에 대한 종, 원산지에 대한 현황, 특성평가 항목을 바탕으로 자원의 군집화를 통한 대표성 및 특이성 분석, 각 특성 간의 상관성을 분석하여 보리에 대한 형태적 형질의 다양성과 함께 상호적 관계를 확인하였다. 종과 원산지 다양성 분석에서 보존자원이 대표적인 식량자원으로서 육종연구가 많이 진행된 H. vulgare subsp. vulgare가 대부분을 차지한 이유로 인하여 나타난 낮은 종 다양성 지수는 보리 유전자원의 작물의 다양성 확보 및 자원 활용성 증대를 위해서는 다양한 종의 자원 수집과 품종 육종, 개량 등의 유전자원의 확보와 보존을 위한 증식방안의 필요성을 시사한다. 반면 특성평가 정보와의 관계에서 종에 대한 유의적 차이(P-value < 0.05)는 종 내에서도 형태적인 다양성이 있음을 유추할 수 있었다. 이를 토대로 수행한 군집 분석에서는 총 15개 군집으로 분류되었으며 전체 97%의 자원이 1번~7번 군집으로 나타났는데, 병와성, 도복, 깜부기병 항목이 공통적으로 동일한 형질을 보였으며, 생장습성, 망활, 한해, 파성, 보리누른모자이크병에서 다른 군집과 차별된 특성이 확인되었다. 또한 기 연구(Kim et al., 2003)에서도 확인된 1수립수와 보리누른모자이크병의 높은 상관성과 함께 천립중, 흰가루병 등 보리 유전자원 특성의 상관성 정보는 우수자원 선발, 신품종개발 및 육종산업에 본 연구결과를 활용함으로써 농업유전자원의 활용성 제고와 연구 선진화에 기여할 수 있을 것이다.
본 연구를 수행함에 있어서 자원의 수집과 각 자원에 대한 특성평가 항목 조사에 대한 다양성에 대한 정보전달과 연구 신뢰에 있어서 미흡한 부분이 존재한다. 특정 종에 집중된 수집, 각 자원에 대한 일부 특성평가 항목의 결측, 달관조사에서 범해질 수 있는 조사자의 경험과 지식에 의존한 인위적 판단 등, 다소 부정확한 특성평가 항목의 조사정보가 수집될 수 있는 점은 분석에 있어서 나타나는 오류를 배제하기는 어렵다. 이러한 문제를 해결하기 위해서는 작물의 형태적 형질에 대한 검증된 범용적 조사방법과 전문가 집단의 검토와 조사된 정보를 원활한 정보 관리를 위한 디지털화가 필요하다. 또한 작물의 다양성 확보를 위해서는 식량산업에 널리 이용되는 종 뿐만 아니라, 비주류에 해당하는 다양한 종도 함께 수집함과 동시에 장기적으로 보존할 수 있는 환경도 뒷받침되어야 할 것이다. 향후 장기적인 자원관리와 보존을 통해 종, 원산지 다양성 측면의 다양한 자원 수집, 지속적이고 안정적인 증식을 통한 자원 관리, 특성평가 항목 조사에 대한 디지털 기술기반 무결성 정보를 바탕으로 본 연구에서 수행한 형질 다양성 분석 및 상관성 연구가 진행된다면, 육종연구를 포함한 농업 전반의 연구에서 유용한 보리 유전자원 선발과 육종연구를 통한 신품종 개발 등을 위한 신뢰 높은 기초자료로 활용될 것으로 기대한다.
적 요
보리는 베타글루칸, 폴리페놀, 안토시아닌 등을 이용한 건강식품 소비 증가로 최근 관심이 높아지고 있다. 따라서 보리에 대한 수요자의 기호에 맞춘 기능성 품종개발과 소재로서 유전자원 활용성을 증대시키기 위해서는 자원의 특성 분석과 종, 원산지과의 관계, 군집화(Clustering)을 통한 유사성과 대표성, 형질 간의 상호관계 등과 같은 유전자원의 다양성 연구가 선행되어야 할 것이다. 본 연구는 농업유전자원센터에서 보존하고 있는 보리 25,104 유전자원(25종, 국적미상을 포함한 102개 원산지)을 대상으로 종과 원산지에 따른 다양성 분석을 수행하였다. 특히 종에 대한 작물 유효수(ENCS)는 1.17로 원산지(24.73)에 비해 매우 낮게 나타났다. 이는 보존하고 있는 보리 유전자원의 대부분이 Hordeum vulgare subsp. vulgare 로 확인되었는데, 원산지에 비해 보존 자원이 특정 종에 편중된 것을 알 수 있지만, 형태적으로 구분한 20가지 특성평가 항목에 대하여 종과 원산지에 따라 유의적인 차이(P-value < 0.05)가 검정되었다. 비록 종 다양성은 낮지만 종과 종간의 차이와 종 내에서도 다양한 특성이 존재함을 추정할 수 있었으며, 이를 토대로 특성평가 항목을 이용한 군집화를 통해서 특성에 대한 다양성을 확인하였다. 특성평가 항목을 바탕으로 cacGMS 알고리즘을 이용한 군집 분석을 수행했을 때, 전체 97%의 자원이 분류된 1번~7번 군집에서 병와성, 도복, 깜부기병 항목이 공통적으로 동일한 형질을 보였다. 반면에 군집 별 특이성에서는 특성평가 항목에 대한 조합의 차이와 함께 생장습성, 망활, 한해, 파성, 보리누른모자이크병에서 다른 군집과 차별되는 특이성이 확인되었다. 이러한 특성평가 항목에 의한 대표성과 특이성, 그리고 각 군집에 따른 특성의 조합은 특성 간의 상호적 관계와 관련이 있을 것으로 추정되어 상관관계를 분석하였다. 그 결과 1수립수와 보리누른모자이크병이 높은 상관성(상관계수 0.79)을 보였고, 종자연구에서 중요한 지표로 사용되고 있는 천립중은 낮은 상관계수이지만 이삭조성(0.31), 이삭길이(0.23), 병와성(0.24), 이삭모양(0.28), 보리누른모자이크병(0.23), 1수립수(0.43), 조단백함량(0.29)과 관련이 있을 것으로 추정된다. 본 연구에서 사용된 연구방법과 결과는 신품종개발, 육종산업에 활용가능한 정보를 제공하고, 이를 통해 농업유전자원의 활용성 제고와 연구 선진화에 기여할 수 있을 것으로 기대한다.