서 언
재료 및 방법
실험 재료
23개 자원들의 조직배양 효율 검증
제초제 저항성 유전자의 형질전환
삽입 유전자의 확인
결과 및 고찰
23개 자원들의 발아율 분석
발아된 자원들의 재분화 효율 분석
다양한 자원들의 형질전환
적 요
서 언
콩(Glycine max (L.) Merr.)은 고단백 유지 작물로 경제적 가치가 높고, 식용 외에 사료용, 녹비용 등 다양한 용도로 재배가 이루어지고 있다(Singh and Hymowitz, 1999; Vagadia et al., 2017). 주요 식량 작물인 콩의 농업적 가치를 향상시키고, 생산성을 증대시키기 위해 전통 육종 및 생명공학 기법을 이용한 품종 개량 연구가 활발하게 진행되고 있다(Kim et al., 2021; Melo et al., 2020). 최근에는 콩의 표준 유전체 정보를 활용하여 농업적으로 유용한 형질에 관련된 유전자 및 분자 표지를 개발하기 위한 연구(Chun et al., 2019)가 진행 중이며, 국내에서는 유전체 정보를 기반으로 유전적, 그리고 표현형적으로 다양성을 가진 콩 핵심집단 약 430개 자원들이 선발되어, 핵심집단을 이용한 콩의 유전 및 육종 연구에 활용이 기대되고 있다(Jeong et al., 2019).
유전체 정보를 바탕으로 대량 분자 표지 및 유용 유전자 발굴이 본격화되면서 새로운 유전자의 기능을 규명하고, 이를 작물에 도입하여 유용 형질을 가진 품종으로 개발하기 위한 형질전환 기술 개발 연구가 이루어지고 있다(Blanca et al., 2012). 최근에는 CRISPR/Cas9과 같은 유전자 가위 기술을 이용하여 목적 유전자를 직접적으로 교정하여 새로운 형질을 가진 작물을 개발하는 연구가 다양한 식물에서 시도되고 있다(Jaganathan et al., 2018). 유전자 교정 기술을 포함한 식물 생명공학 기술의 적용을 위해서는 형질전환을 위한 효율적인 조직배양 기술의 확립이 선행되어야 하지만, 옥수수, 밀, 보리 등과 같이 농업적으로 중요한 작물들의 조직배양 효율은 낮고, 일부 품종에 한정되어 조직배양이 이루어지고 있다(Yang et al., 2011). 콩은 조직배양이 어려운 식물로 알려져 있는데, 미성숙 자엽에서 유도된 체세포 배를 이용하여 Jack을 비롯한 일부 품종에서 particle bombardment를 사용한 형질전환이 주로 이루어지고 있다(Dufourmantel et al., 2005; Kita et al., 2007). Particle bombardment법은 물리적 방법에 의해 목적 유전자를 도입하기 때문에 형질전환이 비교적 용이하지만, 미세 입자로 목적 유전자를 코팅하기 위한 유지 비용 및 고가 장비의 구입 등 고비용이 소요되고, 도입된 유전자의 불안정적 발현 및 다중 도입(multiple copy numbers)의 문제점을 가지고 있다. 반면에, Agrobacterium법은 저비용으로 목적 유전자의 안정적인 도입이 가능하다는 장점을 가지고 있기 때문에 많은 식물에서 기술의 확보를 위한 연구가 이루어지고 있다(Li et al., 2017). 최근, 콩에서는 자엽절(cotyledonary node) 조직을 사용하여 Agrobacterium법에 의한 형질전환 연구가 진행되고 있으며, 표준 유전체 해독이 이루어진 Williams 82 품종에서는 제초제 저항성(Zeng et al., 2004), zein 단백질 축적 유전자(Kim and Krishnan, 2004)의 형질전환 연구가 보고되었고, Jack 품종에서는 α-linolenic acids 생합성에 관여하는 GmFAD3 유전자의 형질전환이 보고된 바 있다(Flores et al., 2008). 그러나, Agrobacterium법은 T-DNA의 식물 세포내 도입을 위한 조건 탐색 및 도입된 세포의 재분화를 위한 효율적인 조직배양 기술의 어려움 때문에 아직까지 일부 식물이나 품종에 한정되어 연구가 진행되고 있다(Seo et al., 2020; Zhao et al., 2019). 콩에서도 Agrobacterium을 이용한 형질전환 연구가 진행되고 있으나 국내에서는 아직까지 콩의 형질전환 효율은 낮고 형질전환 가능한 품종이 제한적이다(Jeon and Chung, 2003; Kim et al., 2017). 따라서, 형질전환 가능한 콩 품종을 확대하고 효율적인 형질전환 기술을 개발하는 연구는 콩의 안정적 생산 및 경제적 가치 향상을 위한 새로운 육종 소재 개발 연구를 위해 매우 중요하다. 식물의 유전형(genotype)은 조직배양 효율 및 형질전환의 성공 여부를 결정짓는 중요한 요소로 알려져 있다(Ono et al., 1994). 최근, 국내에서는 우람, 진품2호, 청자3호, 태광 품종을 사용하여 조직 부위 및 배지 종류에 따른 재분화 효율을 검토(Kim et al., 2016)한 바 있으며, Kim et al. (2008)은 익산나물콩의 발아된 종자의 분열조직을 재료로 사용하여 Agrobacterium법에 의한 안정적인 형질전환을 보고한 바 있다.
본 연구에서는 Agrobacterium법에 의한 형질전환에 적합한 콩 품종을 확대하기 위하여, 콩 핵심집단에서 선발한 자원들의 재분화효율을 검토하고, 선발된 자원을 대상으로 유전자 교정용 CRISPR/Cas9 벡터를 사용하여 Agrobacterium법에 의한 형질전환을 수행하였다. 이들 결과는 향후 콩 유전자 교정을 통한 육종 소재 개발에 활용이 가능할 것이다.
재료 및 방법
실험 재료
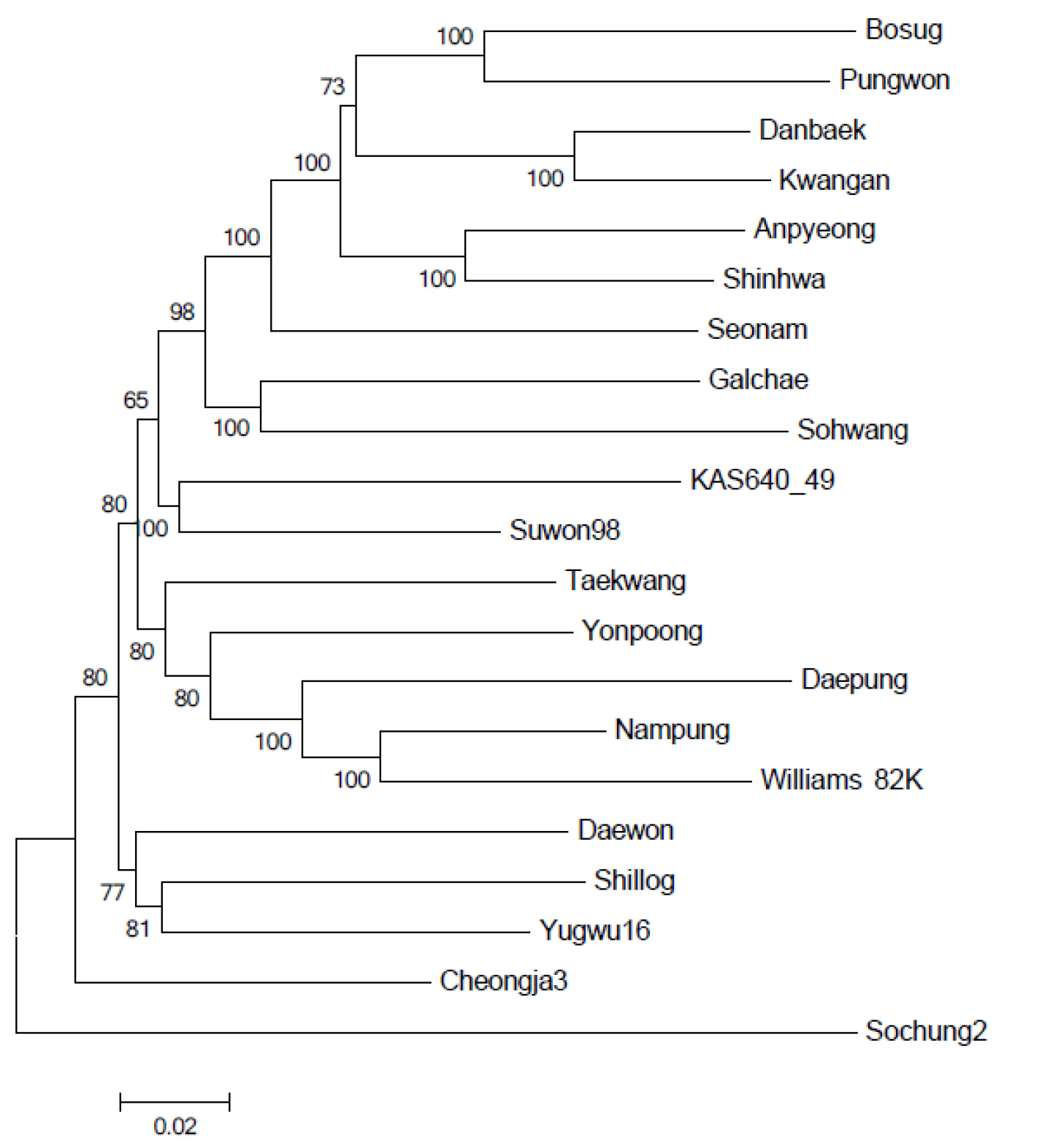
본 연구를 위하여 콩 핵심집단(Jeong et al., 2019)에 속하는 21개 자원들을 선발하여 실험에 사용하였다. 또한, LG팜한농에서 보유한 미국 품종 Jack, Maverick을 제공받아 총 23개 자원들을 사용하여 콩 조직배양 효율을 검토하였다. 핵심집단에 속하는 21개 자원들에 대한 phylogenetic tree는 고밀도 180K SNP chip 데이터(Jeong et al., 2019)를 이용하여 MEGA6 software를 통해 분석되었다.
23개 자원들의 조직배양 효율 검증
총 23개 자원들의 조직배양 효율을 검증하기 위하여 각 자원 별로 종자를 표면 소독하여 배지에 치상하였다. 종자는 70% ethanol로 1분간 표면 살균한 후, 2% sodium hypochlorite 용액에서 20분간 소독하였다. 소독이 끝난 종자는 멸균수로 3회 이상 세정한 후 실온에서 16시간 정도 침지하였다. 침지가 끝난 종자의 물기를 제거한 후, 종피를 제거하고 종자를 반으로 자른 후 3% sucrose, 0.8% agarose를 포함하는 MS 기본 배지에 치상하여 2주간 배양한 후 종자의 발아율을 조사하였다. 발아된 종자는 배축(embryonic axis)을 제거한 후, 2 ㎎/L BAP (6-Benzylaminopurine), 3% phytogel을 포함한 MS 배지에 치상하여 배양 4주 후 자엽절 조직으로부터 재분화된 3 ㎜ 이상의 유식물체를 재분화 개체로 판단하여 재분화 효율을 조사하였다.
제초제 저항성 유전자의 형질전환
콩 형질전환은 Li et al. (2017)의 방법을 이용하여 수행되었다. 형질전환을 위해 사용된 벡터는 제초제 저항성(bar) 유전자가 선발 마커로 삽입된 유전자 교정용 pBAtc:tRNA02 벡터로 한국생명공학연구원으로부터 분양받아 사용하였다. 벡터는 A. tumefaciens GV3101에 형질전환하여, YEP 배지(25 g/L YEP broth, 15 g/L bacto agar, 50 ㎎/L spectinomycin, 100 ㎎/L gentamycin, 50 ㎎/L rifampicin)에서 배양한 후, 형질전환된 콜로니를 채취하여 액체 YEP 배지에서 28℃, 50 rpm 진탕 배양기에서 하룻밤 동안 배양하였다. 증식된 Agrobacterium은 10분간 5000 rpm으로 원심하여 침전된 세포를 모은 후 acetosyringone 40 ㎎/L가 첨가된 액체 감염 배지(CCM; 3.9 g/L MES, 30 g/L sucrose, 1 mM sodium thiosulfate, 1 mM dithiothreitol, 3.3 mM L-cystein, 0.25 ㎎/L GA3, 1.67 ㎎/L benzylaminopurine이 첨가된 B5 기본 배지, pH 5.4)에 OD650=0.6 ~ 0.7 농도로 재현탁하였다. Agrobacterium법에 의한 형질전환을 위해 상기의 조직배양 효율 검토를 통해 선발된 품종들을 대상으로 pBAtc:tRNA02 유전자 형질전환을 수행하였다. 형질전환을 위해 종자는 70% ethanol로 1분간 표면 살균한 후, 2% sodium hypochlorite 용액에서 20분간 소독하였다. 소독이 끝난 종자는 멸균수로 3회 이상 세정한 후 실온에서 16시간 정도 침지한 후 여과지 위에서 종피를 제거하였다. 종피를 제거한 종자는 배축을 중심으로 절반으로 절단한 후 Agrobacterium 현탁액에 30분간 접종하였다. 접종이 끝난 종자는 8 g/L plant agar가 첨가된 고체 감염 배지(CCM)에 치상하여 16/8h 명조건, 25℃에서 3일간 공동배양하였다. 접종이 끝난 종자는 뿌리 부분을 제거한 후, SIM 신초 유도 배지(0.58 g/L MES, 30 g/L sucrose, 1 mM sodium thiosulfate, 1 mM dithiothreitol, 3.3 mM L-cystein, 1.67 ㎎/L benzylaminopurine, 250 ㎎/L cefotaxime, 100 ㎎/L ticarcillin이 첨가된 B5 기본 배지, pH 5.6)에 치상하여 16/8 h 명조건, 25℃에서 10일간 배양하였다. 발아가 관찰된 절편체는 배축 부분을 제거한 후, 5 ㎎/L glufosinate를 첨가한 SIM 신초 유도 배지에 이식하여 25℃ 배양실(16/8h 광조건)에서 glufosinate 저항성 신초를 유도하였다. 절편체로부터 1 ㎝ 이상 재분화된 신초는 0.58 g/L MES, 30g/L sucrose, 1 mM sodium thiosulfate, 1 mM dithiothreitol, 50 ㎎/L asparagine, 100 ㎎/L pyroglutamic acid, 3.5 g/L phytagel, 0.5 ㎎/L GA3, 0.1 ㎎/L IAA (indoleacetic acid), 1 ㎎/L zeatin-riboside, 250 ㎎/L cefotaxime, 100 ㎎/L ticarcillin, 5 ㎎/L glufosinate가 첨가된 MS 재분화(SEM) 배지에 치상하여, 2주 간격으로 계대배양을 수행하였다. 신장된 신초는 30 g/L sucrose, 8 g/L plant agar, 1 ㎎/L IBA(indole-butyric acid), 250 ㎎/L cefotaxime, 100 ㎎/L ticarcillin, 5 ㎎/L glufosinate 가 첨가된 B5 발근 유도 배지에 치상하여 뿌리를 유도하였다. 뿌리가 유도되어 완전한 glufosinate 저항성을 보이는 유식물체는 순화 과정을 거쳐 화분에 이식한 후 온실에 옮겨 정상적인 식물체로 유도하였다.
삽입 유전자의 확인
Glufosinate 저항성 유식물체의 잎에서 Bar-strip과 PCR 분석을 통해 유전자의 삽입을 확인하였다. Bar-strip 분석은 비형질전환 식물체와 형질전환 유식물체의 잎 0.1 g을 분쇄하여 멸균수 0.5 mL를 첨가한 후 LibertyLink® strip을 용액에 넣어 밴드의 유무를 확인하였다. PCR 분석을 위해서는 형질전환체의 잎 0.1g을 분쇄한 후, Quiagen plant mini kit를 사용하여 gDNA를 추출하였다. 분리된 DNA는 bar 유전자 특이적 프라이머(barF ATGAGCCCAGAACGACGC, barR TTCTGGCAGCTGGACTTCAG)를 사용하여 PCR분석을 수행하였다. PCR 은 denature; 94℃ 5 min, annealing; 94℃, 30 sec, 58℃ 30 sec, 72℃ 30 sec, 30 cyles, elongation; 72℃, 5 min으로 수행되었고, 0.8% agarose gel 전기영동을 통해 bar 유전자의 삽입 여부를 확인하였다.
결과 및 고찰
23개 자원들의 발아율 분석
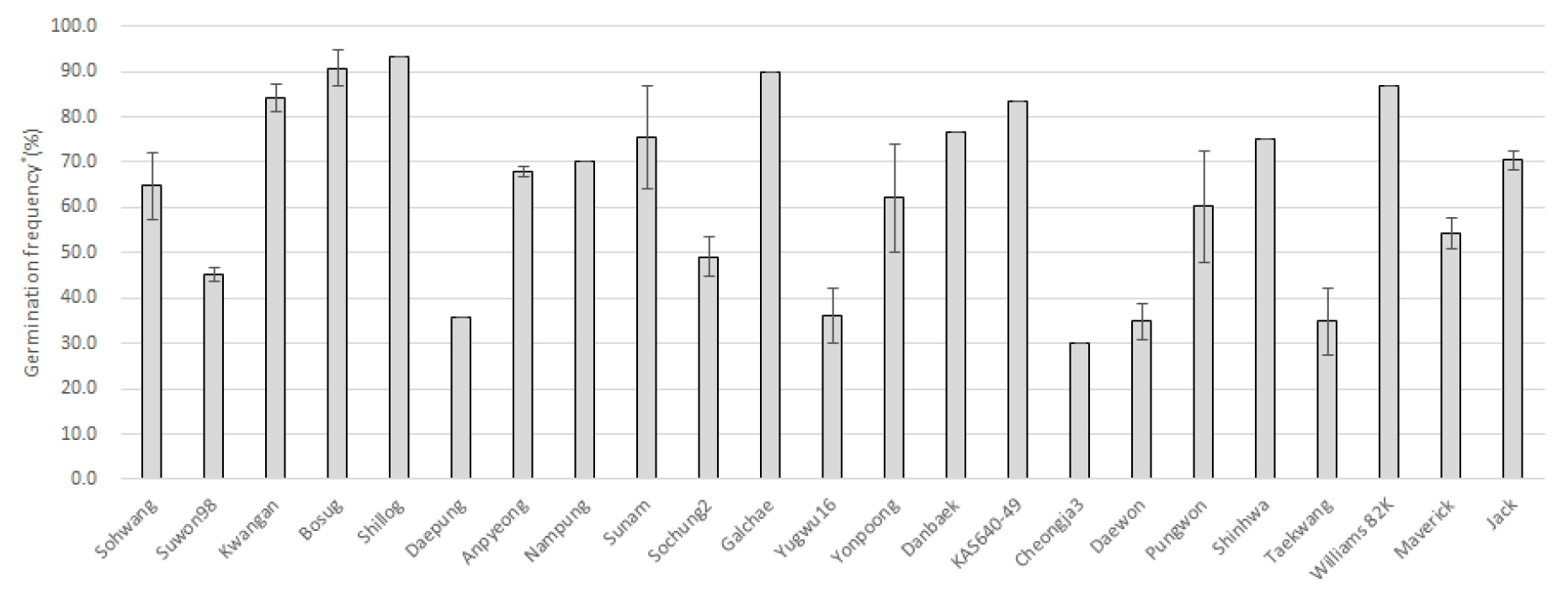
콩 형질전환 적합 자원 선발을 위해 우선 핵심집단 내 21개 자원들과 미국 품종인 Jack, Maverick의 발아율을 조사하였다(Fig. 1). 발아 종자의 자엽절(cotyledonary node) 조직을 통한 재분화 효율 검증을 위해 half-seed로부터 발아 효율을 조사한 결과, 자원에 따라 발아 시기에는 차이가 있으나 약 2주 후 모든 자원들에서 발아가 확인되었고, 16개 자원들에서는 평균 50% 이상의 발아율이 관찰되었다. 반면에 장류용 콩에 속하는 Daepung, Daewon, Taekwang, Yugwu16, 그리고 Cheongja3에서는 40% 이하의 낮은 발아율을 보였다. 일반적으로 종자의 발아율은 여러가지 요인에 의하여 영향을 받는데 특히 종자의 보관 상태 및 발아 기간 동안의 온도, 수분 등과 같은 환경적 요인에 의해 영향을 받는 것으로 알려져 있다(Kim et al., 2005). 최근 콩의 침종처리에 따른 발아율을 조사한 결과, 전기전도율과 총 용존 고형물(Total Dissolved Solid (TDS))이 낮고, 종자의 수분흡수가 낮은 품종들에서 높은 발아율이 관찰되었는데 종자의 크기가 작은 나물용 품종들이 장류용 및 밥밑용 품종들과 비교하여 전기전도율과 TDS, 수분흡수가 낮아 상대적으로 발아율이 높게 관찰되었다(Cho et al., 2015). 이와 함께, 21개 자원들의 유전적 근연관계에 따른 조직배양 효율 분석을 위하여 한국 콩 핵심집단의 고밀도 180K SNP chip 데이터를 사용하여 21개 자원들의 phylogenetic tree 분석을 수행하였다(Fig. 2). 나물용콩으로 개발된 Anpyeong, Kwangan, Shinhwa, Seonam 콩은 유전적 근연 관계가 매우 높게 나타났으며, 표준유전체 해독 품종인 Williams 82는 Nampung 및 Daepung과 같은 장류용콩과 유전적으로 높은 근연 관계를 보였다. 본 실험에서는, 유전적 근연관계가 매우 높은 Kwangan, Anpyeong, Seonam 콩에서 높은 발아율을 보인 반면에, 이들 자원들과 근연관계가 낮고, 장류용콩에 속하는 Daewon, Daepung 콩에서는 낮은 발아율이 관찰되었다. 반면에, Daepung 콩과 같은 장류용콩과 유전적 근연관계가 높지만, 소립콩에 속하는 Williams 82의 경우, 높은 발아율을 보였다. 따라서 종자의 크기 및 유전적 요인이 콩 종자의 발아율에 미치는 영향에 대한 명확한 분석을 위하여 광범위한 유전적 분석 및 형질 조사가 필요할 것으로 사료된다.
발아된 자원들의 재분화 효율 분석
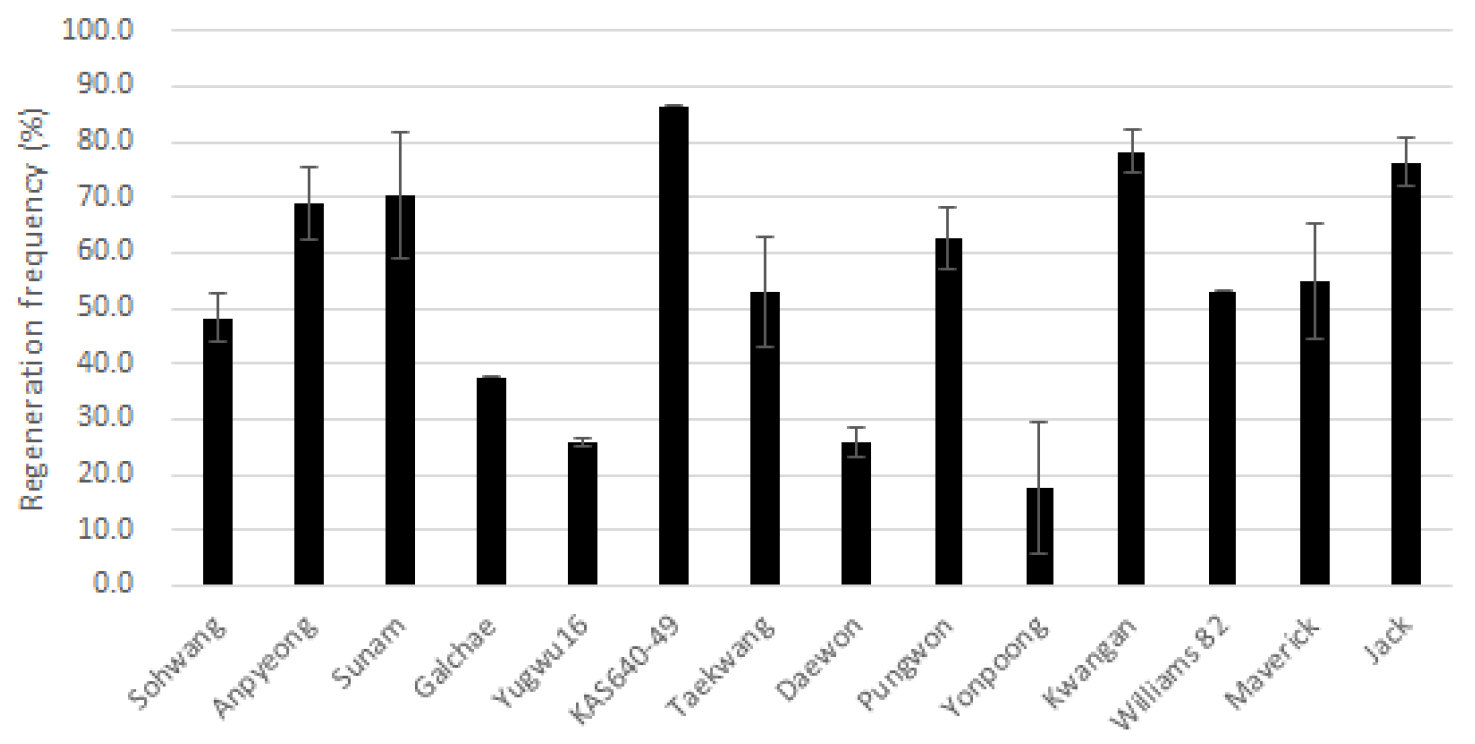
발아가 확인된 종자는 배축을 제거한 후, 자엽절 조직을 재분화 배지에 4주간 배양하였다. 재분화 과정에서 종자 오염 등의 문제로 재분화 효율 검증이 어려운 자원을 제외한 총 14개 자원들의 재분화율을 조사한 결과는 Fig. 3과 같다. 우리나라 재래종으로 알려진 KAS640-49은 86.4%의 가장 높은 재분화 효율이 관찰된 반면, Yonpoong은 평균 17.6%로 가장 낮은 재분화 효율을 보였다. 또한, Anpyeong, Seonam, Kwangan과 같은 나물용 품종들의 경우, Daewon, Taekwang과 같은 장류용 대립종 품종들과 비교하여 높은 재분화 효율을 보였다. 표준 유전체 해독 재료로 사용된 Williams 82의 재분화율은 약 53%를 보였고, 미국 품종인 Jack은 평균 76.3%의 높은 재분화 효율을 보였다. KAS640-49, Kwangan, Jack 등의 품종들은 종자 발아율과 재분화 효율 모두 높은 자원으로 확인되어 향후, 생명공학 기술을 위한 재료로써 사용이 가능할 것으로 사료된다. 식물의 재분화 효율은 배양 배지 및 절편체 종류, 그리고 유전형에 의해 영향을 받는 것으로 알려져 있다(Ge et al., 2006). 특히 유전형은 식물의 재분화 및 형질전환 효율에 영향을 미치는 중요한 요인으로, 조직배양 효율이 높은 자원을 선발하여 생명공학 기술을 적용하는 연구가 활발하게 진행되고 있다(Zhang et al., 1998). 본 실험에서는 Fig. 2와 같이 유전적 근연관계가 높은 Kwangan, Anpyeong, Seonam 그룹에서는 대체적으로 높은 발아율과 재분화율이 관찰된 반면에, 이들 그룹과 유전적 근연 관계가 낮은 Daewon, Yugwu16은 낮은 조직배양 효율을 보였다. 따라서, 콩의 핵심집단 내 자원별 조직배양 효율이 유전형과 밀접한 상관관계를 가지고 있음을 확인할 수 있었다. 콩의 조직으로부터 식물체 재분화에 관한 연구는 최근까지도 활발하게 이루어지고 있다(Karthik et al., 2019; Raza et al., 2017). 특히, 콩의 절편체 종류에 따른 재분화 연구가 진행되었는데, 최근에는 주로 자엽절을 이용한 효율적인 재분화와 Agrobacterium법에 의한 형질전환 연구가 보고되고 있다(Li et al., 2017; Paz et al., 2006).
다양한 자원들의 형질전환
본 연구에서는 Agrobacterium법을 이용한 콩의 형질전환을 위하여 자엽절 조직을 사용하여 다양한 자원에서 발아율 및 재분화 효율을 검토하여 효율적인 형질전환을 위한 자원을 선발할 수 있었다. 콩 21개 자원들의 발아율 및 재분화 효율 검증을 통해 비교적 양호한 조직배양 효율을 나타낸 Kwangan, Anpyeong, Seonam, Pungwon, Maverick 품종을 선발하여 Agrobacterium법을 이용한 형질전환을 수행하였다. 실험에 사용한 유전자는 제초제 저항성(bar) 유전자를 포함하는 유전자 교정용 CRISPR/Cas9벡터인 pBAtc:tRNA02를 사용하였고, half-seed에 Agrobacterium을 감염시켰다. 감염된 절편체는 재분화 유도 배지에서 glufosinate에 저항성을 가진 유식물체를 선발할 수 있었다(Fig. 4). 본 실험의 결과, Kwangan, Pungwon, Seonam, 그리고 Maverick 품종에서 glufosinate 저항성 유식물체가 관찰되었고, 이들 식물체는 bar-strip 검정과 PCR 분석을 통해 제초제 저항성 유전자(bar)의 형질전환 여부를 확인할 수 있었다(Fig. 5). 유전자의 삽입이 확인된 유식물체는 순화 과정을 거쳐 화분에 이식한 후 온실에서 재배하였다. 콩의 자엽절을 이용한 Agrobacteriuim에 의한 형질전환은 2000년도 이후 다양한 품종을 통해 연구가 진행되었다(Eckert et al., 2006; Sato et al., 2004). 표준 유전체 해독 품종인 Williams82에서는 Zeng et al. (2004)이 제초제 저항성 유전자를 형질전환하였고, Flores et al. (2008)은 Jack 품종에서 α-linolenic acids 함량 감소에 관여하는 GmFAD3 유전자의 형질전환을 보고한 바 있다. 우리나라에서는 Agrobacteriuim을 이용한 콩 형질전환 연구는 일부 품종에서 제한적으로 이루어지고 있는데, 자엽절을 이용한 Kwangan콩의 형질전환(Kim et al., 2017)과 배축 조직을 이용한 Iksannamul콩(Kim et al., 2008)의 형질전환이 보고된 바 있다.

Fig. 4.
Agrobacterium-mediated transformation of pBAtc:tRNA vector with bar gene for gene editing in soybean cultivar ‘Kwangan’. (A) Co-cultivated half-seed explants for three days, (B) Half seeds cotyledonary explants in SIM medium, (C) Regenerated shoot in SEM medium containing 5 ㎎/L glufosinate, (D) Rooted plantlet in RM medium containing 5 ㎎/L glufosinate.
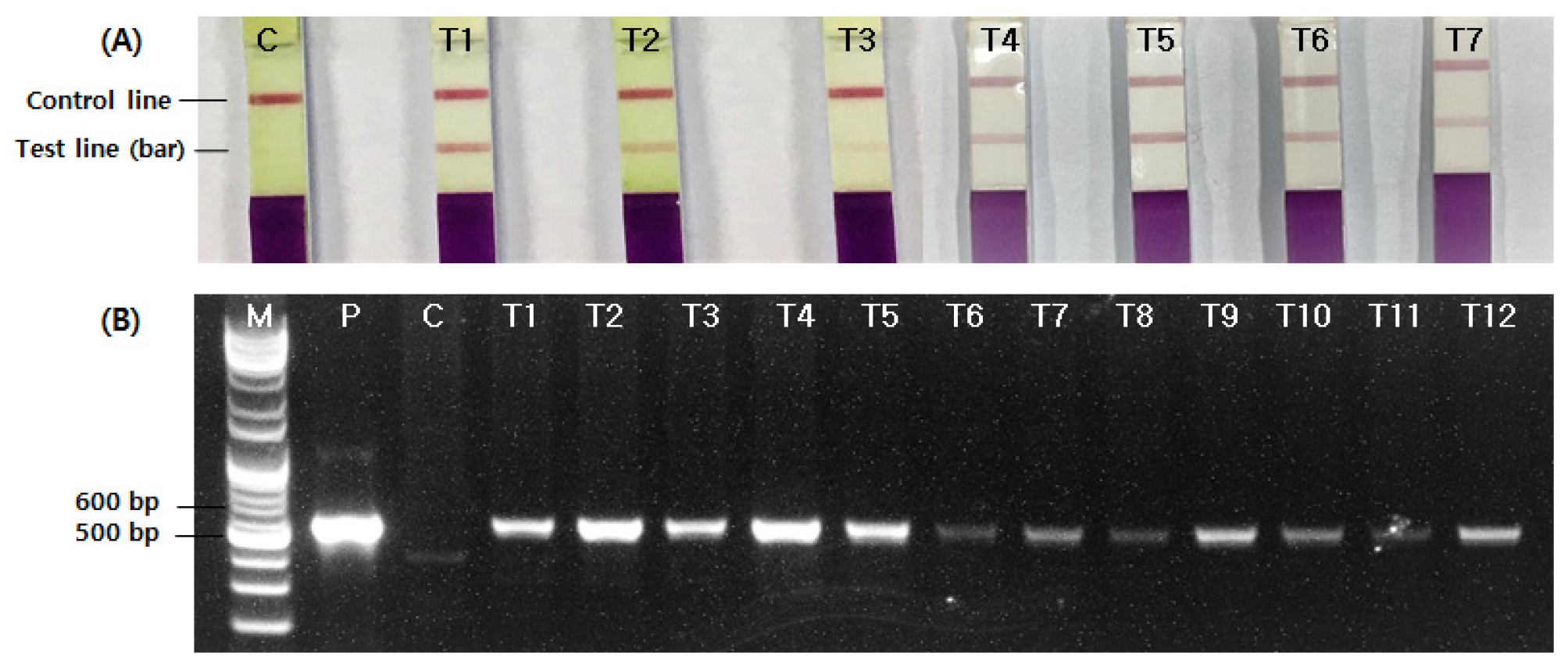
Fig. 5.
Detection of the bar gene in transgenic soybean plants. (A) LibertyLink® strip detection. The first line is control line, and the second line is test line. C; non-transgenic plant, T1~T7; transgenic plants (T1, T2; Kwangan, T3~T4; Maverick, T5~T6; Seonam, T7; Pungwon), (B) PCR amplification of the 506 bp bar gene fragment. M; 1 kb molecular marker, P; pBAtc:tRNA02 vector plasmid, C; non-transgenic plant, T1~T12; transgenic plants (T1~T2; Seonam, T3~T7; Kwangan, T8~T11; Maverick,T12; Pungwon).
최근, GMO의 안정성 문제를 회피할 수 있는 식물 생명공학 기술로서 CRISPR/Cas9과 같은 유전자 가위 기술이 주목받고 있다(Cai et al., 2018). 유전자 가위 기술을 이용하여 원하는 유전자 부위를 교정하여 단기간에 새로운 형질을 가진 품종을 개발하기 위해서는 농업적 가치가 높은 품종에서 효율적인 형질전환 시스템이 구축되어야 할 것이다. 본 논문에서는 유전체 연구가 진행되고 있는 우리나라 콩 핵심집단에서 다양한 자원들을 선발하고, 글로벌 종자 시장 선점을 위한 품종으로 3 품종을 선정하여 조직배양 효율을 검토하였다. 또한 조직배양 효율이 높은 일부 자원들에서는 Agrobacterium법을 이용한 형질전환을 통해 제초제 저항성 유전자의 삽입을 확인할 수 있었다. 본 연구에서 확립된 형질전환 시스템을 이용한다면, 향후 유용 형질 관련 유전자 교정을 통한 새로운 육종 소재 개발이 활발하게 이루어질 수 있을 것이다.
적 요
유전자 가위 기술 등 생명공학 기술을 콩에 적용하여 새로운 품종을 개발하기 위해서는 효율적인 조직배양 기술이 필수적이다. 식물의 유전형은 조직배양 효율에 의존하는 형질전환 기술의 성공 여부를 결정짓는 중요한 요소로 알려져 있다. 본 연구에서는 우리나라 콩 핵심 집단 내 21개 자원들을 선발하여, 외래 품종 2종과 함께 조직배양 효율을 조사하였다. 그 결과, 근연 관계가 높은 Kwangan, Anpyeong, Seonam은 발아율과 재분화 효율이 높았으며, Daepung, Daewon 품종은 발아율과 재분화율 모두 낮게 관찰되었다. 또한 3종의 외래 품종에서는 표준 유전체 해독에 사용된 Williams82와 Jack, Maverick 모두 높은 조직배양 효율을 보였다. 조직배양 효율이 높은 자원들을 대상으로 Agrobacterium법에 의한 형질전환을 수행하여 PCR 및 bar-strip 분석한 결과 Kwangan, Pungwon, Seonam, 그리고 Maverick 품종에서 제초제 저항성 유전자의 삽입을 확인할 수 있었다. 이들 결과를 바탕으로 농업적 가치가 높은 다양한 콩 품종들의 형질전환을 통한 새로운 품종 개발이 가능할 것이다.