서언
재료 및 방법
식물 재료와 RNA 분리
‘조생혈도’와 ‘미백도’의 RNA–Seq data를 이용한 SNP 분석
HRM (High Resolution Melting) analysis
결과 및 고찰
복숭아 RNA-Seq data를 이용한 SNP 분석
HRM 분석을 이용한 SNP 분자표지 개발
적 요
서언
복숭아나무는 장미과(Rosaceae) 자두속(Prunus) 복숭아아속(Amygdalus)에 속하는 온대 낙엽성 과수로 사과나무나 배나무보다 유전적 변이가 적고 조기 결실 및 자가 수정이 가능하여 많은 유전 양식이 보고되었다(Monet et al., 1996). 복숭아 육종 프로그램은 수요자의 다양한 요구를 충족하기 위해 진행되어 왔으며, 이를 통해 많은 품종들이 개발, 보급되어 왔다. 최근에는 과실의 기능성에 대한 소비자 요구가 증대됨에 따라 기능성 과수 품종 육성도 중요한 육종목표가 되었다. 복숭아 육종의 소재가 되는 유전자원들은 수확기의 과육색을 기준으로 백색 과육(백육계), 적색 과육(적육계) 및 황색 과육(황육계)으로 나뉜다. 우리나라에서는 백육계 복숭아 품종이 많이 재배, 생산되고 있지만, 기능성 성분은 적육계 및 황육계에 더 많이 함유되어 있다. 따라서 복숭아 기능성 물질 함량 증진을 위해서 적육계 및 황육계 등 유색 품종 육성은 중요한 육종 목표 중 하나가 될 수 있다(Cevallos-Casals et al., 2006). 육종 소재로 사용할 수 있는 적육계의 복숭아 유전자원으로는 ‘조생혈도’, ‘흑고랑(黑姑娘)’ 품종 등이 있으나 그 수가 많지 않다. 또한 적육계 복숭아 유전자원은 대체로 산미가 높아 소비자의 기호도가 낮기 때문에 식미를 개량하기 위해서 산미가 적고 당도가 높은 백육계 또는 황육계 품종들과 교배를 통해 세대를 진전시켜야 한다. 이와 같이 과수는 유년기가 길기 때문에 과수 육종 프로그램의 효율을 높이기 위해서는 과육색과 같이 목적 형질과 연관된 분자표지의 개발과 활용으로 조기에 교배 집단을 축소할 필요가 있다.
복숭아 과피의 붉은색은 MYB10 유전자인 PpMYB10.1, PpMYB10.2, PpMYB10.3에 의해 안토시아닌 생합성을 조절함으로써 발현하는 것으로 알려져 있다(Tuan et al., 2015). 복숭아 과육의 붉은 색은 bf, 단일 열성 유전자에 의해 발현되는 것으로 알려져 있고(Werner et al., 1998), 이 유전자는 linkage group 4에 위치한다고 보고되었다(Gillen and Bliss, 2005). 그러나 최근 이러한 단일 열성 유전자에 의해 발현되는 ‘Harrow Blood’ 품종과는 달리 중국의 ‘Wu Yue Xian’ 품종의 붉은색 과육은 단일 우성 유전자인 DBF (Dominant Blood-Flesh)에 의해 발현되고 bf의 대립인자가 아니며 linkage group 5의 윗부분에 위치하고 dihydroflavonol-4-reductase 유전자군 중 3개의 유전자가 DBF를 조절하는 것으로 밝혀졌다. DBF는 AMPP157과 AMPP178과 같은 SSR 분자표지로 조기선발이 가능한 것으로 보고되었다(Shen et al., 2013). 또한 NAC transcription factors (PpNAC1)가 안토시아닌 생합성을 조절하여 복숭아의 과육에서 적색 발현을 조절한다는 연구결과가 보고되었다(Zhou et al., 2015). 이외에도 복숭아의 붉은색 과육에 관련한 quantitative trait loci (QTLs)가 linkage group 1과 linkage group 3에 위치하고 있음이 보고되었고(Quilot et al., 2004), 이러한 결과는 복숭아에서 붉은색 과육을 나타내는 표현형이 한 개 유전자에 의해서 조절되는 것이 아니라 보다 복잡한 기작에 의해서 발현되는 것임을 알 수 있다.
본 연구는 기존에 보고된 DBF 유전자에서 유래한 SSR 분자표지 외에 적색 과육 복숭아를 조기 선발할 수 있는 분자표지 개발을 위해 적색 과육 복숭아 품종 ‘조생혈도’와 백색 과육 복숭아 품종 ‘미백도’의 EST 분석 결과를 바탕으로(Kim et al., 2012) 안토시아닌 발현에 관여하는 유전자들을 비교 분석하였다. HRM 분석방법을 사용하여 적색 과육 복숭아 품종과 백색 과육 복숭아 품종의 PCR 증폭산물에서 한 개의 서로 다른 염기서열을 구분하여 분리할 수 있는 SNP 위치를 확인하였다. 이 방법은 식물의 변이종을 분석하기 위해 많은 식물에서 이용되어 왔고, 유전자의 염기다형성 분포를 통해 특정 재배지역에서 지표로 사용되는지에 대한 연구결과도 보고되었다(Kwon and Chung, 2018). 적색 과육 발현에 관련한 특이적 SNP 분자표지를 탐색하여 복숭아 유전자원과 육성품종 및 계통 중에서 적색 과육 품종과 백색 과육 품종을 구분할 수 있는 분자표지를 개발하였다. 본 연구에서 개발한 SNP 분자표지를 활용하여 다양한 복숭아 유전자원과 교배집단에 적용하여 적육계 품종의 육종연한을 단축할 수 있는 방향을 제시하고자 한다.
재료 및 방법
식물 재료와 RNA 분리
본 연구에 사용한 재료는 농촌진흥청 국립원예특작과학원에 재식되어 있는 적색 과육 복숭아 품종, ‘조생혈도(Josanghyeoldo)’와 백색 과육 복숭아 품종인 ‘미백도(Mibaekdo)’의 과실을 시기별로 채취하여 액체질소를 사용하여 분쇄하였다. 만개 후 8 주부터 과실을 수확하여 1 주 간격으로 ‘조생혈도’는 5 회, ‘미백도’는 10 회 수확하여 시기별 시료의 RNA를 분리하였다. RNA 분리 방법은 pine tree methods (Shujun et al., 1993)에 따라서 수행하였다.
‘조생혈도’와 ‘미백도’의 RNA–Seq data를 이용한 SNP 분석
‘조생혈도’와 ‘미백도’ 전사체 분석 결과(Kim et al., 2012)에서 찾은 SNP를 매트릭스로 만들어 변이 과정을 거친 후 동형(homozygous) SNP와 이형(heterozygous) SNP로 나누었고, SNP 선발 조건은 3 이상으로 하였다. ‘조생혈도’와 ‘미백도’의 SNP 비교분석은 동일한 SNP 위치에서 두 시료 간에 서로 동형 SNP인 경우만을 선발하였다. GO분석은 TAIR10_ID을 이용하여 DAVID (http://david.abcc.ncifcrf.gov/tools.jsp)에서 제공하는 Gene Ontology Annotation Tool을 사용하였다. 참조한 유전체의 유전자는 선택적인 splicing form을 포함하고 있기 때문에 1개의 SNP 위치는 1개 이상의 전사체에 포함될 수 있으며, 이후 gene ontology 분석 및 SNP 프라이머 디자인은 다형성을 나타내는 SNP 104개만을 사용하여 분석을 진행하여 72개의 HRM 프라이머 조합을 제작하였다(Table 1).
Table 1. Summary of the development of SNP markers in 'Josanghyeoldo' and 'Mibaekdo'
| Sample | Total SNP | Homozygous SNP | Heterozygous SNP | Polymorphism SNP | HRM primer |
| Josanghyeoldo | 265 | 265 | 256 | 104 | 72 |
| Mibaekdo | 346 | 346 | 437 |
HRM (High Resolution Melting) analysis
HRM 분석(Wittwer et al., 2003)을 위한 1차 gDNA 추출은 적색 과육 복숭아 6 품종 흑고랑(黑姑娘, Heiguniang), 홍도(紅桃, Hongtao), 조생혈도(Josanghyeoldo), 홍항(紅港, Redhaven), 스칼렛펄(Scarlet Pearl), 예품도(禮品桃, Lipintao) 백색 과육 복숭아 6 품종 감조백도(Kansuke Hakuto), 천중도백도(Kawanakajima Hakutou), 미백도(Mibaekdo), 미홍(Mihong), 영봉(Reihou), 유미(Yumi)에서 잎을 채취하여 DNeasy Plant kit (Qiagen)을 사용하여 gDNA를 분리하였다(Prince et al., 1997). 2차 HRM 분석을 위한 gDNA 추출은 적색 과육 복숭아 8 품종 흑고랑 우량계(黑姑娘 優良系, Selected Heiguniang), 흑도황후(黑桃皇后, Heitaohuanghou), 흑고랑(黑姑娘, Heiguniang), 홍도(紅桃, Hongtao), 조생혈도(Josanghyeoldo), 홍항(紅港, Redhaven), 스칼렛펄(Scarlet Pearl), 예품도(禮品桃, Lipintao), 백색 과육 복숭아 22 품종 백향(Baekhyang), 붉은백도(Bulgeonbaekdo), 이노센스(Innocence), 진미(Jinmi), 한비도(Kanhito), 천중도백도(Kawanakajima Hakuto), 미백도(Mibaekdo), 미홍(Mihong), 미스홍(Misshong), 서반3호(瑞蟠3號, Ruipan3), 설홍(Seolhong), 선미(Seonmee), 수미(Soomee), 스프링타임(Spring Time), 트로픽스노우(Tropic Snow), 쓰쿠바 No.4 (Tsukuba No.4), 하수성꽃복숭아 OP4 (Weeping Peach OP4), 유미(Yumi), 유명(Yumyeong), 7-2-1-24, 7-2-1-50, 7-3-1-14에서 추출한 DNA는 NanoDrop ND-1000 (Nanodrop Technologies, USA)를 이용하여 정량하고 최종 농도가 20 ng이 되도록 희석하였다. HRM 분석은 7500 Fast Real-time PCR machine (Applied BioSystems)을 사용하여 수행하였다. PCR 반응액은 gDNA 20 ng에 각 SNP 분자표지에 해당하는 1쌍의 프라이머를 넣고 PCR 반응은 95℃에서 초기 변성을 수행한 뒤, 95℃에서 15초간 변성과 60℃에서 60초간 복원하는 과정을 40회 반복하였다. 마지막으로 95℃에서 10초간 변성 반응을 시킨 후, 60℃에서 95℃까지 0.2℃씩 올리면서 각 온도에서 5초간 유지하는 동안 형광을 측정하여 HRM melting 그래프를 그렸다.
결과 및 고찰
복숭아 RNA-Seq data를 이용한 SNP 분석
복숭아 반수체 품종 ‘Lovell’의 유전체 염기서열이 공개된 이후에 본 논문에서 사용한 Roche 454와 Illumina/Solexa를 사용하여 복숭아 육종에 사용할 수 있는 6,654 SNP data set를 보고한 결과가 있으며(Ahmad et al., 2011), 유전자 지도 작성과 복잡한 농업적 특성에 유용하게 사용할 수 있는 9,000 SNPs를 보고한 연구 결과를(Verde et al., 2012) 참조하여 SNP 분석을 수행하였다. 본 연구의 목적인 적색 과육 복숭아 품종과 백색 과육 복숭아 품종을 구분할 수 있는 분자표지를 개발하기 위해서 복숭아 NGS 데이터 결과(Kim et al., 2012)를 참조 유전체에 매핑(mapping)하여 genome wide SNP를 찾고, 적색 과육 품종인 ‘조생혈도’와 백색 과육 품종인 ‘미백도’에서 다형성을 나타내는 SNP를 선발하였다. 사용한 chromosome 8개의 총 길이는 218,265,032 bp이고, 유전자 개수는 27,256개, 유전자의 총 길이는 71,294,128 bp, 전사체 수는 28,087개, 전사체의 크기는 37,560,900 bp이다. 참조 유전체 대비, 배열 결과 ‘조생혈도’는 정리된 read 중에서 44.35 %인 198,555 read가 매핑되었고, ‘미백도’는 38.88 %인 102,481 read가 매핑되었다. ‘조생혈도’와 ‘미백도’에서 찾은 SNP를 매트릭스로 만들어 변이 과정을 거친 후, 동형 SNP와 이형 SNP로 나누어 정리하였고(Table 1), SNP 선발 조건은 3 이상으로 하였다. ‘조생혈도’와 ‘미백도’의 SNP 비교분석은 동일한 SNP 위치에서 두 품종간에 서로 동형 SNP인 경우만을 선발하였고, 그 결과 다형성을 나타내는 104개의 SNP를 선발하였다. ‘조생혈도’와 ‘미백도’ 간에 탐색되는 SNP 104개의 주석(JGI Phytozome)을 찾은 결과 전사체 수는 105개이고, TAIR10_ID 기준으로는 89개이다. SNP 수와 전사체 수가 다른 이유는 참조 유전체의 유전자는 한 개의 SNP 위치가 한 개 이상의 전사체에 포함될 수 있기 때문이다.
HRM 분석을 이용한 SNP 분자표지 개발
‘조생혈도’와 ‘미백도’ 간에 탐색되는 polymorphism SNP 104개 중에서 72개의 프라이머 조합을 이용하여 HRM 분석을 하였다(Table 2). 72개의 SNP 분자표지의 유전자 정보를 gene ontology 분석 결과 기능별로 차지하는 유전자군을 보면 생화학 수준에서 유전자 생산물에 관여하는 molecular function에 해당하는 유전자 14개, 생물학적으로 다양한 목적에 관여하는 biological process 3개, 유전자 생산물의 위치에 관여하는 cellular component 1개, 나머지는 gene ontology 분석에서 분류되지 않았다. 색소 생성에 관련된 유전자들 중에서 chalcone synthase (CHS), flavanone 3-hydroxylase (F3H), dihydroflavonol 4- reductase (DFR), leucoanthocyanidin dioxygenase (LDOX), UDP glucose: flavonoid 3-O-glucosyltransferase (UFGT)는 적색 과육 복숭아와 백색 과육 복숭아에서 발현 차이가 명확하게 나타나지만(Kim et al., 2012), 염기서열은 동일하였다. 이러한 결과는 배나무에서도 관찰 되는데 적색 과피 배와 녹색 과피 배(돌연변이 계통)의 anthocyanin 합성 관련 유전자들의 염기 서열을 분석해 보면 다형성이 없음을 확인할 수 있다(Yang et al., 2013). 72개의 유전자 중에서 색소 생성에 관련된 유전자는 cytochrome P450, family 82, subfamily G, polypeptide 1으로 flavonoid 생합성 또는 leucodelphinidin, leteolin 생합성에 관여하는 것으로 알려져 있다(Matsubara et al., 2005). 이 유전자로부터 유래한 SNP ID, ppa004569m 분자표지는 복숭아 적색 과육 6 품종과 백색 과육 6 품종이 구분되지 않았다. 이러한 결과는 색소 생성 및 발현 과정 이전 단계에서 조절되는 것으로 생각할 수 있다. 72개의 SNP 분자표지 중에서 과육 색에 따른 복숭아 품종을 적색과 백색 2군으로 구분할 수 있는 SNP 분자표지는 1종이었다(Table 2). SNP ID, ppa012770m의 유래는 chaperone DnaJ-domain superfamily protein으로 단백질 결합, 분해, 수송 그리고 다양한 전사 및 복제 인자의 활성화에 관여하는 것으로 알려져 있으며(Cheetham and Caplan, 1998), 이 유전자는 linkage group 7에 위치한다. 이 유전자의 기능은 안토시아닌 생합성에서 적색소 생성과는 직접적으로 관계가 없지만 다양한 전사 및 복제 인자의 활성화에 관여하는 것으로 알려져 있다. 색소 발현에 관련된 다른 과종의 연구 결과를 보면 과실이 성숙해지면 적색이 사라지는 배 품종인 ‘Red Bartlett’ 과 적색이 변하지 않는 ‘Starkrimson’의 비교에서 색소가 사라지는 이유가 액포로 anthocyanin이 이동하기 때문이며 이와 관련해서 실제로 색소 발현에 직접적으로 관여하지 않는 vacuolar peroxidases 유전자를 전사체 분석 결과 중요한 유전자로 지목하고 있다(Wang et al., 2017). SNPID, ppa012770m의 SNP 차이는 ‘조생혈도’에서는 A 염기서열이 ‘미백도’에서 G로 바뀌었고, 1차 HRM 분석에서 적색 과육 복숭아 6 품종과 백색 과육 복숭아 6 품종을 구분할 수 있었다(Fig. 1). 1차 HRM 분석에서 적색 과육 및 백색 과육 복숭아의 시료수가 12종으로 많지 않아서 SNP 분자표지 활용 가능성을 재확인하기 위해 2차 HRM 분석에서는 적색 과육 복숭아 8 품종, 백색 과육 복숭아 22 품종을 분석하였다. 적색 과육 복숭아 시료가 적은 이유는 적육계 특성을 보이는 유전자원의 수가 많지 않고, 대체로 산미가 높아 품종 개발이 활발하게 이루어지지 않고 있기 때문이다. SNP ID, ppa012770m에서 유래한 프라이머 조합을 이용하여 2차 HRM 분석 결과 적색 과육 복숭아 8 품종, 백색 과육 복숭아 21 품종을 구분할 수 있었고, 백색 과육 품종인 ‘미스홍’은 적색 및 백색 과육 복숭아에 들어가지 않고 또 다른 그룹으로 나뉘었다(Fig. 2). 실제로 백색 과육 품종인 ‘진미’에서 적색소 발현이 많이 되어서 HRM 분석을 하였지만 ‘진미’ 품종은 백색 과육 품종 그룹에 속하였다.
Table 2. PCR amplicons showing polymorphic HRM profiles in peach cultivars
yVar.:Variation, Reference:Josanghyeoldo/Mibaekdo.
xChr.:Reference genome (Prunus persica) chromosome.
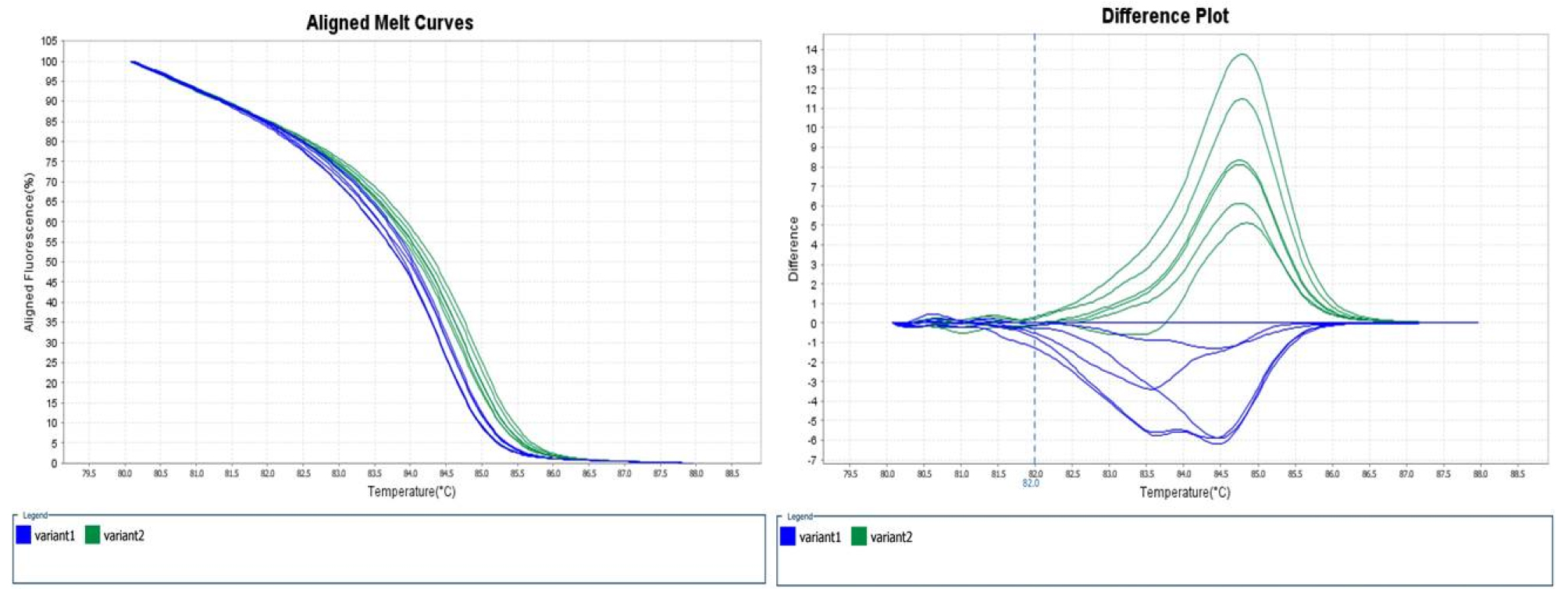
Fig. 1.
HRM analysis of 6 red-fleshed peach cultivars (Heiguniang, Hongtao, Josanghyeoldo, Redhaven, Scarlet Pearl, Lipintao) and 6 white-fleshed peach cultivars (Kansuke Hakuto, Kawanakajima Hakutou, Mibadkdo, Mihong, Reihou, Yumi). (A) Normalized graph of SNP ID, ppa012770m. (B) Difference graph of SNP ID, ppa012770m. Green variant2 indicate red-fleshed peach cultivars and blue variant1 indicate white-fleshed peach cultivars.
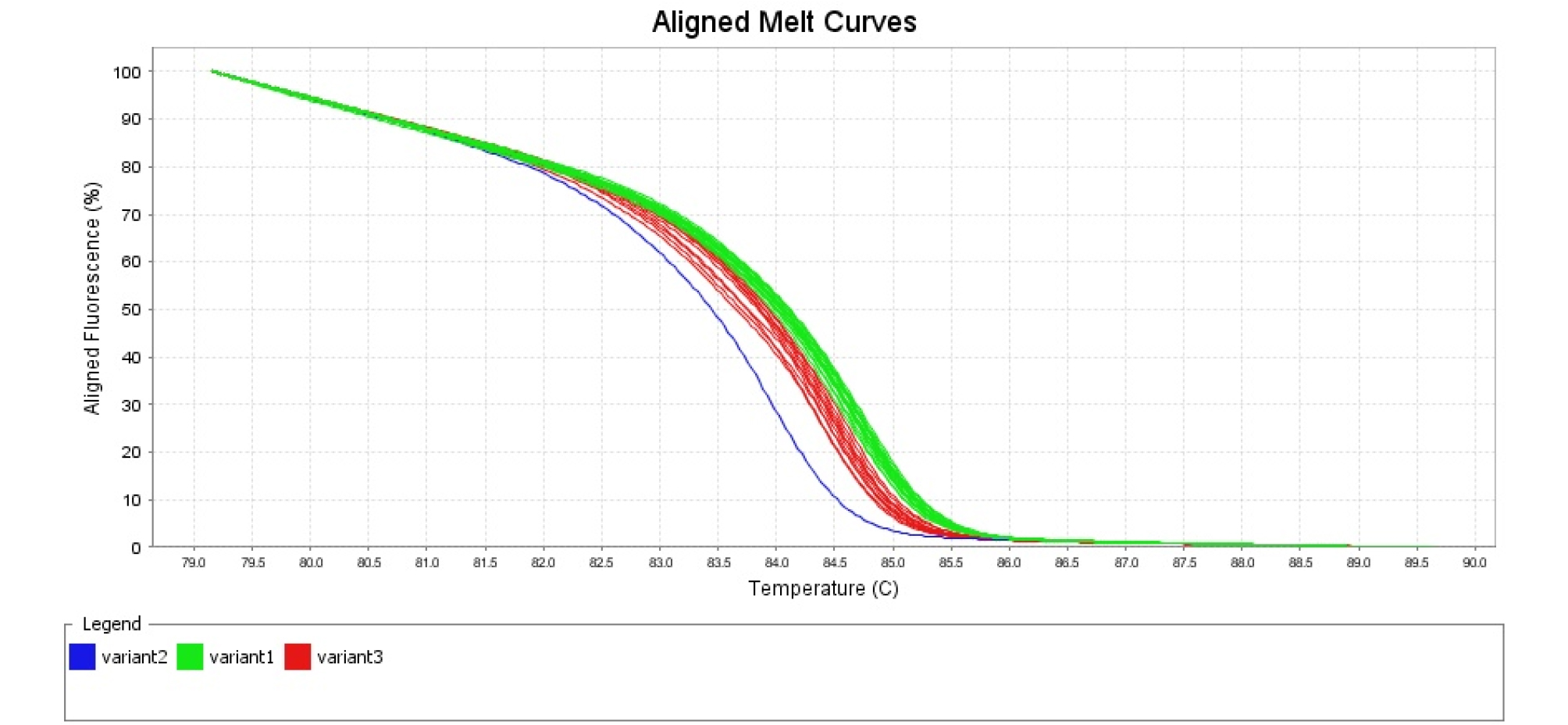
Fig. 2.
HRM analysis of 8 red-fleshed peach cultivars (Selected Heiguniang, Heitaohuanghou, Heiguniang, Hongtao, Josanghyeoldo, Redhaven, Scarlet Pearl, Lipintao) and 22 white-fleshed peach cultivars (Baekhyang, Bulgeonbaekdo, Innocence, Jinmi, Kanhito, Kawanakajima Hakutou, Mibadkdo, Mihong, Misshong, Ruipan3, Seolhong, Seonmee, Soomee, Spring Time, Tropic Snow, Tsukuba No.4, Weeping Peach OP4, Yumi, Yumyeong, 7-2-1-24, 7-2-1-50, 7-3-1-14). Red variant3 indicate red-fleshed peach cultivars and green variant1 indicate white-fleshed peach cultivars, blue variant2 indicate ‘Misshong’ in white-fleshed peach cultivars.
본 연구에서는 RNA-Seq data를 사용하여 복숭아 적육계와 백육계를 구분할 수 있는 SNP 분자표지를 개발하고자 하였다. 본 연구에서 개발한 1종의 SNP 분자표지가 적육계 복숭아 품종과 백육계 복숭아 품종을 구분할 수 있는 정확한 기작에 대해서는 계속적인 연구가 필요하며 ‘미스홍’ 품종이 백육계 품종임에도 불구하고 백육계 및 적육계 품종 그룹에 포함되지 않고 또 다른 그룹으로 분류되는지에 대한 연구도 필요하다. 또한 다양한 유전자원과 교배집단에 적용하여 조기 선발 분자표지로서의 가능성이 있는지 확인할 필요가 있다. 고부가 기능성 복숭아 품종을 육성하는데 있어 전통육종방법은 상품성이 있으면서 적육색과 같은 특정형질만 발현시키는 데 시간과 노력이 필요하다. 복숭아나무는 재식 후 2~3년이 경과되어야 과실의 특성을 볼 수 있기 때문에 적육계 계통 선발을 위한 교배 집단에서 분자표지의 개발은 육종 연한을 단축시킬 수 있는 중요한 수단이다. 본 연구에서는 고부가 기능성 품종으로 복숭아 적육계 계통을 육성하기 위해 전사체 분석방법으로 적육계와 백육계에서 발현량이 다른 유전자들을 분석하였고, HRM 방법을 이용한 SNP 분자표지를 개발하여 복숭아 유전자원 및 육성품종, 3개의 선발 계통 등 32 품종에 적용함으로써 과육색을 구분할 수 있는 분자표지의 가능성을 검토하였다. 본 연구에서 개발한 SNP 분자표지의 활용을 위해서는 더 많은 수의 복숭아 유전자원 및 적색 과육 품종과 백색 과육 품종 간 교배로 얻어진 실생 집단에 적용해서 재현성을 확인해야 하며, 이러한 연구 결과는 적색 과육 복숭아 신품종의 육종 기한을 단축시키고 교배 육종의 효율성을 높이는데 이용할 수 있을 것이다.
적 요
과피와 과육의 다양한 색은 복숭아 분류에 가장 널리 사용되는 상업적 기준 중 하나이다. 새로운 적색 과육 품종을 육성하기 위해서는 많은 교배 조합과 세대가 진전되어야 한다. 따라서 육종 효율을 높이기 위해서는 목적 형질을 가진 개체에 적용할 조기 선발 분자표지를 개발할 필요가 있다. 과육색이 다르게 발현되는 복숭아 품종의 유전자 발현을 비교하기 위해 2개의 cDNA library를 제작하였다. 적색 과육 품종인 ‘조생혈도’와 백색 과육 품종인 ‘미백도’의 유전자 발현 차이를 보기 위해 차세대 염기서열 분석(NGS) 기술을 사용하였고, 두 품종으로부터 얻은 EST의 염기서열을 결정하고 기존에 보고된 유전자와의 상동성을 분석하였다. ‘조생혈도’와 ‘미백도’의 EST database로부터 72쌍의 SNP 분자표지를 선발하였고, 적색 과육 품종 8개와 백색 과육 품종 24개를 구분할 수 있는 SNP 분자표지를 HRM 방법으로 분석하였다. 본 연구에서는 복숭아 EST database를 기반으로 HRM 분석 방법을 이용하여 복숭아 품종의 적육계와 백육계를 구분할 수 있는 효율적인 SNP 분자표지를 개발하였다. 이러한 SNP 분자표지는 복숭아 육종에 유용하게 사용할 수 있으며, 복숭아 품종의 다양한 색 변화에 관한 분자 기작 연구에 좋은 참고자료가 될 수 있을 것이다.




