서 언
재료 및 방법
식물 재료와 RNA, DNA 분리
Next generation sequencing과 bioinformatics analysis
RNA–Seq data를 이용한 SNP 분석
HRM 분석
결과 및 고찰
‘장호원황도’와 ‘미백도’의 RNA-Seq data 분석
RNA-Seq data를 이용한 SNP 분석
HRM 분석을 이용한 품종 선발용 SNP 마커
적 요
서 언
복숭아는 중국이 원산지인 온대 낙엽성 과수로 장미과(Rosaceae) 자두속(Prunus) 복숭아아속(Amygdalus)에 속하며 세계적으로 널리 재배되고 있다(Ahmad et al., 2004). 복숭아는 2배체로 같은 장미과에 속하는 사과(742 Mbp)에 비해 유전체 크기가 220~230 Mbp 정도로 작아(Dhanapal and Crisosto, 2013) 장미과 유전체 연구의 소재로 활용되고 있다. 2000년도부터 시작되었던 Prunus species의 염기서열 분석은 미국, 이탈리아, 칠레, 스페인, 프랑스가 참여하는 IPGI (International Peach Genome Initiative)에 의해 유전체 분석 결과가 2013년에 발표되었다(Verde et al., 2013). 이러한 복숭아 유전체 분석결과를 통해 유전자형과 표현형 사이의 관계를 해석하고 유전적 측면에서 구조와 기능을 이해할 수 있게 되었다.
복숭아 육종 프로그램은 소비자의 다양한 요구를 충족하기 위해 진행되어 왔으며, 최근에는 과실의 기능성에 대한 요구가 증대됨에 따라 기능성이 품종 육성의 중요한 육종 목표의 하나가 되었다. 복숭아 육종의 소재가 되는 유전자원들은 수확기 과육색을 기준으로 백육계, 적육계 및 황육계로 분류되며 백육계와 적육계를 구분할 수 있는 SNP 마커가 개발되었다(Kim et al., 2019). 또한 복숭아는 폴리페놀 함량이 높아 항암효과가 있는 것으로 알려져 있으며 황육계와 적육계가 백육계에 비해 카로티노이드와 안토시아닌이 많아 항산화 성분 등 기능성 물질의 함량이 높다(Brandi et al., 2011). 우리나라에서는 주로 백육계 품종이 재배되고 있지만, 기능성 물질 함량 증진을 위해서 황육계 및 적육계 같은 유색 품종 육성의 중요도가 높아지고 있어(Cevallos-Casala et al., 2006), 최근 유색 품종의 색소 발현에 관련되어있는 카로티노이드, 또는 플라보노이드 성분에 관한 유전자 발현 연구도 활발하게 진행되고 있다(Forkmann and Martens, 2001). 복숭아 과육색을 조절하는 것은 linkage group 1에서 Y (yellow)로 명명되어 있는 유전자좌이며(Bliss et al., 2002), 백색이 황색에 대해서 우성으로 멘델의 법칙을 따른다(Connors, 1920). 백육계 복숭아에서 카로티노이드 분해에 관여하는 주요한 요인은 carotenoid cleavage dioxygenase 4 (CCD4)로 알려져 있으며, SSR 마커로 개발하여 활용하고 있다(Falchi et al., 2013).
본 연구는 기존에 보고된 CCD4 유전자에서 유래한 SSR 마커 외에 황육계 복숭아를 조기 선발할 수 있는 마커 개발을 위해 황육계 복숭아 ‘장호원황도’와 백육계 품종인 ‘미백도’의 EST 분석 결과를 바탕으로 카로티노이드 발현에 관여하는 유전자들과 품종 간 발현량 차이가 큰 유전자들을 비교 분석하였다. HRM 분석방법을 사용하여 황육계 품종과 백육계 품종의 PCR 증폭산물에서 SNP 위치를 확인하고, 다양한 복숭아 유전자원과 육성품종 및 계통 중에서 황육계 품종과 백육계 품종을 구분할 수 있는 SNP 마커를 개발하고자 하였다.
재료 및 방법
식물 재료와 RNA, DNA 분리
본 연구에 사용한 재료는 농촌진흥청 국립원예특작과학원에 재식되어 있는 황육계 복숭아 품종인, ‘장호원황도(Changhowon Hwangdo)’와 백육계 복숭아 품종인 ‘미백도(Mibaekdo)’의 과실을 만개 후 8주부터 1주 간격으로 7회 수확하여 액체질소를 사용하여 분쇄하였다. ‘장호원황도’는 2019년 7월 22일, ‘미백도’는 7월 6일의 시료에서 pine tree methods (Shujun et al., 1993)에 따라서 RNA를 분리하였다. HRM 분석(Wittwer et al., 2003)을 위한 gDNA 추출은 황육계 17 품종 [‘금수(Jinxiu)’, ‘등염(Chengyan)’, ‘롱의택골드(Takinosawa Gold)’, ‘선골드(Sun Gold)’, ‘선프레(Sunfre)’, ‘수홍(Suhong)’, ‘오헨리(O’Henry)’, ‘장호원황도(Changhowon Hwangdo)’, ‘조생유도반도(Zaoshengyoutaopantao)’, ‘준프린스(Juneprince)’, ‘찌요마루(Chiyomaru)’, ‘천홍(Cheonhong)’, ‘하홍(Hahong)’, ‘환타지아(Fantasia)’, ‘황후(Hwanghoo)’, ‘히타치레드(Hitachired)’, 7-2-1-102(산리황금도×진미)] 백색 과육 복숭아 22 품종 [‘미백도(Mibaekdo)’, ‘미스홍(Misshong)’, ‘미홍(Mihong)’, ‘백향(Baekhyang)’, ‘붉은백도(Bulgeonbaekdo)’, ‘서반3호(Ruipan3)’, ‘선미(Seonmee)’, ‘수미(Soomee)’, ‘스프링타임(Spring Time)’, ‘쓰쿠바 No.4 (Tsukuba No.4)’, ‘설홍(Seolhong)’, ‘이노센스(Innosense)’, ‘유명(Yumyeong)’, ‘유미(Yumi)’, ‘진미(Jinmi)’, ‘천중도백도(Kawanakajima Hakutou)’, ‘트로픽스노우(Tropic Snow)’, ‘하수성꽃복숭아계통4 (Weeping Peach OP4)’, ‘한비도(Kanhito)’, 7-2-1-24(진미 자연교잡실생), 7-2-1-50 (진미 자연교잡실생), 7-3-1-14 (감조백도×미스리까)]에서 잎을 채취하여 DNeasy Plant kit (Qiagen)을 사용하여 분리하였다(Prince et al., 1997).
Next generation sequencing과 bioinformatics analysis
분리한 RNA는 cDNA 합성과정을 거쳐 high throughput sequencing 방법(Margulies et al., 2005)으로 Roche GS FLX를 사용하였고, 생성된 염기서열은 Roche GS FLX software (2.6 version)를 이용해 de novo transcriptome 조립을 진행하였다. 생성된 isotig들은 blast (2.2.25+ version) (Altschul et al., 1990)를 이용해 GO database (Ashburner et al., 2000)와 NCBI의 NR database에 alignment를 수행하였다. Singleton에 대해서는 SeqClean과 Lucy (Hui-Hsien and Michael, 2001) software를 이용하여 sequence trimming을 진행하였고 isotig와 같은 방법으로 blast를 이용하여 GO 및 NR DB에 alignment를 수행하였다.
RNA–Seq data를 이용한 SNP 분석
‘장호원황도’와 ‘미백도’ 전사체 분석 결과에서 찾은 SNP를 매트릭스로 만들어 변이 과정을 거친 후 homozygous SNP와 heterozygous SNP로 나누었고, SNP 선발 조건은 3 이상으로 하였다. ‘장호원황도’와 ‘미백도’의 SNP 비교 분석은 동일한 SNP 위치에서 두 시료 간에 서로 homozygous SNP인 경우만을 선발하였다. GO분석은 TAIR10_ID을 이용하여 DAVID에서 제공하는 Gene Ontology Annotation Tool을 사용하였다. 참조한 유전체의 유전자는 선택적인 splicing form을 포함하고 있기 때문에 1개의 SNP 위치는 1개 이상의 전사체에 포함될 수 있으며, 이후 gene ontology 분석 및 SNP 프라이머 디자인은 다형성을 나타내는 SNP 486개만을 사용하여 분석을 진행하여 238 개의 HRM 프라이머 조합을 제작하였다(data not shown).
HRM 분석
HRM 분석(Wittwer et al., 2003)은 7500 Fast Real-time PCR machine (Applied BioSystems)을 사용하여 수행하였다. PCR 반응은 gDNA 20 ng에 각 SNP 마커에 해당하는 1쌍의 프라이머를 넣고 95℃에서 초기 변성을 수행한 뒤, 95℃에서 15초간 변성과 60℃에서 60초간 복원하는 과정을 40회 반복하였다. 마지막으로 95℃에서 10초간 변성 반응 후, 60℃에서 95℃까지 0.2℃씩 올리면서 각 온도에서 5초간 유지하는 동안 형광을 측정하여 HRM melting 그래프를 그렸다.
결과 및 고찰
‘장호원황도’와 ‘미백도’의 RNA-Seq data 분석
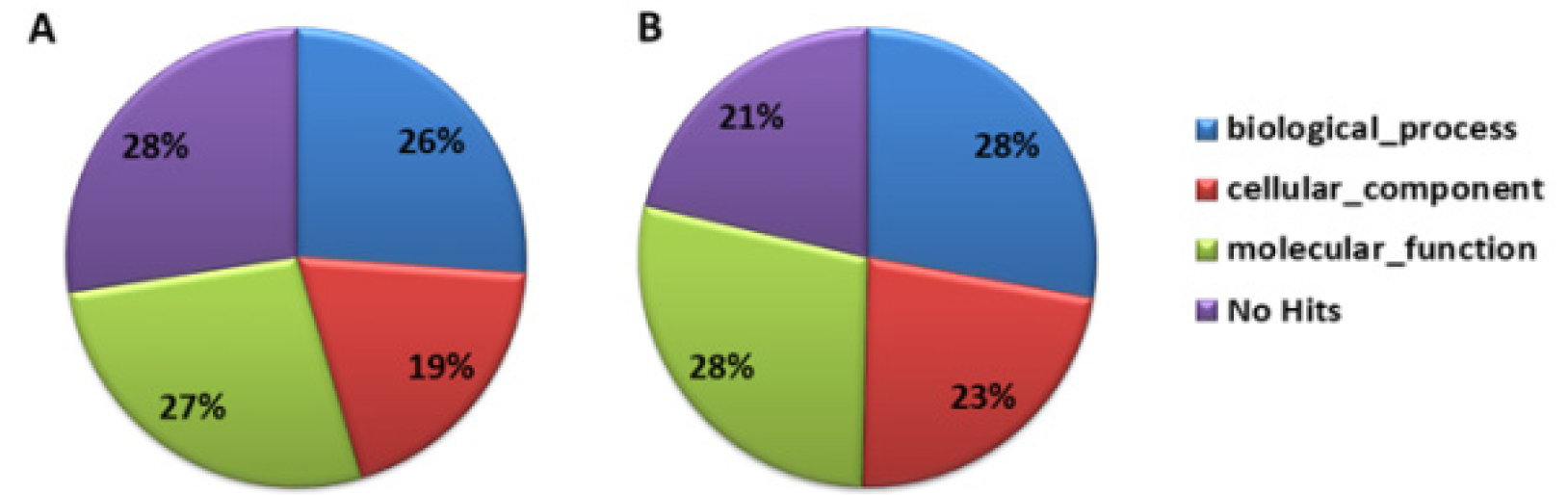
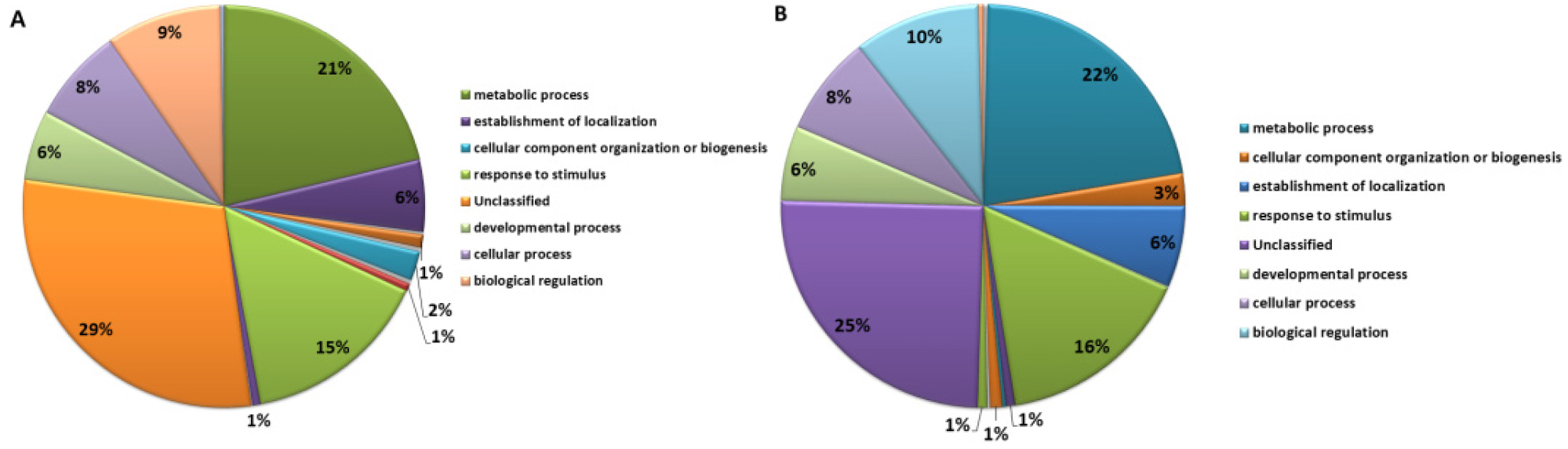
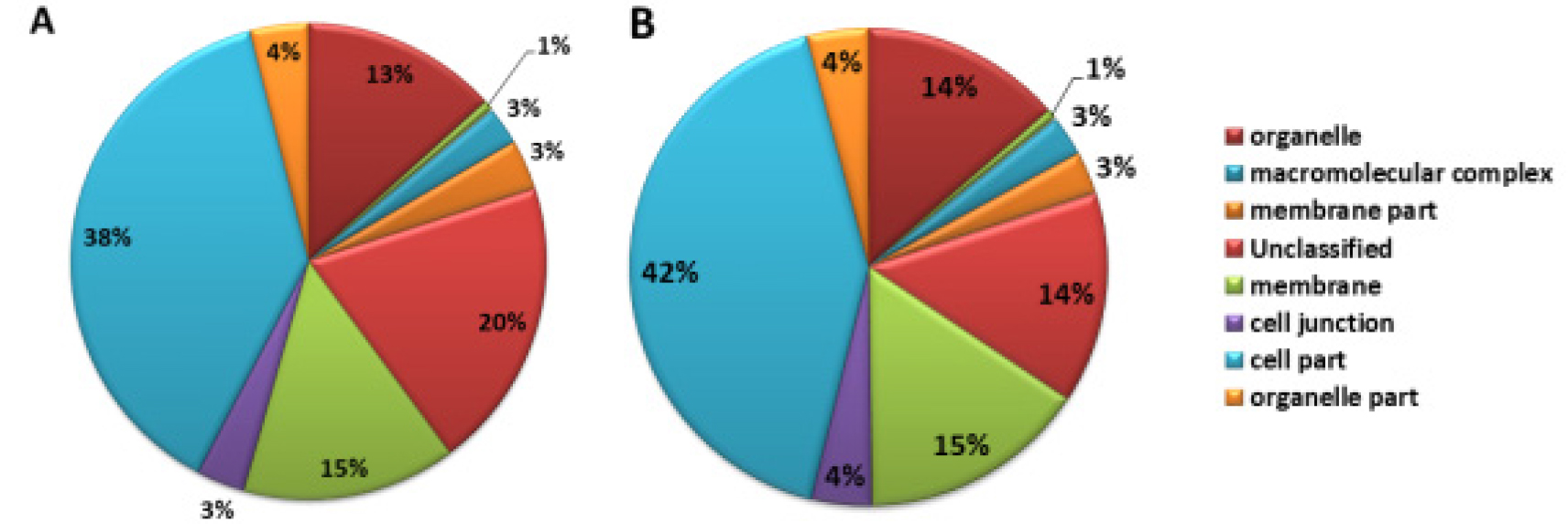
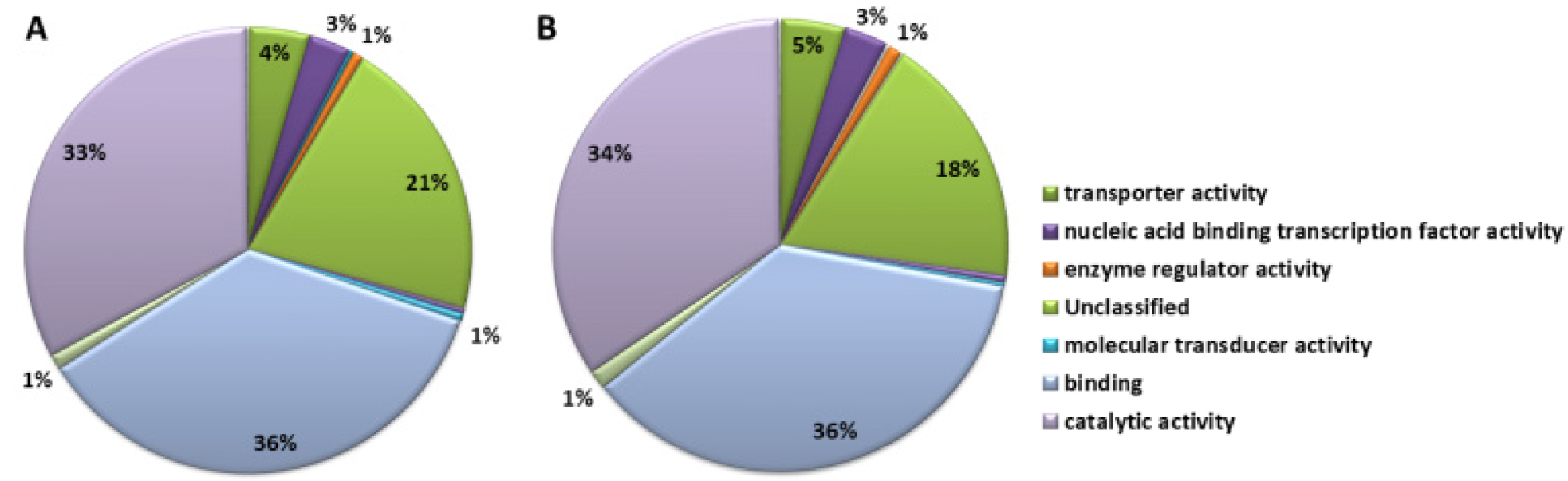
‘장호원황도’와 ‘미백도’의 RNA-Seq data 분석 결과, ‘장호원황도’의 총 read 수는 603,605, 총 염기서열 길이는 225,835, 526 bp, read의 평균 염기서열 길이는 374bp였다. ‘미백도’의 총 read 수는 620,970, 총 염기서열 길이는 221,772,064 bp, read의 평균 염기서열 길이는 357 bp였다(Table 1). ‘장호원황도’의 gene ontology 분석 결과 기능별로 차지하는 유전자군을 보면 생물학적으로 다양한 목적에 관여하는 biological process는 26%, 유전자 생산물의 위치에 관여하는 cellular component를 구성하는 유전자는 19%, 생화학수준에서 유전자 생산물에 관여하는 molecular function에 해당하는 유전자가 27%를 차지하고 있었으며, 보고된 유전자와 상동성을 나타내지 않는 유전자가 28%였다. 이에 비해 ‘미백도’는 biological process에 관여하는 유전자가 28%, cellular component는 23%, molecular function에 해당하는 유전자가 28%, 보고된 유전자와 상동성을 나타내지 않는 유전자가 21%였다(Fig. 1). 두 품종 간에 차이가 나는 부분은 생산물의 위치에 관여하는 cellular component를 구성하는 유전자로 ‘장호원황도’는 19%인데 비해서 ‘미백도’는 23%로 조금 더 많은 유전자가 알려져 있고, 보고되지 않은 유전자의 비율도 ‘장호원황도’가 28%로 높게 나타났다. Biological process에 해당하는 유전자를 세분해 보면 ‘장호원황도’의 경우 unclassified가 29%, metabolic process 21%, response to stimulus 15%, biological regulation 9%, cellular process 8%, developmental process 6%, establishment of localization 6%, cellular component organization 또는 biogenesis 2%였고, ‘미백도’의 경우 unclassified가 25%, metabolic process 22%, response to stimulus 16%, biological regulation 10%, cellular process 8%, developmental process 6%, establishment of localization 6%, cellular component organization 또는 biogenesis 3%였다(Fig. 2). Biological process에 관여하는 유전자의 비율을 보면 전체 염기서열 분석 결과에서처럼 ‘장호원황도’가 ‘미백도’ 보다 알려지지 않은 유전자가 많은 것으로 보아 주로 재배되고 있는 백육계 품종의 연구가 더 많이 보고된 것을 알 수 있다. 유전자 생산물의 위치에 관여하는 cellular component에 해당하는 유전자를 세분해 보면 ‘장호원황도’의 경우 cell part 38%, unclassified 20%, membrane 15%, organelle 13%, organelle part 4%, cell junction 3%, membrane part 3%, macromolecular complex 3%였고, ‘미백도’의 경우 cell part 42%, unclassified 14%, membrane 15%, organelle 14%, organelle part 4%, cell junction 4%, membrane part 3%, macromolecular complex 3%로 cell part와 unclassified를 제외하고는 ‘장호원황도’와 비슷한 유전자 분포를 보이고 있었다(Fig. 3). 생화학 수준에서 유전자 생산물에 관여하는 molecular function에 해당하는 유전자를 세분한 결과 ‘장호원황도’는 binding 36%, catalytic activity 33%, unclassified 21%, transporter activity 4%, nucleic acid binding transcription factor activity 3%, enzyme regulator activity 1%, molecular transducer activity 1%였고, ‘미백도’는 binding 36%, catalytic activity 34%, unclassified 18%, transporter activity 5%, nucleic acid binding transcription factor activity 3%, enzyme regulator activity 1%, structural molecule activity 1%였다(Fig. 4). 생물학적으로 다양한 목적에 관여하는 biological process와 유전자 생산물의 위치에 관여하는 cellular component를 구성하는 유전자 분포는 두 품종 간에 차이가 있었지만 생화학수준에서 유전자 생산물에 관여하는 molecular function에 해당하는 유전자 분포는 차이가 없었고, 전체적인 전사체 분석 결과 황육계 품종인 ‘장호원황도’ 보다 백육계 품종인 ‘미백도’에서 유전자 분석 연구결과가 더 많이 보고되었음을 알 수 있었다.
Table 1.
Summary of RNA-Seq data in ‘Changhowon Hwangdo’ and ‘Mibaekdo’
| Total read | Total base pair | Average length | |
| Changhowon Hwangdo | 603,505 | 225,865,526 | 374 |
| Mibaekdo | 620,970 | 221,772,064 | 357 |
RNA-Seq data를 이용한 SNP 분석
Roche 454와 Illumina/Solexa를 사용하여 복숭아 반수체 품종 ‘Lovell’의 유전체 염기서열이 공개된 이후에 복숭아 육종에 사용할 수 있는 6,654 SNP data set를 보고한 결과가 있으며(Ahmad et al., 2011), 유전자 지도 작성과 농업적 특성에 유용하게 사용할 수 있는 9,000 SNPs를 보고한 연구 결과를(Verde et al., 2012) 참조하여 SNP 분석 후 ‘장호원황도’와 ‘미맥도’의 RNA-seq data 결과를 참조 유전체에 매핑(mapping)하여 genome wide SNP를 찾고, 황육계 품종인 ‘장호원황도’와 백육계 품종인 ‘미백도’에서 다형성을 나타내는 SNP를 선발하였다. 사용한 chromosome 8개의 총 길이는 218,265,032 bp이었고, 유전자 개수는 27,256개, 유전자의 총 길이는 71,294,128 bp, 전사체 수는 28,087개, 전사체의 크기는 37,560,900 bp였다. 참조 유전체 대비, 배열 결과 ‘장호원황도’는 정리된 read 중에서 225,835,526 bp를 확보하여 77.96%가 매핑되었고, ‘미백도’는 221,772,064 bp를 확보하여 75.94%가 매핑되었다(Table 2). ‘장호원황도’와 ‘미백도’에서 찾은 SNP를 매트릭스로 만들어 변이 과정을 거친 후, homozygous SNP와 heterozygous SNP로 나누어 정리하였다(Table 3). ‘장호원황도’와 ‘미백도’의 SNP 비교분석에서 다형성을 나타내는 486개의 SNP가 선발되었고, 주석(JGI Phytozome)을 찾은 결과 전사체 수는 365개였고, TAIR10_ID 기준으로는 330개였다.
Table 2.
Reference genome of Prunus persica and mapping rate of ‘Changhowon Hwangdo’ and ‘Mibaekdo’
Table 3.
Summary of the development of SNP markers in ‘Changhowon Hwangdo’ and ‘Mibaekdo’
|
Total SNP |
Homozygous SNP |
Heterozygous SNP |
Polymorphism SNP |
HRM primer | |
| Changhowon Hwangdo | 4,071 | 1,529 | 2,542 | 486 | 238 |
| Mibaekdo | 4,308 | 2,010 | 2,298 |
HRM 분석을 이용한 품종 선발용 SNP 마커
‘장호원황도’와 ‘미백도’ 간에 다형성을 보인 SNP 486개 중에서 238개의 프라이머 조합을 이용하여 HRM 분석을 하였다. 238개의 SNP 마커의 유전자 정보를 gene ontology 분석 결과 기능별로 차지하는 유전자군을 보면 생물학적으로 다양한 목적에 관여하는 biological process 17개, 유전자 생산물의 위치에 관여하는 cellular component 12개, 생화학 수준에서 유전자 생산물에 관여하는 molecular function에 해당하는 유전자군 5개로 분류되었다. 카로티노이드 색소 생성에 관련된 유전자들 중에서 phytoene synthase (PSY), phytoene desaturase (PDS), carotenoids isomerase (CRITISO), nonheme diiron carotene hydroxylase (HYD), carotenoid cleavage dioxygenase (CCD)는 ‘장호원황도’와 ‘미백도’에서 발현 차이가 있지만, 염기서열은 동일하였다. 이러한 결과는 적육계 복숭아와 백육계 복숭아에서도 관찰되는데 ‘조생혈도’와 ‘미백도’의 anthocyanin 합성 관련 유전자들의 염기서열을 분석해 보면 다형성이 없음을 확인할 수 있다(Kim et al., 2019). 238개의 유전자 중에서 카로티노이드 생합성에 관련된 유전자는 zeta-carotene desaturase로 (Shumskaya and Wurtzel, 2013) 6번 염색체에 위치하고 있고, 이 유전자로부터 유래한 SNP ID, ppa003478m:utr는 HRM 프라이머 위치를 찾을 수 없었다. 238개의 SNP 마커 중에서 과육 색에 따른 복숭아 품종을 황색과 백색 2군으로 구분할 수 있는 SNP 마커는 2종이었다(Table 4). SNP ID, ppa002847m:cds는 1번 염색체에 위치하고 있었고, plant neutral invertase family protein 또는 alkaline/neutral invertase A protein과 상동성을 갖는다. 이 유전자의 기능은 명확하게 밝혀지지 않았지만 주로 세포 소기관에 존재하는 것으로 알려져 있다(Murayama and Handa, 2007). SNP ID, ppa002540m:cds는 5번 염색체에 위치하고 있었고, AMP-dependent synthetase and ligase family protein과 상동성이 높으며 metabolism에 관여하는 것으로 알려져 있고, 식물 병원체의 황색 색소 형성과 연관이 있다고 보고되었다(Cao et al., 2018). SNP ID, ppa002847m:cds와 SNP ID, ppa002540m:cds 마커를 이용하여 HRM 분석한 결과, 황색 과육 복숭아 17 품종과 백색 과육 복숭아 21 품종을 구분할 수 있었고, 백색 과육 품종인 ‘미스홍’은 황육계 및 백육계 품종군에 들어가지 않고 또 다른 그룹으로 나뉘었다(Fig. 5).
Table 4.
PCR amplicons showing polymorphic HRM profiles in cultivars
| SNP ID | Var.y | Chr.x | TAIR10 hit | Forward primer | Reverse primer | Product |
| ppa002847m:cds | A:C/A | Chr1 |
plant neutral invertase family protein |
GAGAAGACGGTTG ACTGCCA |
GCCAATGGCTGAT TCACCAA | 138 |
| ppa002540m:cds | G:G/A | Chr5 |
AMP-dependent synthetase and ligase family protein |
TCCTGGAAACCCA ATGCTTG |
AAGCAACCTATGG CAAAGGA | 100 |
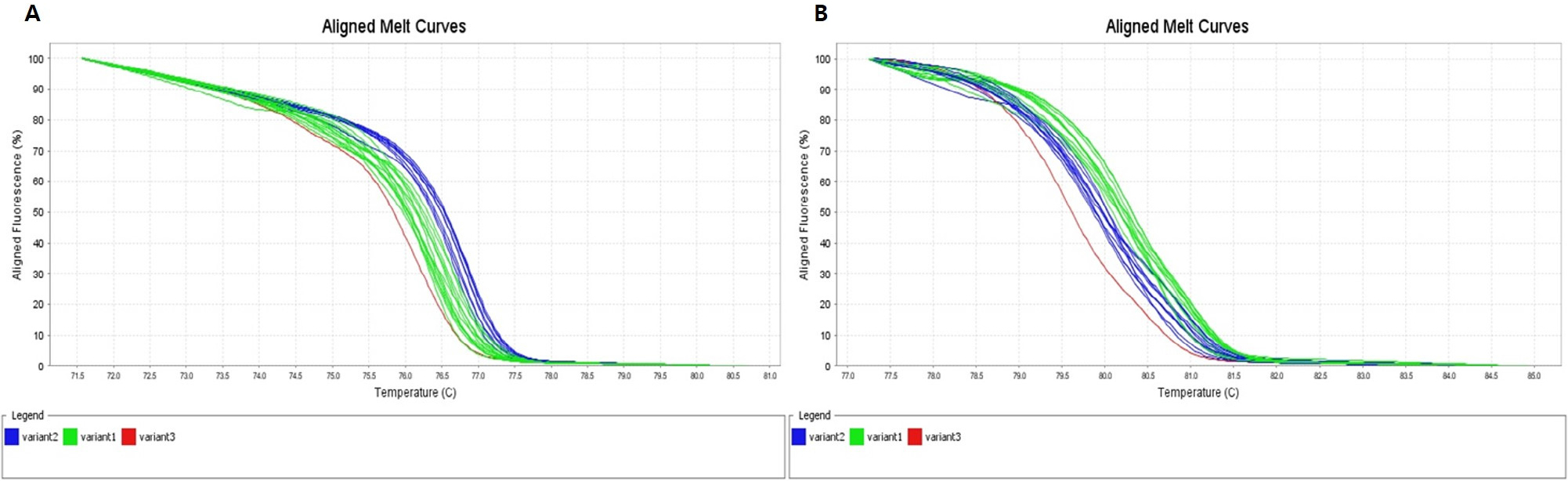
Fig. 5.
HRM analysis of 17 yellow-fleshed peach cultivars (Jinxiu, Chengyan, Takinosawa Gold, Sun Gold, Sunfre, Suhong, O’Henry, Changhowon Hwangdo, Zaoshengyoutaopantao, Juneprince, Chiyomaru, Cheonhong, Hahong, Fantasia, Hwanghoo, Hitachired) and 22 white-fleshed peach cultivars (Mibaekdo, Misshong, Mihong, Baekhyang, Bulgeonbaekdo, Ruipan3, Seonmee, Soomee, Spring Time, Tsukuba No.4, Seolhong, Innosense, Yumyeong, Yumi, Jinmi, Kawanakajima Hakutou, Tropic Snow, Weeping Peach OP4, Kanhito, 7-2-1-24, 7-2-1-50, 7-3-1-14). (A) Normalized graph of SNP ID, ppa002847m:cds. (B) Normalized graph of SNP ID, ppa002540m:cds. Blue variant2 indicate yellow-fleshed peach cultivars and green variant1 indicate white-fleshed peach cultivars, red variant3 indicate ‘Misshong’ in white-fleshed peach cultivars.
본 연구에서 개발한 2 종의 SNP 마커가 황육계 복숭아 품종과 백육계 복숭아 품종을 구분할 수 있는 정확한 기작에 대해서는 후속 연구가 필요하다. ‘미스홍’ 품종은 백색 과육 품종인데도 불구하고 또 다른 그룹으로 분류되었는데, 이러한 결과는 적육계와 백육계를 구분하는 마커 개발 결과(Kim et al., 2019)에서도 동일하게 나왔으며 이에 대한 연구도 필요하다. 고부가 기능성 복숭아 품종을 육성하는데 있어 전통육종방법은 유년기가 지나야지 과실의 특성을 볼 수 있기 때문에 상품성이 있으면서 황육색과 같은 특정형질만 발현시키는 데 시간과 노력이 필요하다. 본 연구에서는 고부가 기능성 품종으로 복숭아 황육계 계통을 선발하기 위해 RNA-Seq data 분석 방법으로 황육계와 백육계에서 발현량이 다른 유전자들을 분석하였고, HRM 방법을 이용한 SNP 마커를 개발하여 복숭아 유전자원 및 3개의 선발 계통 등 39 품종에 적용함으로써 황육색을 구분할 수 있는 마커의 가능성을 검토하였다. 본 연구에서 개발한 SNP 마커의 활용을 위해서는 다양한 복숭아 유전자원 및 황육계 품종과 백육계 품종 교배 집단에 적용해서 재현성을 확인해야 하며, 이러한 연구 결과는 황육계 복숭아 신품종의 육종 연한을 단축시키고 전통 육종의 효율성을 높일 수 있을 것이다.
적 요
복숭아 과육색은 상업적으로 중요한 분류 기준이며 영양 품질에 영향을 미친다. 카로티노이드가 다량 함유된 새로운 황색 과육 품종을 육성하기 위해서는 많은 교배 조합과 세대가 진전되어야 한다. 따라서 육종 효율을 높이기 위해서는 경제적으로 중요한 형질을 가진 교배 집단과 유전자원에 적용할 조기 선발 마커를 개발할 필요가 있다. 과육색이 다르게 발현되는 복숭아 품종의 유전자 발현을 비교하기 위해 2개의 cDNA library를 제작하였다. 황색 과육 품종인 ‘장호원황도’와 백색 과육 품종인 ‘미백도’의 유전자 발현 차이를 보기 위해 차세대 염기서열 분석 기술을 사용하였고 두 품종으로부터 얻은 EST의 염기서열을 결정하고 기존에 보고된 유전자와의 상동성을 분석하였다. EST 데이터로부터 황색 과육 품종 17개와 백색 과육 품종 22개를 구분할 수 있는 2종의 SNP 마커(SNP ID, ppa002847m:cds와 SNP ID, ppa002540m:cds)를 선발하였고, HRM 방법으로 분석하였다. 본 연구 결과는 복숭아 육종에 유용하게 사용할 수 있으며 복숭아 품종의 다양한 색 변화에 관한 분자 기작 연구에 좋은 참고자료가 될 수 있을 것이다.