서 언
재료 및 방법
야생 동충하초 채집
동충하초의 균주 분리 및 배양
Genomic DNA 분리
rDNA 단편의 PCR
rDNA 단편의 클로닝
염기서열분석
결 과
밀양지역의 자생동충하초의 기주곤충의 분류 계급별 채집 개황
ITS 영역의 PCR을 통한 서열분석
상동성 분석
유연관계 분석
고 찰
적 요
서 언
동충하초(冬蟲夏草)라는 이름은 겨울에는 곤충의 몸에 있다가 여름에는 풀처럼 돋아나는 모습에서 연유된 것으로, 동충하초균이 곤충의 몸에 침입하여 기주의 영양분을 이용하여 내생균핵을 만든 다음, 밖으로 자실체를 형성하는데, 이것이 바로 동충하초 버섯이다(Sung et al., 1993). 전술의 동충하초에 대한 기원은 학술적 측면의 정의이나 한국의 식품의약품안전처에서는 고시 제 2020-73호(2020.08.25)에 따라 동충하초를 생약명 ‘Cordyceps’로 칭하면서 “이 약은 동충하초균(冬蟲夏草菌) Cordyceps sinensis Sacc(맥각균과 Hypocreaceae)이 박쥐나방과(Hepialidae) 곤충의 유충에 기생하여 자란 자실체(子實體)와 유충의 몸체이다.”라고 정의하고 있다. 자연계에 자생하는 동충하초중 자실체 즉 버섯을 형성하는 대부분의 동충하초균은 자낭균강(Ascomycetes), 맥각균목(Clavicipitales), 맥각균과(Clavicipitaceae)의 동충하초속(Cordyceps)등 8개 속에 속하며, 한국을 비롯하여 중국, 일본 등 세계적으로 널리 알려져 있다. 하지만 현재 곤충을 비롯한 절지동물, 균류, 또는 고등식품의 종자 등에 기생하는 균류에 속하는 모든 균류도 넓은 의미에서 동충하초라고 하며, 이들 중에서 곤충을 숙주로 하는 동충하초균은 세계적으로 약 800여 종이 알려져 있고, 그중에서 자실체를 형성하는 것으로 알려진 것은 전술한 자낭균류의 동충하초속(Cordyceps)등 8개속에 속하는 균들로서 약 300 ~ 400여종이 보고되었고(Shimizu, 1997; Song et al., 1998; Sung et al., 1993; Sung et al., 1995), 또한 한국에서 동충하초로 채집, 분류된 것도 70여종에 이른다(Sung, 1996; Sung et al., 1997).
중국의 고서들에 의하면 동충하초는 암질환, 간질환, 폐질환, 뇌질환, 순환기 질환, 심장병, 당뇨병 등에 탁월한 효과를 나타낼 뿐만 아니라, 현대실용중약전에는 폐결핵, 노인쇠약성 만성결핵 빈혈, 허약체질 등을 치료한다고 나타나 있다. 최근에는 종양 억제율이 83%로 높은 항암효과 및 마약중독의 해독제로서도 효과가 있는 것으로 보고되어 있어(Bok et al., 1999; Kinjo et al., 1996; Song et al., 1998), 이에 대한 관심이 증가되고 있다(Furuya et al., 1983; Kiho et al., 1999). 특히 우리나라의 경우 Paecilomyces japonica/P. tenuipes(눈꽃동충하초), Cordyceps militaris(번데기 동충하초 또는 밀리타리스 동충하초)가 식품의 원료로, C. sinensis (동충하초)가 생약으로 허용되어 있어 수요가 점점 증가되고 있는 추세이다(Ban et al., 1998; Moon et al., 1997). 또한 백강균속의 균주들은 이미 진딧물류, 모기 등의 해충 방제에 활용하고 있고, 상기 언급한 동충하초의 주요 기주생물이 곤충이므로 이러한 점을 활용하여 해충방제용 생물농약으로서의 이용가치가 높고 산업자원으로서도 개발 가치가 높다고 할 수 있다(Cho et al., 2006; Lee et al., 2017; Rehner and Buckley, 2005; Scholte et al., 2004).
동충하초의 전통적 분류방법은 자실체, 포자 및 균사의 형태, 색 등의 유사성에 기초한 형태적 특징에 의존하여왔는데 환경적 요인과 생리적 조건변화로 인한 분류학상 문제점을 가지고 있다(Anderson et al, 1989). 반면, 분자생물학적 계통분류는 Random Amplified Polymorphic DNA (RAPD), Restriction Fragment Length Polymorphism (RFLP) 및 핵 ribosomal DNA로 크게 3가지로 나눌 수 있다. 그 중 rDNA에 의한 분류방법은 계통분류에 많이 응용되는데, 그것은 모든 생물체내에 고루 편재되어 있으며 coding 영역과 noncoding 영역으로 구분되어 종에 따라 수백에서 수천가지 보존적인 염기서열을 가지고 있으나 noncoding 영역은 종속 간에 다양성을 나타내기 때문이다(Toyama and Ogawa, 1974). 따라서 생명자원의 분류는 형태학적인 요인뿐만 아니라 유전적 요인을 바탕으로 분류하려는 노력들이 수행되어지고 있다(Hyun et al., 2020; Moon and Jang, 2020; Mwangi et al., 2019). 특히 Internal transcribed spacer (ITS)부위는 가운데의 5.8S를 포함하여 약 500 bp 정도로, 그 염기서열을 결정하기가 간편하고, 시간적인 제약을 덜 받기 때문에 다수의 균 종을 대상으로 하는 분류학적 연구에 적합하며, 그 분자 진화속도가 빨라 염기서열의 다양성을 요구하는 종, 속의 분류에 적합하다(Chen et al., 2001; Park et al., 2001). 더구나, 보존성이 높은 5.8S 부위를 포함하고 있으므로 ITS 부위와의 각각의 비교가 가능하며, 이러한 풍부한 정보량과 간편성으로 동일 속내의 종간 및 속간의 유연관계 연구에 유용한 수단으로 평가되고 있다(Chen et al., 2001; Song et al., 1998).
따라서 본 연구는 예로부터 약용생물이 많다는 의미로 전해지는 밀양의 재약산(약재가 많다는 의미)등 밀양근교의 일부 지역에 자생하는 야생 동충하초 균주를 수집, 분리하여 유전자원의 분류학적 정체성(identity)을 확보하고 ITS sequences에 의한 분류가 효과적일 수 있다는 일부 보고(Chen et al., 2001; Nam et al., 1999; Park et al., 2001)를 참조하여, ITS1, 5.8S rDNA 및 ITS2 부위의 염기서열을 비교함으로써(White et al., 1990) 염기서열 변이에 의한 속간, 종간, 종내의 계통적 유연관계를 비교하고, 한국의 미기록 동충하초종의 출현 여부를 확인하고, 그 특징을 살펴보기 위해 수행하였다.
재료 및 방법
야생 동충하초 채집
우리나라와 같이 4계절이 뚜렷한 온대 지방에서는 자실체의 형성 시기가 6월부터 9월까지로 이 시기가 채집하기에 적당한 시기다. 본 연구에서 야생 동충하초의 채집은 2013년 9월 중순부터 10월 중순 사이에 채집활동을 하였으며 그 결과 6속 32균주의 야생 동충하초를 채집할 수 있었다. 균종의 동정은 우선 선험적인 형태 형질을 이용하여 분류하고(Lee, 1990; Seok et al., 2013; Shimizu, 1997; Sung, 1996) 최종적으로 본 연구의 ITS 서열변이 검토 결과를 참조하여 분류하였다. 먼저 밀양시에 소재하는 재약산 일대에서 동충하초속(Cordyceps)의 노린재 동충하초(Cordyceps nutans), 번데기 동충하초(Cordyceps militaris)를 각각 1균주씩 채집하였고, 꽃동충하초속(Paecilomyces)의 fumosoroseus1균주와 백강균속(Beauveria) 1균주, 그리고 매미에 기생하는 나방꽃동충하초속(Isaria)의 takamizusanensis 1균주를 채집하였다. 또한 밀양시 가지산 일대에서 동충하초속(Cordyceps)의 벌동충하초(Cordyceps sphecocephala) 1균주, 꽃동충하초속(Paecilomyces)의 눈꽃동충하초 1균주와 나방꽃동충하초(Isaria) 1균주를 채집하였으며, 밀양시 청도면 조천계곡 주변에서는 모두 10균주를 채집하였는데 꽃동충하초속(Paecilomyces)의 눈꽃동충하초 6균주, 동충하초속(Cordyceps)의 1균주, Aranthomyces 속(한국 미기록종)의 나방흰가시동충하초 1균주와 백강균속(Beauveria)의 백강균 2균주를 채집하였다. 그리고 울주군 가지산 일대에서는 꽃동충하초속(Paecilomyces)의 눈꽃동충하초 2균주, 백강균속(Beauveria)의 백강균 5균주, Himenostilbe 속의 잠자리동충하초 1균주를 채집하였다. 밀양시 삼랑진읍 숭진리 일대에서는 모두 6균주를 채집하였는데 5균주는 백강균속(Beauveria)의 백강균이며, 1균주는 꽃동충하초속(Paecilomyces)이었다. 창녕군 화왕산 일대에서도 채집활동을 하였으나 단 한 균주도 채집하지 못하였다. 화왕산 일대의 계곡에 물이 많이 부족한 상태였고 주변의 습도도 매우 낮은 편이었다. 채집 장비로는 작은 모종삽과 핀셋, 손전등 그리고 채집한 동충하초를 각각 한 개체씩 담을 수 있는 샘플 통을 여러 개 준비하였다. 마르기 쉬운 동충하초는 샘플 통에 이끼 또는 풀잎을 함께 넣어 수분을 유지 시켜 주었다.
동충하초의 균주 분리 및 배양
채집한 동충하초는 핀셋 등을 이용하여 불순물을 깨끗이 제거한 다음 세균의 오염을 방지하기 위하여 자실체는 sodium hypocholride에 1 ~ 2분간 침지 살균한 후 멸균수로 세척하고 여과지에서 물기를 제거한 다음 water agar가 들어있는 페트리디쉬(Petri-dish) 뚜껑의 내면에 멸균한 실험용 색 태이프로 고정시킨 후 뚜껑을 덮어 26℃ 항온기에 넣어 두고 뚜껑 하면의 water agar 배지 위에 동충하초 자실체에 존재하는 포자가 떨어지게 하였다. 배지 위에 포자가 떨어져 발아한 포자는 감자한천배지(PDA)로 옮겨 분리 배양하였다. 자실체를 형성하지 않는 동충하초는 곤충의 몸 위에 형성된 포자를 살균된 백금이를 이용하여 직접 떼어내어 PDA배에서 배양하였다.
Genomic DNA 분리
채집한 동충하초 32균주(Table 1)를 PDA배지에서 14일간 배양하여 균사 군락을 핀셋으로 수확한 후 DNA 분리에 이용하였다. Genomic DNA 분리를 위하여 Promega 사의 WizardⓇ Genomic DNA Purification Kit를 이용하여 제시된 방법대로 시행하였다.
Table 1.
The list of the Cordyceps strains (samples) collected from Miryang region in the study
rDNA 단편의 PCR
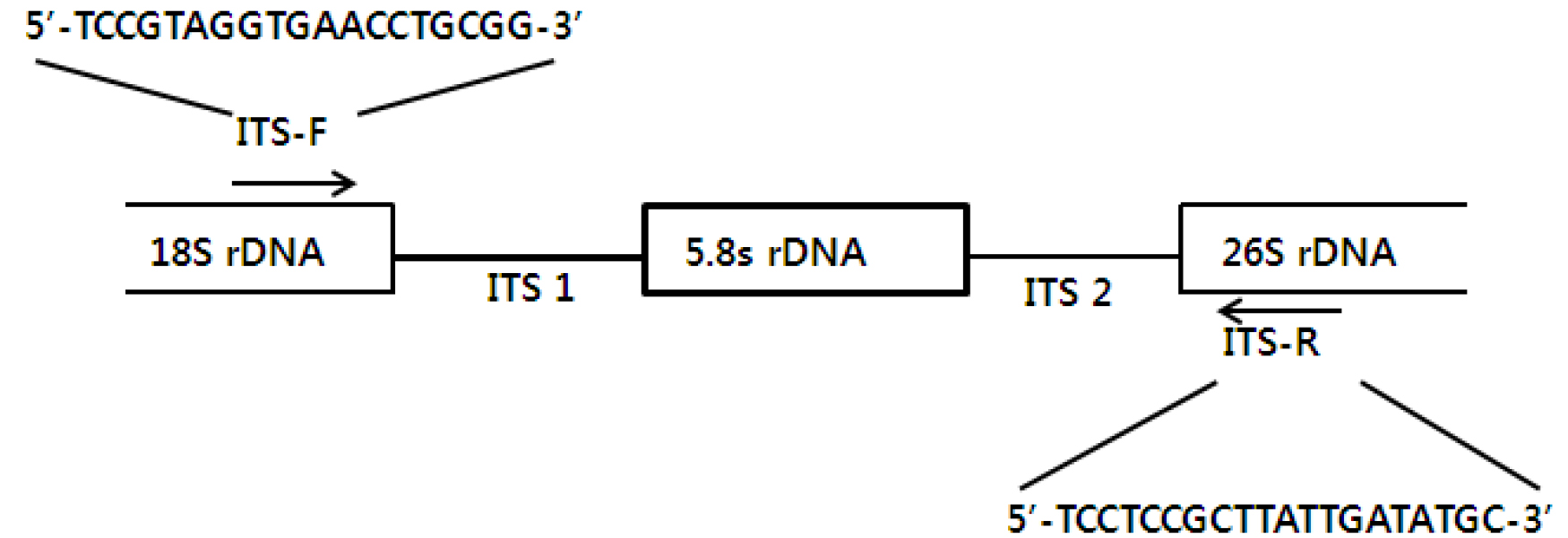
rDNA 단편의 증폭을 위하여 primer는 ITS-F (forward), 5'- TCCGTAGGTGAACCTGC-GG-3'와 ITS-R (revers), 5'- TCCTCCGCTTATTGATATGC-3'를 사용하였으며(Bachellerie and Qu, 1993) (Fig. 1), PCR 반응에 필요한 template DNA는 2 μL (100 ng/μL)를 사용하였고, primer는 ITS-F, ITS-R을 각각 2 μL (10 pmol)씩 취하여 14 μL의 D.W.에 미리 녹여둔 Premix (Bioneer Co, Korea)에 혼합하여 total volum 20 μL에서 증폭하였다. 반응조건은 1st step에서 initial denaturation으로 94℃ 5분간 1 cycle, 2nd step에서 denaturation 94℃ 1분간, annealing 55℃ 1분간, extension 72℃ 1분간의 반응을 순서대로 30 cycle 반복하였으며, 3rd step에서는 72℃ 10분간 extension을 추가하여 반응 완료 후 1% agarose gel에서 PCR 결과물을 확인하였다.
rDNA 단편의 클로닝
증폭한 rDNA 단편은 gel purification kit (Bioneer Co, Korea)를 이용하여 제시된 방법대로 회수하여 pGEM-T Easy vector system (Promega Co.)을 이용하여 클로닝하였다. 클로닝을 위하여 PCR 산물 25 ng, vector 50 ng, T4 DNA ligase 3 unit, 2 × ligase buffer 5 μL 그리고 멸균수를 혼합하여 total volum을 10 μL로 하여 천천히 혼합한 다음 5℃에서 overnight 시켰다. 재조합 DNA의 형질전환을 위하여 5 μL의 반응물과 50 μL의 competent cell (HITTM-DH5a 617, RBC Co.)을 혼합하여 얼음 위에서 20분간 반응한 뒤 42℃에서 1분간 열충격을 가하고 다시 얼음 위에 20분간 방치하였다. 이후 곧 ampicillin, X-gal, IPTG가 도말된 LB agar 배지에 도말하여 37℃에서 15 ~ 16시간 배양한 다음 Blue/White screening의 방법으로 white colony를 선발 배양하여 Plasmid Purification Kit (Bioneer Co, Korea)를 이용하여 plasmid DNA를 분리하여 1% agarose gel에서 확인하였다.
염기서열분석
분리 정제한 plasmid DNA는 ABI PRISM 3730XL analyzer (Applied Biosystems, USA)를 이용하여 염기서열을 결정하였으며, primer는 T7 primer와 SP6 primer를 이용하였다. Sequencing에 의해 밝혀진 염기서열은 종간의 상동성을 분석하기 위하여 Gene Bank에 이미 등록되어있는 다른 종의 염기서열과 함께 Clustal W program을 이용하여 multi-alignment한 후 Mega 프로그램을 이용하여 계통 관계를 분석하였다.
결 과
밀양지역의 자생동충하초의 기주곤충의 분류 계급별 채집 개황
본 연구를 위해 채집한 야생동충하초의 기주곤충별 채집개황을 조사한 결과(Table 1), 딱정벌레목(Coleoptera) 1 균주, 노린재목(Hemiptera) 1 균주, 나비목(Lepidoptera) 12 균주, 벌목(Hymenoptera) 1 균주, 거미목(Arachnida) 2 균주, 사마귀목(Mantodea) 6 균주, 잠자리목(Odonata) 1 균주, 파리목(Diptera) 모기과(Culicidae) 1 균주, 매미목(Homoptera) 2 균주, 무당벌레과(Coccinellidae) 1 균주, Cocoidea 1 균주와 Unknowm 3 균주로 나비목(Lepidoptera)이 가장 많았으며, 또한 기주 곤충의 분류계급을 무시한 발육 단계별로 균주를 분류해 보면 유충시기의 균주가 7 균주, 번데기 시기 4 균주, 성충시기 18 균주와 발육단계미상 3 균주로 확인되어 발육 시기 중에서 성충 시기의 균주가 조사 샘플 가운데 가장 많은 56%를 차지하였다.
ITS 영역의 PCR을 통한 서열분석
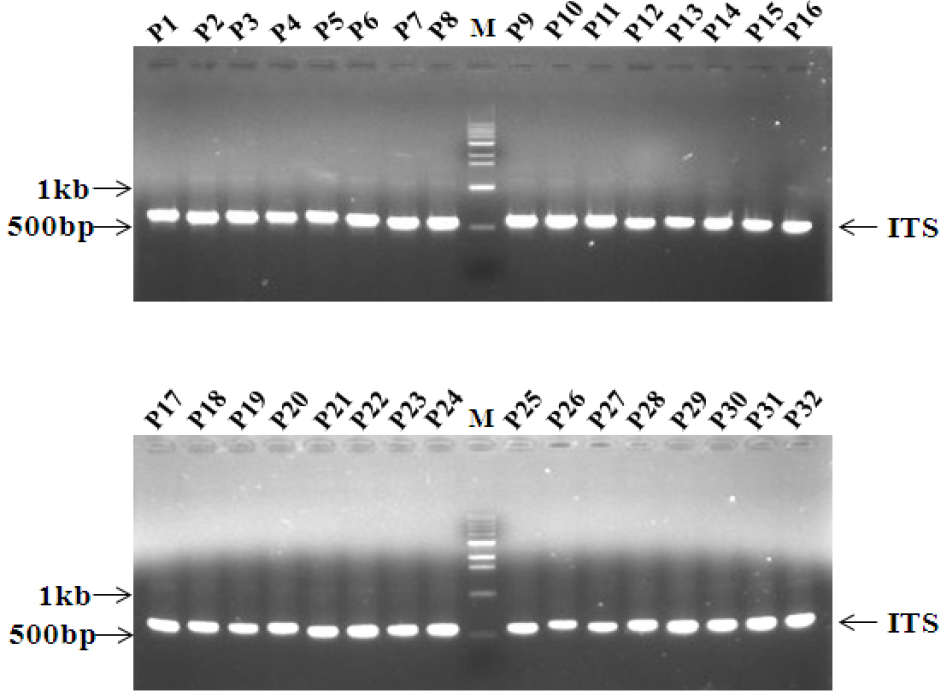
유연관계를 분석하기 위하여 ITS 영역을 증폭하였다. 아가로스겔(agarose gel)을 사용한 전기영동 결과 약 600 bp 크기의 단일밴드를 얻을 수 있었으며 32 균주별 PCR 산물의 크기는 육안으로 큰 차이를 인정할 수 없었다(Fig. 2). 32개 균주의 ITS영역별 크기를 분석한 결과(Table 2), ITS1의 경우, 백강균속(Beauveria)은 13개의 샘플 모두 162 bp로 그 크기가 같아 속 내의 샘플 간에 크기의 변이가 관찰되지 않았고, Cordycep속은 C. militaris - P2(번데기동충하초)는 161 bp, C. nutant - P1(노린재동충하초)는 148 bp, C. sphecocephala – P3(벌동충하초)는 195 bp, Cordyceps sp.- P14(사마귀동충하초)는 181 bp로 같은 동충하초 속(genus)이지만 속내의 종에 따라 ITS1영역에 있어서 크기의 차이가 최대 47 bp로 동일 속내의 종간에 영역의 크기가 심하였다. 꽃동충하초속(Paecilomyces)의 ITS1의 경우 동일 속내의 동일종간(P. tenuipes 종간)에는 183 bp로 크기가 동일하며, 종이 다를 경우(P. tenuipes 와 P. fumosoroseus 종간)에도 그 차이는 1 bp로 무시해도 좋을 정도이다. 상기 샘플 외의 Isaria takamizusanensis-P24 187 bp, Isaria sp.-P 156 bp, Aranthomyces aculeatus-P10 157 bp, Himenostibe odonatae- P18 195 bp 로 계통 간 최대 39 bp 의 크기 차이가 인정되었다. 그리고 5.8S 영역은 균주의 속이나 종에 관계없이 32균주 모두 157 bp로 본 연구에서 검정한 6개속에 속한 종간에 크기의 변이가 관찰되지 않았다. 그리고 ITS2 영역의 경우, 백강균속(Beauveria)은 13개의 샘플 모두 ITS1의 162 bp 보다 1 bp 적은 161 bp로 ITS1과 마찬가지로 동일속의 종간(샘플간) 크기의 차이가 관찰되지 않았다. 또한 동충하초속(Cordyceps)은 C. militaris – P2(번데기동충하초) 160 bp, Cordyceps sp - P14(사마귀동충하초) 161 bp, C. nutans(노린재동충하초) - P1 164 bp, C. sphecocephala - P3(벌동충하초)는 165 bp로 나타나 종에 따라서 최대 5 bp 크기의 근소한 차이가 있으나 그 길이가 종에 따라 다르게 나타나 변이가 비교적 강하게 일어남을 알 수 있었다. Paecilomyces 속은 P. tenuipes와는 종이 다른 P. fumosoroseus 2균주를 포함한 11개 샘플 모두 158 bp로 종간에 또는 동일종간의 채집 샘플별 ITS2 영역의 크기는 차이 없이 동일하였다. 또한, C. sphecocephala - P3(벌동충하초)과 H. odonata - P18(잠자리동충하초)은 속(genus)과 종(species)이 다름에도 불구하고 ITS1 영역의 길이가 각각 195 bp로 서로 같고, ITS2 영역도 165 bp로 상호 동일하였으며, ITS1과 ITS2 영역의 대부분의 염기서열도 일치하는 등의 결과는 매우 특이하고 재미있는 현상으로 보여 진다(Fig. 3).
Table 2
Sizes of ITS1, 5.8S, ITS2 regions of ribosomal DNA from isolates
상동성 분석
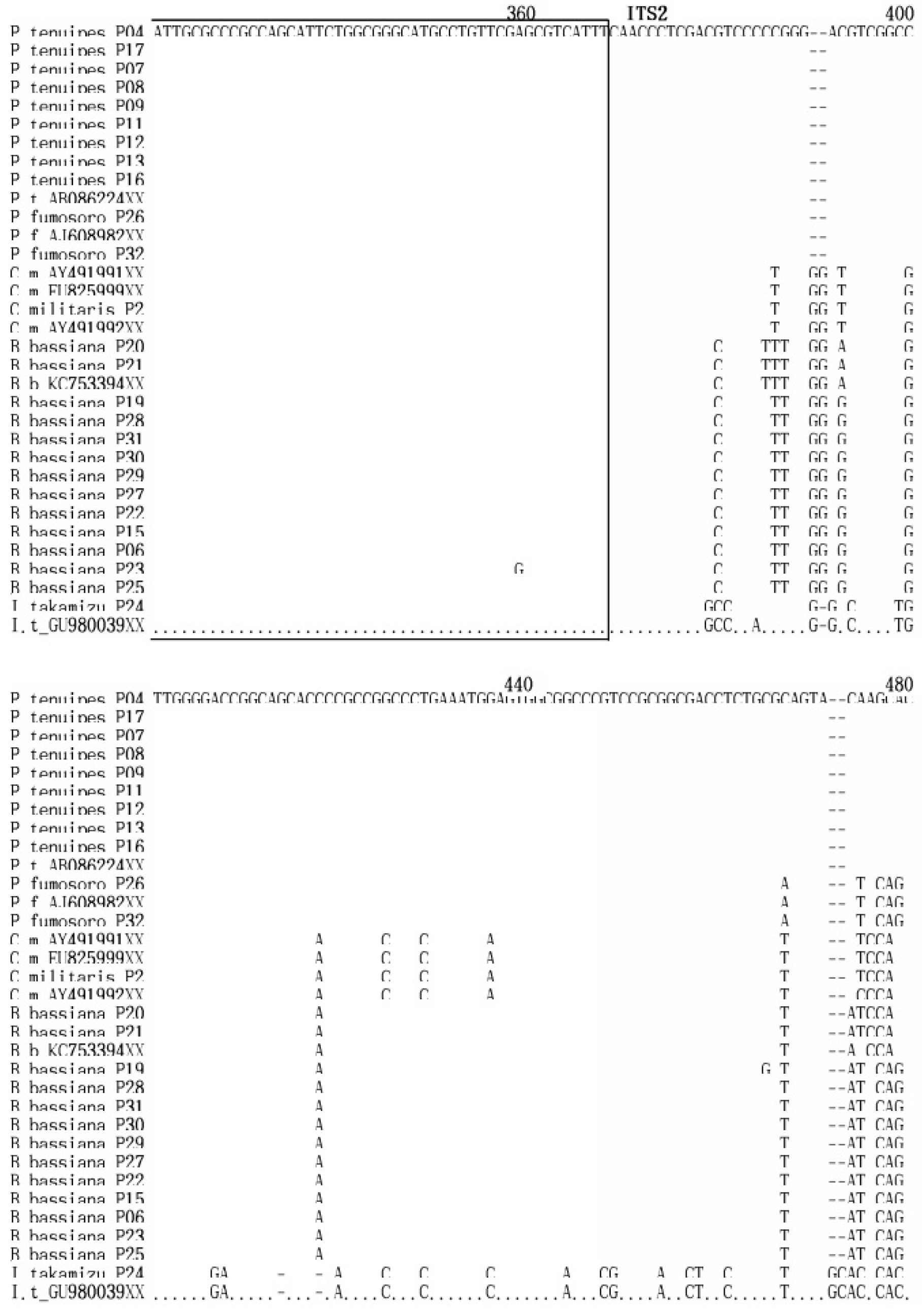
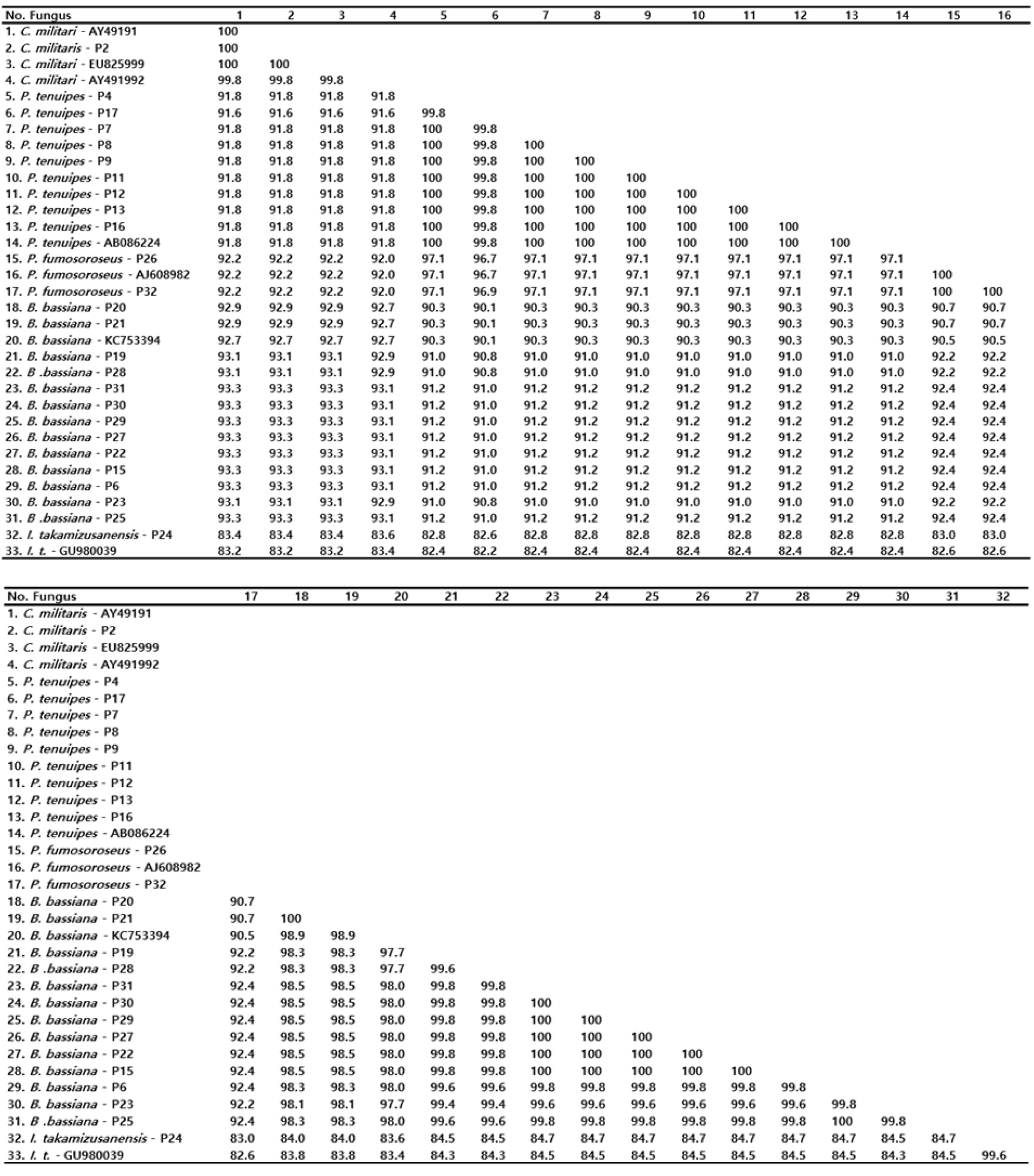
밀양시 인근 32개 자생 동충하초 샘플로부터 획득한 ITS1, ITS2 및 5.8S DNA 서열을 GenBank에 등록된 해당 균주의 염기서열과 상동성을 비교 분석 하였다(Fig. 4). 밀양의 재약산에서 채집한 Cordyceps militaris (P2)는 Gene Bank에 등록된 EU825999, AY49191과 100%의 상동성을 나타냈으며, AY49191와도 99.8%의 높은 상동성을 보였다.
Paecilomyces 속 9 균주들은 Gene Bank에 등록된 AB086224와 상동성을 비교한 결과 9개의 균주 즉, P4, P7, P8, P9, P11, P12, P13, P16 균주와 100%의 상동성을 보여 같은 종의 계통임이 확인되었고, P17 균주와도 99.8%의 상동성을 보여 지역에 따른 유전적 변이가 거의 일어나지 않았음을 알 수 있었다.
Beauveria 속의 13 균주는 Gene Bank에 등록된 KC753394와 상동성을 비교한 결과, P20, P21과 98.9%의 상동성을 보였으며, P6, P15, P22, P25, P27, P29, P30, P31과는 98%의 상동성을, P19와 P23, P28과는 97.7%의 상동성을 보였다. 울주군 가지산에서 채집한 P19, P20, P21, P22, P23은 같은 지역의 균주임에도 서로 다른 상동성을 보여 종의 분화가 일어나고 있음을 알 수 있었다. 그리고 P15, P22, P27, P29, P30, P31은 지역간 차이가 있음에도 상호 간에 100%의 상동성을 보여 분화가 일어나지 않은 동일종임을 확인할 수 있었다.
P. fumosoroseus 균주는 Gene Bank에 등록된 AJ608982의 염기서열과 상동성을 비교 분석한 결과 밀양 재약산의 P26, 밀양 삼랑진의 P31과 서로 간에 100%의 상동성을 보여 같은 종임을 확인하였고, 한국의 버섯목록에 기재되지 않은 미기록종으로(Seok et al., 2013) 사료된다(Fig.4).
I. takakumizusanensis 균주는 Gene Bank에 등록된 GU980039의 염기서열과 상동성을 비교 분석한 결과 99.6%의 높은 상동성을 나타냈고, 이 동충하초 역시 한국의 버섯목록에 기재되지 않은 미기록종으로(Seok et al., 2013) 사료된다(Fig. 4).
유연관계 분석
채집한 동충하초 균주의 계통유전학적 분석을 위하여 Gene Bank로부터 확보한 종을 포함하여 P. tenuipes 10 균주, P. fumosoroseus 3 균주, C. militaris 4 균주, B. bassiana 14 균주를 이용하여 이들의 종내 또는 종간의 관계를 분석하였다(Fig. 5). P. tenuipes 9 균주, P. fumosoroseus 2 균주, C. militaris 균주는 Gene Bank로부터 확보한 종과 종내 관계를 비교했을 때 같은 종으로 분류되었다. 그러나 B. bassiana 14 균주는 Gene Bank로부터 확보한 종과 종내 관계를 비교했을 때 울주군 가지산의 B. bassiana P20과 P21 상호 간에는 100%의 일치하는 균주이며, Gene Bank 등록 균주 KC753394와 98.9%의 유연관계가 있었으며, 다른 11종과도 97.7% 이상의 유연관계를 형성하고 있어 종의 계통분화가 미세하게 일어나고 있음을 알 수 있었다(Fig. 5).
고 찰
본 연구의 결과는 보존성이 높은 부분, 중간 정도의 부분, 그리고 변이가 심한 부분이 공존하고 있는 ITS1, 5.8S, ITS2 부위의 염기서열을 비교함으로써 속(genus) 및 종(species)간의 유연관계의 해석에 유용할 것으로 판단된다. 생물의 종의 분류를 위해서는 주로 형태적 특성을 이용하는 방법이 많이 사용되고 있으나 이 방법은 서언에서 기술한 바와 같이 환경적 요인 등에 의해 영향을 받는 경우가 많아 추가적인 분류방법이 모색되어져 왔는데, 이러한 대체분류방법(Alternative taxonomic character system)에는 isozymes (St. Leger et al., 1992), chemotaxonomic characters (Mugnai et al., 1989), mitochondrial RFLP (Hegedus and Khachatourians, 1993), immunological approaches (Shimizu and Aizawa, 1988), rRNA sequencing (Rakotonirainy et al., 1991), RFLP (Maurer et al., 1997), introns in the large subunit rDNA (Neuveglise et al., 1997), RFLP and nucleotide sequences of ITS (Coates et al., 2002), SSCP analysis of taxon specific markers (Hegedus and Khachatourians, 1996), RAPD markers (Cravanzola et al., 1997), 형태와 RAPD markers (Glare and Inwood, 1998) 등이 있으며 이들 대부분이 계통분류에 사용된다. 최근에는 ITS1,2 (the ribosomal internal transcribed spacer I and II) 서열을 이용한 종의 분류방법을 선호하는 편이다. rDNA의 coding sequences 들은 유전적으로 거리가 먼 종 사이에도 매우 잘 보존되어 있어 그 일부 서열이 universal primer 작성에 사용되며, 반면에 non-coding rDNA spacer sequence는 서로 유전적으로 가까운 종간에도 그 크기와 염기서열 구성에 변이가 심하여 생물종의 속간 또는 종간의 계통분류에 많이 이용된다(Hackett et al., 2000). 본 연구에서도 위와 같은 이유로 ITSI 및 II 서열을 중심하여 밀양지역에서 채집한 야생동충하초를 분자생물학적으로 분류하였다. 본 연구 결과에서 ITS1구간은 속간, 종간 염기서열 차이가 가장 뚜렷하였으며 특히 백강균속(B. bassiana)은 다른 속과의 염기서열 크기 및 구성에 있어 독특한 차이를 보인 것이 특징이며, 다른 개별 속(genus)의 종내의 계통간에는 종에 따라 차이가 있으나 그 차이는 매우 미미하였다(Table 2, Fig. 4, Fig. 5). 여기에서 백강균속(B. bassiana)의 ITS1 영역에서 다른 속 및 종과 염기서열 및 구성에서의 차이는 매우 흥미로운데 그 이유는 백강균이 곤충을 공격하여 기생하지만 자실체 즉 버섯을 형성하는 다른 동충하초 종과는 달리 곤충체에 자실체를 형성하지 않는다는 큰 생태적 생리적 차이점에 기인함이 크다고 사료된다. 5.8S rDNA 구간은 거의 모든 샘플 동충하초에서 동일한 염기서열을 보였으나 B. bassiana 계통들은 이 구간에서 크기는 157 bp로 다른 속 또는 종과 같았지만, 염기서열의 구성에 있어서는 다소 특이한 차이를 보였다(Table 2, Fig. 3). 이 같은 현상은 rRNA coding region인 5.8S DNA 영역의 서열은 진화과정에서 잘 보존된 서열이기 때문으로 생각된다. ITS2 구간에서도 ITS1 구간과 마찬가지로 염기서열 차이를 많이 보였으나 ITS1부위보다는 변이율이 높지 않았다.
또한, 채집한 동충하초의 larvae, pupae, adult별 감염된 균주 간에는 대체로 특별한 염기서열 차이가 나타나지 않았으며, B. bassiana는 대부분 성충을, P. tenuipes는 대부분 유충을 숙주로 삼아 발육단계 특이적 감염 특이성을 보이나 명확한 결론을 내리기는 어렵다. 본 연구결과에서는 나타내지 않았으나 야생 상태의 성충감염, 번데기감염, 유충감염상태의 동충하초균을 누에를 대상으로 실험한 결과 발육단계 특이적 감염 현상은 관찰되지 않았던 실험적 경험이 있었기 때문이다. B. bassiana-P25는 유충에, P. tenuipes-P4는 번데기를 숙주로 하였으나 ITS영역 염기서열을 비교하였더니 차이가 없었다. 즉, 발육단계 특이적 감염문제를 ITS 영역의 서열과 상관시키는 것은 현재로선 부적절하다고 생각하며 다만 이 문제에 대해서는 추가적인 학술적 연구가 수행되면 흥미로운 결과가 도출되리라 사료된다. 채집한 32 균주 샘플드에 대한 종간의 유연관계 분석을 위하여 ITS 영역을 클로닝하여 염기서열을 비교 분석한 결과 채집 야생 동충하초 계통 P. tenuipes 9 균주(-P4, -P7,-P8, -P9, -P11, -P12, -P13,- P16, -p17 샘플)는 Gene Bank의 P. tenuipes-AB086224와 채집된 야생 동충하초 계통 P. fumosoroseus 2 균주(P. fumosoroseus-P26, -32)는 Gene Bank의 P. fumosoroseus-AJ608982와 염기서열이 일치하였고, 채집된 야생 동충하초 계통 C. militaris-P2는 균주는 Gene Bank의 C. militaris-AY491991, -AY491992 및 -EU825999의 염기서열과 일치하여 각각 분자생물학적으로 동일종으로 분류되었다(Fig. 5). 그러나 B. bassiana 13 균주는 Gene Bank로부터 확보한 종과 종내 관계를 비교한 결과, 울주군 가지산의 B. bassiana P20과 P21은 상호 간에는 100% 일치하는 균주였고, Gene Bank 등록 균주 KC753394와는 98.9%의 유연관계가 있었으며 다른 11종과도 97.7% 이상의 유연관계를 형성하고 있어 미세한 종의 분화가 일어났음을 추론할 수 있다(Fig. 4, Fig. 5).
채집 야생 동충하초 계통 P. fumosoroseus 균주는 Gene Bank에 등록된 AJ608982의 염기서열과 상동성을 비교 분석한 결과, 밀양 재약산의 P26, 밀양 삼랑진의 P31과 상호 간에 100%의 상동성을 보여 같은 종임을 확인하였고 이 계통은 한국의 버섯목록에 기재되지 않은 미기록종으로 추후 연구가 기대된다(Fig. 4, Fig. 5). 또한, 채집 야생 동충하초 계통 I. takakumizusanensis 균주도 Gene Bank에 등록된 GU980039의 염기서열과 상동성을 비교 분석한 결과, 99.6%의 높은 상동성을 나타냈고, 이 동충하초 역시 한국의 버섯목록에 기재되지 않은 미기록종으로(Seok et al., 2013) 사료된다(Fig. 4, Fig. 5). 이 2 계통의 미기록(등록) 균주에 대해서는 추후 상세한 연구가 진행되리라 기대한다. 이상의 결과에서 무수한 종류의 야생 동충하초의 분자생물학적 분류에 rDNA의 ITS영역을 이용하는 방법이 상당히 유효할 것으로 생각되며, 또한 미분류 또는 미기록종을 찾는 데에도 상당히 기여할 것으로 사료된다.
따라서, 야생동충하초의 정확한 분류 및 동정을 위해 전통적인 형태적 분류법과 분자생물학적 기법을 병행하는 것이 이상적이라 사료되며, 특히 약리효과 등의 시험에 사용되는 균주에 대해서는 학술적으로 확실히 계통분류가 이루어진 공인 균주를 이용하여 생화학적 연구나 산업적 연구가 이루어져야 할 것으로 본다. 또한 ITS 서열을 이용한 동충하초의 종 분류에 있어 보다 많은 야생계통 또는 종을 공시하여 분자생물학적 유연관계를 조사하면 이들 균주의 진화생물학적 발전에 크게 기여할 수 있을 것으로 보이며 또한 실험실에서 새롭게 계량된 변이주를 구분하는 연구의 기본 자료로 이용될 것으로 생각된다.
적 요
남부 지방의 밀양 근교에 자생하고 있는 야생 동충하초 균주 (Cordyceps sp., Paecilomyces sp., Beauveria sp., Aranthomyces sp., Isaria sp., Himenostilbe sp.)를 채집 분리하여 rDNA 및 internal transcribed spacer (ITS) 부위의 염기서열을 비교하였다. ITS 영역에 특정적인 프라이머인 ITS1과 ITS4를 이용하여 PCR을 수행하여 증폭하였다. 다양한 균주에서 같은 크기의 PCR 생산물을 얻을 수 있었고, 이들의 서열분석을 위하여 pGEM-T easy 벡터에 클로닝하였으며, ITS1, 5.8S, ITS2 영역 부위의 염기서열들을 BLAST 수행하여 유연관계를 분석하였다. 밀양 근교에서 분리한 32개의 균주 중에 Cordyceps militaris는 서열이 등록된 유전정보 AY49191, EU825999, AY491992와 100% 일치하였으며, 몇 개의 종은 보고된 서열과 모두 일치하지는 않았다. 예를 들어 strain P17은 울주군 가지산에서 분리한 P. tenuipes로서 밀양시 조천읍 가지산에서 분리한 P. tenuipes 와는 서열상에서 다른 부분들이 있었다. 결론적으로 밀양 근교의 균주들을 분리하는데 ITS영역분석이 분류와 검정에 효율적이고, 본 연구를 통하여 동충하초의 생태학적인 유전자원들을 확보할 수 있었다.