서 언
재료 및 방법
독성 식물 시료 및 DNA 추출
PCR 증폭 및 염기서열 획득
염기서열 분석
결과 및 고찰
PCR 증폭 및 염기서열 획득 용이성
분자 유전학적 종 식별
7개 DNA 바코드의 단일 종 식별 유용성
적 요
서 언
식물의 절도 및 마약성 식물 소지 사건의 수사, 침입외래식물의 관리 등 다양한 분야에서 식물의 종 식별 및 개체 식별이 꾸준히 요구되고 있다(Chang et al., 2021; Hwang et al., 2013; Lee et al., 2020; Yoon et al., 2019). 특히 식물성 식품 원료의 진위 판별 및 국내산 품종판별 등 먹거리 안전과 관련된 식물의 종 식별 요구가 증가하고 있다(Chunet al., 2019; Ha et al., 2012). 식품의약품안전처의 통계에 따르면 2010년부터 2019년까지 10년간 자연 독으로 인해 발생한 식중독은 총 21건으로 135명의 환자가 발생하였으며, 2009년 한 해 급성 중독으로 전국 응급의료센터에 내원한 환자 3,500명 중 50명은 식물에 의한 중독환자로 나타났다(Sung et al., 2012).
식중독을 유발하는 미국자리공(Phytolacca americana), 박새(Veratrum oxysepalum) 등의 일부 독성 식물은 표현형이 산나물과 유사하여 이를 식용식물로 오인하여 섭취 후 병원에 내원한 사례가 다수 보고되었다(Bae et al., 2013; Jin et al., 2004; Hwang and Kim, 2015). 이외에도 다양한 독성 식물에 의한 식중독 사고가 꾸준히 발생하고 있으며, 이에 따라 독성 식물에 대한 정확한 종 식별이 요구되고 있다. 일반적으로 식물은 꽃과 뿌리, 잎의 형태 등을 이용한 형태학적 분류법을 통해 종 식별이 가능하지만 감정기관에 의뢰되는 식물은 옷이나 증거물에 묻은 식물 파편 혹은 죽은 사람의 위 내용물 등 다양한 형태로 의뢰되고 있기 때문에(Coyle, 2004; Lee et al., 2018), DNA 바코드를 이용한 분자 유전학적 종 식별이 필수적이다.
식물의 종 식별에 사용되는 DNA 바코드의 종류에는 trnH-psbA, trnL-trnF, trnL intron, psbK-psbI, rpoB, rbcL, matK 등 엽록체 DNA에 존재하는 구간을 대상으로 하는 바코드와, internal transcribed spacer (ITS), 18S rRNA 등 핵 DNA에 존재하는 구간을 대상으로 하는 바코드가 있다. 엽록체 DNA에 존재하는 구간을 대상으로 하는 DNA 바코드 중 trnH-psbA, trnL-trnF, 그리고 trnL intron 바코드는 염기서열의 변화정도가 낮은 tRNA 유전자를 포함하기 때문에 보존적인 영역을 기반으로 하는 primer의 제작이 가능하고, 돌연변이 발생 빈도가 높은 비암호화 영역을 대상으로 하기 때문에 식물의 종 식별에 많이 사용되는 DNA 바코드이다(Stech et al., 2003; Taberlet et al., 1991). rbcL과 matK 바코드 또한 보존적인 염기서열을 가지고 있어 식물의 계통발생학 연구 및 종 식별에서 널리 사용되는 DNA 바코드이다(Kress et al., 2005; Shaw et al., 2005). 핵에 존재하는 구간을 대상으로 하는 DNA 바코드 중 18S rRNA 바코드는 고등 생물의 계통 분석에 많이 사용되며(Guo et al., 2015; Jansen et al., 2006; Sӧrhannus, 2004), ITS 바코드는 보존적인 영역을 기반으로 하는 primer의 제작이 가능하고 증폭 및 근연종 간의 식별이 용이하여 동식물의 종 식별에 널리 이용되고 있다(Yao et al., 2010). 이와 더불어 Kress and Erickson (2007)이 수행한 연구에서 trnH-psbA, rbcL, matK, ITS1 바코드 등을 포함한 9개 DNA 바코드 각각의 종 식별력은 모두 79% 이하였으나 trnH-psbA 바코드와 rbcL 바코드를 조합하여 사용할 경우 종 식별력이 88%까지 증가함을 보고하였다(Kress and Erickson, 2007). 이처럼 식물의 종 식별에서는 단일 DNA 바코드를 사용하였을 때 종 식별력이 70% 이하인 경우가 흔하게 발생하기 때문에(Hollingsworth et al., 2011), DNA 바코드를 이용한 식물의 종 식별력을 높이기 위한 방법으로 두 가지 이상의 DNA 바코드를 조합하여 사용하는 multi-locus 방법이 지향되고 있다(Fazekas et al., 2008). 2009년 CBOL (China Plant BOL) Plant Working Group의 연구에서는 rbcL과 matK 바코드를 조합한 DNA 바코드가 범용성 핵심 바코드(core DNA barcode)로 제시되었으며, 이와 더불어 ITS 바코드를 종자식물의 핵심 바코드로 추가하여야 한다는 내용이 보고된 바 있다(CBOL Plant Working Group, 2009; China Plant BOL Group et al., 2011). 이러한 핵심 바코드 조합 외에도 다양한 식물 종에서 각각의 종 식별에 용이한 DNA 바코드 조합이 제시되었으며, 감정 기관에 종 식별이 의뢰되는 경우 신속하고 정확한 식별이 요구되기 때문에 각각의 식물에서 종 식별에 가장 적합한 DNA 바코드 조합을 찾아 데이터베이스를 구축하는 것이 필요하다.
본 연구는 국내에 자생하는 독성 식물을 수집하여 각각의 식물에서 증폭 및 염기서열 분석에 유리한 DNA 바코드를 파악하고자 하였으며, DNA 바코드 구간의 염기서열을 확보하여 추후 요구되는 독성 식물의 종 식별 감정에서 활용할 수 있는 데이터베이스를 구축하고자 하였다. 우선적으로 제주도에 자생하는 19종의 독성 식물을 수집하여 trnH-psbA, trnL-trnF, trnL intron, rbcL, matK, ITS1-ITS4, 18S rRNA 바코드에 대한 염기서열 데이터베이스를 구축하였으며, 각각의 종 식별에 가장 적합한 DNA 바코드를 확인하여 독성 식물의 감정에 신속하고 정확하게 적용할 수 있는 DNA 바코드 데이터베이스를 구축하였다.
재료 및 방법
독성 식물 시료 및 DNA 추출
본 연구에 사용된 식물 시료는 국내에 분포한다고 알려진 330여종의 독성 식물 중 제주도에 분포하고 있는 19종을 선정하여 농업회사법인 제주생물자원(주)으로부터 표본을 구입하였다(Fig. 1). 19종의 독성 식물은 2013년부터 2020년까지 제주도 지역에서 채취된 식물로, 농업회사법인 제주생물자원(주)에서 형태학적 분류법에 따라 종 식별이 수행되었으며 각각의 식물 별 독성 성분 정보는 North Carolina Extension Gardener Plant Toolbox (https://plants.ces.ncsu.edu)와 국립수목원에서 발간된 독성 식물 필드 가이드북을 참고하여 정리하였다(Korea National Arboretum, 2020; 2021) (Table 1). 실험에 사용된 독성 식물의 genomic DNA는 시료의 잎을 약 500 ㎎ 채취하여 AUTOMILL 파쇄기(Tokken, Japan)를 이용해 1600 r/min으로 3분 동안 파쇄한 후, DNeasy Plant Mini Kit (QIAGEN, Germany)를 이용하여 사용자 매뉴얼에 따라 추출하였다. 추출된 DNA는 0.8% 아가로즈 젤 전기영동 후 Image Analyzer (UVP)로 확인하였으며, Nanodrop spectrophotometer (Thermo Fisher Scientific, USA)를 이용하여 농도를 측정하였다.

Fig. 1.
Nineteen poisonous plants used in this study. (A) Nerium oleander, (B) Arisaema amurense, (C) Arisaema ringens, (D) Pinellia ternata, (E) Xanthium strumarium, (F) Impatiens textori, (G) Euonymus alatus, (H) Euphorbia esula, (I) Euphorbia helioscopia, (J) Euphorbia jolkinii, (K) Ricinus communis, (L) Canavalia lineata, (M) Veratrum nigrum, (N) Cocculus trilobus, (O) Phytolacca americana, (P) Pulsatilla cernua, (Q) Solanum lyratum, (R) Solanum americanum, (S) Laportea bulbifera.
Table 1.
Morphological identification and information of poisonous plants used in this study
PCR 증폭 및 염기서열 획득
엽록체에 위치하는 trnH-psbA, trnL-trnF, trnL intron, rbcL, matK 바코드와 핵에 위치하는 ITS1-ITS4, 18S rRNA 바코드의 primer를 사용하여 PCR 증폭하였다(Table 2). 증폭 조성은 정방향(forward), 역방향(reverse) primer 각각 10 pmole, Ampli Taq Gold DNA polymerase 2.5 unit (Thermo Fisher Scientific), Gold ST*R 10X buffer 2.5 μL (Promega, USA), 주형 DNA 약 30 ng을 넣어준 후, 멸균 증류수를 total volume 25 μL가 되게 채웠다. 증폭 장비는 ABI 9700 system (Applied BioSystems, USA)을 사용하였으며, 95℃, 11 분 pre-denaturation, 95℃, 30 초 denaturation 후, primer 별 Tm에 따라 50℃-58℃, 30 초 annealing, 72℃ extension 1 분을 28 cycle로 반복하였고 72℃에서 7 분 동안 final extension을 수행하였다. 증폭 산물은 2% 아가로즈 젤 전기영동 후 Image Analyzer (UVP)로 증폭여부를 확인하였다. 염기서열 분석을 위해 증폭된 PCR 산물을 ExoSAP-IT (Thermo Fisher Scientific)로 정제하였다. 이 후, ABI PRISM BigDye Terminator v3.1 Cycle Sequencing Kit (Applied BioSystems)를 이용하여 direct sequencing을 수행한 다음, DyeEX™ 2.0 Spin Kit (QIAGEN)를 이용하여 반응에 사용되지 않은 잔여 형광물질을 제거하였다. 최종적으로 정제된 증폭 산물은 3500xl Genetic analyzer (Applied BioSystems)를 사용하여 사용자 매뉴얼에 따라 염기서열을 분석하였다.
Table 2.
Information of DNA barcode primers used in this study
| Locus | Primer name | Primer sequence (5'-3') | Tm (℃) | Reference |
| Chloroplast | trnH-psbA | F : GTTATGCATGAACGTAATGCTC | 56 | Sang et al., 1997; Tate and Simpson 2003 |
| R : CGCGCATGGTGGATTCACAATCC | ||||
| trnL-trnF | F : AAAATCGTGAAGGTTCAAGTC | 53 | Sang et al., 1997; Taberlet et al., 1991 | |
| R : GATTTGAACTGGTGACACGAG | ||||
| trnL intron | F : CGAAATCGGTAGACGCTACG | 58 | Taberlet et al., 1991 | |
| R : GGGGATAGAGGGACTTGAAC | ||||
| rbcL | F : AGTTCCGTCAGAAGAAGCAGGAG | 50 | Ng et al., 2016 | |
| R : CATTCAAGTAATGCCCTTTGATTTC | ||||
| matK | F : CGATCTATTCATTCAATATTTC | 50 | Cuénoud et al., 2002 | |
| R : TCTAGCACACGAAAGTCGAAGT | ||||
| Nuclear | 18SrRNA | F : TGCAGTTAAAAAGCTCGTAG | 53 | Rollo et al., 2002; Soltis et al., 1997 |
| R : GGTTGAGACTAGGACGGTATCTG | ||||
| ITS1-ITS4 | ITS1 : TCCGTAGGTGAACCTGCGG | 54 | White et al., 1990 | |
| ITS4 : TCCTCCGCTTATTGATATGC |
염기서열 분석
Sequencing으로 얻어진 염기서열은 MEGA software (Molecular Evolution Genetics Analysis 5.2 software, USA)로 정렬 및 비교하였고, NCBI (National Center for Biological Information, USA) GenBank의 BLAST (Basic Local Alignment Search Tool)를 이용하여 가장 유사한 종을 검색하였다. 계통수 분석은 MEGA software를 이용하여 BLAST 결과 염기서열 일치율이 가장 높았던 3종의 염기서열을 NCBI에서 내려 받아 alignment한 후, Neighbor joining 방법으로 bootstrap replication (1000)을 적용하여 수행하였다.
결과 및 고찰
PCR 증폭 및 염기서열 획득 용이성
분자 유전학적 종 식별을 위해 19종의 독성 식물에서 7개의 DNA 바코드를 PCR 후 염기서열을 분석하였다(Fig. 2). DNA 바코드별로, trnH-psbA 바코드는 19종 중 15종, trnL-trnF 바코드는 19종 중 19종, trnL intron 바코드는 19종 중 17종, rbcL 바코드는 19종 중 18종, matK 바코드는 19종 중 17종, 18S rRNA 바코드는 19종 중 19종, ITS1-ITS4 바코드는 19종 중 19종에서 각각 증폭되어 염기서열을 분석할 수 있었다. 독성 식물 19종 모두에서 증폭되며 염기서열 획득에 가장 용이한 DNA 바코드는 trnL-trnF, 18S rRNA, 그리고 ITS1-ITS4 바코드였다.
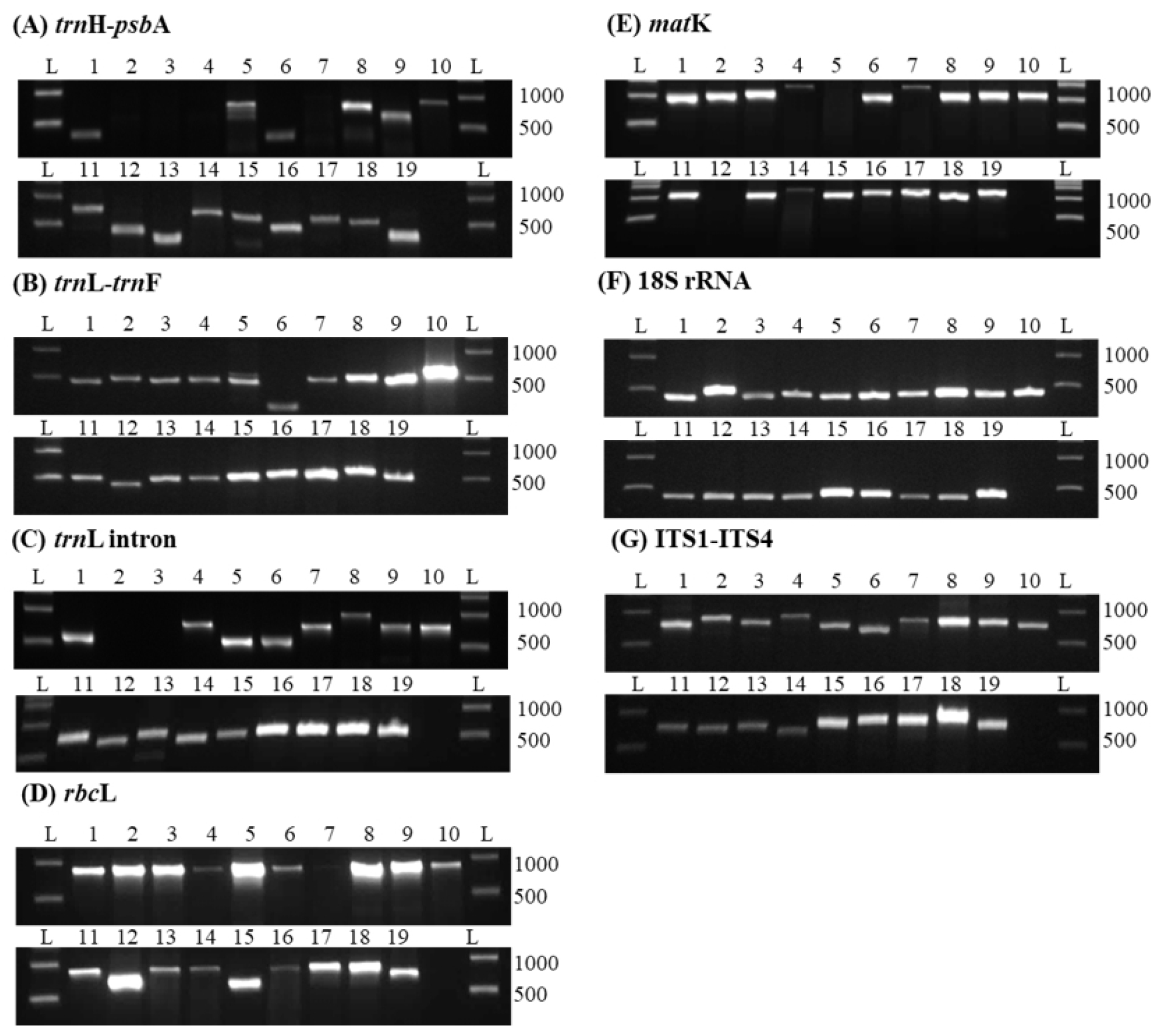
Fig. 2.
PCR amplification of seven DNA barcodes. (A) trnH-psbA, (B) trnL-trnF, (C) trnL intron, (D) rbcL, (E) matK, (F) 18S rRNA, (G) ITS1-ITS4. 1-19 represents NFS No. of each plant indicated in Table 1. L: ladder marker (1 kb).
본 연구에서 trnH-psbA 바코드의 경우 천남성과(Araceae)에 속하는 독성 식물 3종(NFS_TP02, NFS_TP03, NFS_TP04)과 노박덩굴과(Celastraceae)에 속하는 Euonymus alatus (NFS_TP07)에서 증폭되지 않았는데, 이러한 결과는 Liu et al. (2021)의 종 식별 연구에서 trnH-psbA 바코드의 경우 한약재 천남성(Arisaematis Rhizoma)으로 사용되는 천남성과의 식물에서 PCR 증폭이 되지 않는 결과와 동일하였다(Liu et al., 2021). 이는 천남성과에 속하는 식물들의 trnH-psbA 바코드 구간은 아데닌(A)과 티민(T) 염기의 비율이 평균 70%로 높아 sanger sequencing을 이용한 염기서열 분석에 어려움이 있다는 연구에서 그 원인을 유추할 수 있다(Luo et al., 2009). 따라서 천남성과에 속하는 독성 식물의 종 식별에는 trnH-psbA 바코드를 제외한 다른 DNA 바코드를 이용하는 것이 적합할 것으로 사료된다. 이 외에도 trnL intron 바코드는 천남성속(Arisaema)에 속하는 독성 식물 2종(NFS_TP02, NFS_TP03)에서, rbcL 바코드는 E. alatus (NFS_TP07)에서, matK 바코드는 Xanthium strumarium (NFS_TP05)과 Canavalia lineata (NFS_TP12)에서 각각 증폭되지 않아 해당 독성 식물의 종 식별을 위해서는 다른 DNA 바코드를 이용하는 것이 적합할 것으로 사료된다.
분자 유전학적 종 식별
각각의 독성 식물에서 획득한 DNA 바코드 구간의 염기서열을 이용하여 BLAST를 통한 상동성 비교 검색을 수행하였다(Table 3). trnH-psbA 바코드는 염기서열을 분석한 15종 중 12종(80%)의 염기서열이 NCBI GenBank에 등록된 염기서열과 매칭되었으며, trnL-trnF 바코드는 19종 중 15종(78.95%), trnL intron 바코드는 17종 중 14종(82.35%), rbcL 바코드는 18종 중 16종(88.89%), matK 바코드는 17종 중 16종(94.12%), 18S rRNA 바코드는 19종 중 6종(31.58%), ITS1-ITS4 바코드는 19종 중 19종(100%)의 염기서열이 매칭되었다. 종 별로 NCBI GenBank에 해당하는 DNA 바코드 구간의 염기서열이 등록되어있지 않아 대조할 수 없는 경우 본 연구에서 분석한 염기서열 정보를 NCBI GenBank에 등록하였으며, 해당 정보를 Table 3에 표기하였다.
Table 3.
Molecular genetic identification using 7 DNA barcodes in poisonous plants
분자 유전학적 종 식별 결과 19종 중 17종이 형태학적 분류법을 통한 식별 결과와 일치하는 종으로 식별되었으나 NFS_TP13과 NFS_TP18 표본은 형태학적 종 식별 결과와 다른 식물로 식별되었다. NFS_TP13 표본은 형태학적 분류법에서 Veratrum oxysepalum으로 식별되었으나 BLAST를 통한 상동성 비교 검색 결과 Veratrum nigrum과 매칭되었으며, NFS_TP13 샘플과 NCBI GenBank에 등록된 V. oxysepalum의 바코드별 염기서열 일치율은 trnH-psbA 바코드 94.92%, trnL-trnF 바코드 94.67%, trnL intron 바코드 91.62%, rbcL 바코드 99.12%, matK 바코드 97.71%, 18S rRNA 바코드 100%, ITS1-ITS4 바코드 93.52%로 18S rRNA를 제외한 6개의 바코드에서 V. nigrum 보다 낮은 일치율을 보였다. V. oxysepalum과 V. nigrum은 잎의 모양이 매우 유사하여 꽃의 색깔이 형태학적 종 식별에 중요한 역할을 하며, 두 종의 개화 시기는 6~8월로 알려져 있다. 본 연구에 사용된 식물 시료는 3월에 채집된 식물의 표본으로 채집 시기에 개화가 되지 않아 형태학적 종 식별에 혼선이 있었을 것으로 사료된다.
NFS_TP18 표본은 형태학적 분류법에서 Solanum nigrum으로 식별되었으나 BLAST를 통한 상동성 비교 검색결과 Solanum americanum과 매칭되었다. S. americanum과 S. nigrum은 화서의 종류와 꽃의 색깔 등 형태학적인 차이가 있어 두 종을 이종으로 보기도 하지만, 데이터베이스에 따라 S. americanum Mill.을 정명으로 하고, S. nigrum L.을 이명으로 처리하여 동종이명으로 보기도 한다(The Plant List ver. 1.1, 2013, http://www.theplantlist.org). NFS_TP18 표본과 NCBI GenBank에 등록된 S. nigrum의 바코드별 염기서열 일치율은 trnH-psbA 바코드 99.81%, trnL-trnF 바코드 97.97%, trnL intron 바코드 99.58%, rbcL 바코드 100%, matK 바코드 99.71%, 18S rRNA 바코드 99.22%, ITS1-ITS4 바코드 100%로, trnH-psbA와 rbcL 바코드를 제외한 DNA 바코드에서 S. americanum 보다 낮은 일치율을 보였으나 trnL-trnF 바코드를 제외한 모든 DNA 바코드의 염기서열 일치율이 99% 이상으로, 정확한 종 식별을 위해 추가적인 분석이 필요하다고 판단되었다. 본 연구에서 획득한 NFS_TP18 표본의 바코드 구간 염기서열을 NCBI GenBank에 등록된 S. nigrum과 S. americanum의 염기서열과 alignment하여 분석한 결과 trnL-trnF 바코드 구간에서 S. nigrum과 S. americanum 사이에 1개의 SNP와 삽입/결실이 존재했으며, NFS_TP18 표본의 염기서열은 S. americanum과 일치하였다. 또한 trnL-trnF 바코드 구간에서 ‘TCATGTT’ motif가 S. nigrum의 경우 2번 반복하며 존재하는 반면, S. americanum은 1번 존재하였으며 NFS_TP18 샘플 역시 1번 존재하였다(data not shown). 따라서 NFS_TP18 표본은 최종적으로 S. americanum으로 판단되었다.
7개 DNA 바코드의 단일 종 식별 유용성
BLAST를 통한 상동성 비교 검색에서 trnH-psbA 바코드를 사용하여 종을 식별할 경우 염기서열이 매칭된 12종 중 8종(66.67%)이 같은 속 내의 근연종과 2% 이상의 염기서열 일치율 차이를 보이며 단일 종으로 식별되었으며, trnL-trnF 바코드는 15종 중 10종(66.67%), trnL intron 바코드는 14종 중 5종(35.71%), rbcL 바코드는 16종 중 6종(37.50%), matK 바코드는 16종 중 7종(43.75%), 18S rRNA 바코드는 6종 중 4종(66.67%), ITS1-ITS4 바코드는 19종 중 11종(57.89%)이 단일 종으로 식별되었다. 단일 종으로 식별되지 않은 종은 본 연구에서 획득한 염기서열과 NCBI GenBank에 등록된 해당 종 및 BLAST 결과 염기서열 일치율이 가장 높았던 3종의 염기서열을 alignment한 후, Phylogenetic tree를 작성하여 계통수 분석을 수행하였다(Fig. 3). 계통수 분석을 추가적으로 수행한 결과, trnH-psbA 바코드를 사용하여 종을 식별할 경우 12종 중 8종(75%)이 다른 종들과 구분되며 단일 종으로 식별되었으며, trnL-trnF 바코드는 15종 중 12종(80%), trnL intron 바코드는 14종 중 8종(57.14%), rbcL 바코드는 16종 중 10종(62.50%), matK 바코드는 16종 중 12종(75%), ITS1-ITS4 바코드는 19종 중 16종(84.21%)이 단일 종으로 식별되었다.
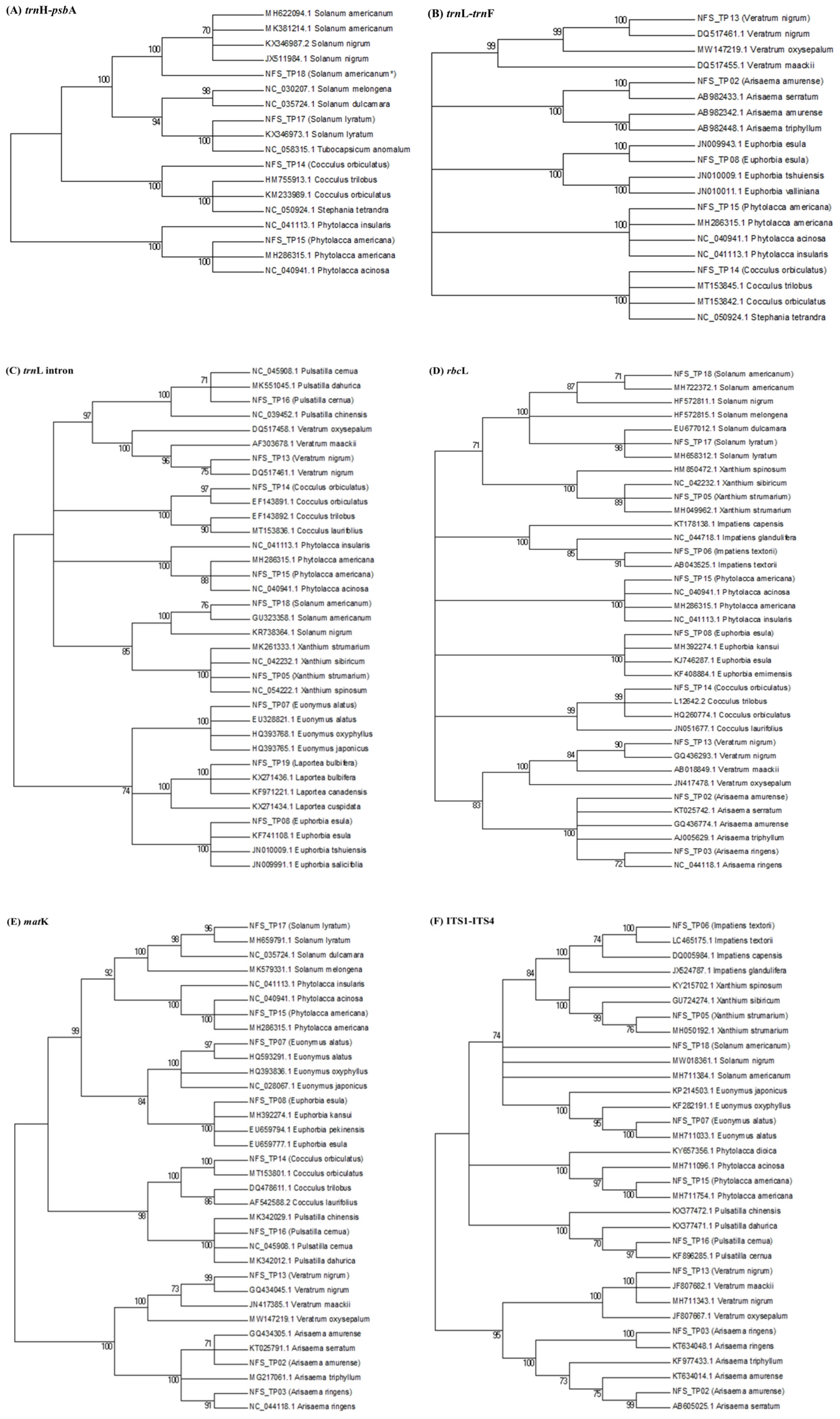
Fig. 3.
Neighbor Joining (NJ) phylogenetic tree. Phylogenetic trees by NJ statistics with 1,000 bootstrap replicated based on (A) trnH-psbA, (B) trnL-trnF, (C) trnL intron, (D) rbcL, (E) matK, (F) ITS1-ITS4. Phylogenetic trees were constructed with species that were not identified as a single species in the BLAST results. The numbers shown on the branch nodes indicate bootstrap values (> 50%).
단일 종 식별률이 가장 높았던 DNA 바코드의 조합은 trnL-trnF 바코드와 ITS1-ITS4 바코드 조합으로, 두 개의 DNA 바코드를 조합하여 종 식별을 수행하면 BLAST 상동성 비교만으로 19종 중 16종(84.21%)의 식물에서 단일 종 식별이 가능하였으며, phylogenetic tree를 이용한 계통수 분석을 추가적으로 수행하면 19종 중 18종(94.74%)의 식물에서 단일 종 식별이 가능하였다. 따라서 미지의 독성 식물에 대한 감정이 의뢰되면 우선적으로 trnL-trnF 바코드와 ITS1-ITS4 바코드를 조합하여 사용하는 것이 신속한 종 식별에 도움이 될 것으로 사료된다. 18S rRNA 바코드는 PCR 증폭 및 염기서열 획득에는 용이하였으나 본 연구에서 종 식별을 수행한 독성 식물 19종 중 13종의 염기서열이 NCBI GenBank에 등록되어있지 않아 염기서열 대조에 어려움이 있었으며, 계통수 분석을 수행할 수 없었다. 따라서 18S rRNA 바코드를 국내에 자생하는 독성 식물의 분자 유전학적 종 식별에 활용하기 위해서는 NCBI에 독성 식물들의 염기서열 정보 등록이 선행되어야 할 것으로 사료된다.
국립과학수사연구원은 국내에 자생하는 독성 식물의 신속하고 정확한 종 식별을 위해 DNA 바코드 데이터베이스를 구축하는 것을 목표로 하고 있으며, 본 연구는 기초 연구로서 제주도에 자생하는 독성 식물 19종을 선정하여 7개의 DNA 바코드를 이용한 종 식별을 수행하였다. DNA 바코드의 효율을 평가할 때, PCR 증폭이 용이하며, 수동적인 염기서열의 교정 작업이 없어도 고품질 양방향 염기서열을 획득할 수 있으며, 근연종 간의 높은 식별력을 가진 DNA 바코드가 이상적인 DNA 바코드로 판단된다(CBOL Plant Working Group, 2009). 이러한 기준에 따라 19종의 독성식물 각각에서 DNA 바코드 별 1. PCR 증폭이 용이, 2. 고품질 염기서열 획득 용이, 3. BLAST 상동성 비교 검색만으로 단일 종 식별 가능, 위 세 가지 조건의 충족 여부를 확인하였으며, 최종적으로 각각의 종 식별에 용이한 DNA 바코드의 데이터베이스를 구축하였다(Table 4). 본 연구를 통해 확보된 DNA 바코드 데이터베이스를 활용할 경우, 다양한 독성 식물의 종 식별 감정에 빠르게 대응할 수 있을 것으로 기대되며, 이와 더불어 더욱 정확하고 신속한 종 식별 감정을 위해 국내에 자생하는 독성 식물을 대상으로 추가적인 DNA 바코드 데이터베이스 확보가 필요할 것으로 사료된다.
Table 4.
Recommended DNA barcodes for species identification of 19 poisonous plants in this study
적 요
독성 식물로 인한 식중독 사고는 매년 발생하고 있으며, 일부 독성 식물은 식용 식물로 오인 섭취되어 식중독을 유발하기 때문에 독성 식물의 정확한 종 식별이 요구되고 있다. 이러한 요구에 따라 감정기관에서는 독성 식물의 종 식별에 적합한 DNA 바코드를 찾아 신속하고 정확한 종 식별에 활용할 수 있는 데이터베이스를 구축하는 것이 필요한 실정이다. 따라서 본 연구는 다양한 독성 식물에서 DNA 바코드 구간의 염기서열을 확보하고, 각각에 적합한 DNA 바코드를 확인하여 데이터베이스를 구축하고자 하였으며, 기초 연구로써 제주도에 자생하는 독성식물 19종을 선정하여 7개의 DNA 바코드 (trnH-psbA, trnL-trnF, trnL intron, rbcL, matK, ITS1-ITS4, 18S rRNA)를 이용한 종 식별을 수행하였다. 종 식별 결과 trnL-trnF 바코드와 ITS1-ITS4 바코드가 PCR 증폭 및 염기서열 획득에 가장 용이하였으며, 두 개의 바코드를 조합하여 사용하면 19종 중 18종의 식물에서 단일 종 식별이 가능하였다. 따라서 미지의 독성 식물에 대한 감정이 의뢰되었을 때 trnL-trnF 바코드와 ITS1-ITS4 바코드를 조합하여 사용하면 신속한 종 식별에 도움이 될 것으로 사료된다. 본 연구에서 제시된 독성식물 19종의 염기서열 및 DNA 바코드 데이터베이스는 더욱 신속하고 정확한 독성 식물의 종 식별 감정에 도움이 될 것이다.




