서 언
재료 및 방법
식물 재료 및 특성 조사
통계분석
Genomic DNA 추출 및 마커 적용
염기서열 분석
결 과
멜론 유전자원의 생육 특성 조사
멜론 유전자원의 PCA 분석
과육색 구별 분자 표지 적용과 염기서열 분석
고 찰
적 요
서 언
멜론(Cucumis melo L.)은 풍부한 비타민과 섬유질을 제공하는 중요한 과일로 국내 재배 면적은 약 1,500 ㏊이며, 생산량은 약 39,800 톤이다(KOSIS, 2019). 소비 형태는 다양하여 국내에서는 녹색 과실을 가지는 얼스 타입이 선호되나 일본, 미국, 유럽의 경우는 오렌지색 과육을 갖는 캔탈롭 형태도 재배 및 소비되고 있다(Torres et al., 2020).
멜론은 이배체 종으로(2n=24) 약 450Mb로 추정되는 유전체를 가지며(Arumuganathan and Earle, 1991), 유전체 및 전사체 정보가 공개되어 있으며(https://cuccap.org), 과실의 과육발달에 관한 모델식물로 주목을 받고 있다(Garcia-Mas et al., 2012). 멜론의 과육색은 카로티노이드와 엽록소 색소의 함량에 의해 결정되며, 크게 흰색, 녹색 그리고 오렌지색으로 구분된다. 이 중, 오렌지색은 카로티노이드 성분 중에서 베타 카로틴(β-carotene)에 의한 것으로 알려져 있다(Burger et al., 2009).
식물의 생장과 더불어 사람에게도 중요한 영양성분으로 잘 알려진 카로티노이드는 광합성을 하는 모든 식물에서 생산되며 몇몇 박테리아와 곰팡이에서도 생합성되며, 식물의 경우는 과실의 향기 성분을 만드는 식물 호르몬인 ABA의 전구물질이기도 하다(Walter and Strack, 2011). 또한, 카로티노이드는 항산화 물질로 식물이 빛 또는 UV 등에 대한 환경스트레스에 견디도록 할뿐만 아니라(Niyogi, 1999) 암조건에서 발아하는 경우 황색체(etioplast)가 적절한 기능을 수행하는데 있어 카로티노이드의 생합성과 축적은 중요하다고 알려져 있다(Rodriguez- Concepcion, 2010). 멜론의 경우 생합성 관련 유전자가 유전체에 26개 존재한다고 보고되었다(Garcia-Mas et al., 2012). 멜론의 과육색 결정에 대한 정확한 기작은 아직까지 알려져 있지 않으나 3개의 유전자가 중요한 역할을 한다고 알려져 있다. 한가지는 녹색(green flesh)을 결정하는 gf (green flesh) 유전자와 흰색 과육(white flesh)를 결정하는 wf (white flesh) 유전자이다. 오렌지색 과육은 gf 유전자의 우성인 Gf 또는 CmOr 유전자에 의해 결정되며, gf 유전자를 가지는 경우는 멜론의 과육이 녹색 또는 흰색 과육의 표현형을 나타낸다고 하였다(Clayberg, 1992; Tzuri et al., 2015). CmOr 유전자는 브로콜리 BoOr의 homolog 유전자로써 클로닝되었으며, 멜론 유전체의 9번 염색체에 위치하는 것으로 밝혀졌다(Tzuri et al., 2015). Or 유전자는 DnaJ Cys-rich zinc finger를 갖고 있는 단백질로 식물 종간에 아미노산 염기서열이 매우 잘 보존되어져 있으며, 식물에만 특이적으로 존재하는 유전자로 알려졌다(Kim et al., 2018). 멜론 외에도 애기장대(Yuan et al., 2015; Zhou et al., 2015), 고구마(Goo et al., 2015; Kim et al., 2013), 수수(Yuan et al., 2015), 콜리플라워(Lu et al., 2006) 등에서도 Or 유전자의 homolog가 존재한다고 보고되었으며, 그 기능은 식물 종에 따라 약간의 차이가 있다고 보고되었다. 애기장대(Arabidopsis thaliana; AtOR)에서는 카로티노이드 축적과 phytoene synthase (PSY) 유전자 조절 기능을 가지며(Yuan et al., 2015; Zhou et al., 2015), 수수(Sorghum bicolor; SbOR)와 애기장대 캘러스를 이용한 실험에서는 카로티노이드 축적(Yuan et al., 2015), 고구마[Ipomoea batatas (L.) Lam; IbOr]에서는 뿌리 조직에서 카로티노이드 축적(Park et al., 2015), 캘러스를 이용한 실험에서 카로티노이드 축적과 염류 스트레스 증가(Kim et al., 2013), abiotic 스트레스에 대한 tolerance 증가(Goo et al., 2015), 광합성 조절(Kang et al., 2017), PSY 단백질 안정화(Park et al., 2016) 등에 관한 기능이 보고되었다. 멜론의 경우는 베타카로틴의 축적과 스트레스 환경하에서 함량을 일정하게 유지하는 항상성에 관여하고 있으며(Kim et al., 2018), 오렌지와 녹색 과육의 멜론을 대상으로 염기서열을 분석한 결과, 총 6개의 단일염기다형성(single nucleotide polymorphism; SNP)이 CmOr 유전자에 존재함이 보고되었다(Tzuri et al., 2015). 이중 1개의 SNP가 G에서 A로 바뀌면 이에 해당하는 아미노산 서열이 arginine (Arg)에서 histidine (His)으로 바뀌고, 이 SNP는 멜론의 오렌지색과 녹색/흰색 과육색 결정을 하는 것으로 보고하였다. 이러한 결과는 Chayut et al. (2015)도 bulk segregant transcriptome 방법으로 이 유전자의 발현량을 조사한 결과 멜론 녹색과 오렌지색 과육 개체 사이에서 발현량의 차이가 없었다고 보고한 바 있다.
본 연구는 멜론 육종과 연구를 위한 기본적인 정보를 제공하기 위해 농업유전자원센터에서 수집한 자원들을 대상으로 생육특성을 조사하였으며, 원예적으로 중요한 형질인 과육색을 예측하는 마커의 정확도를 평가하였다.
재료 및 방법
식물 재료 및 특성 조사
본 연구에 사용된 총 219개 멜론 유전자원은 농촌진흥청 국립농업과학원 농업유전자원센터에서 수집 및 보관 중인 종자를 분양 받았다(Table 1). 이 자원들은 경기도 이천에 위치한 동오시드 R&D 센터의 비닐 하우스 시설에서 재배되었다. 2020년 4월 7일 파종하여, 자원당 8~12개씩 주간 간격은 40 ㎝, 열간 간격은 20 ㎝으로 5월 14일 정식하였다. 유전자원의 형질조사는 농업유전자원센터에서 제시한 기준에 따라 수행되었다. 조사 항목은 꽃의 성표현(sex expression; SE), 절간 길이(internode length; IL), 잎 크기(leaf size; LS), 잎 가장자리의 톱날모양(leaf teeth of the margin, LTM), 과형(fruit shape; FS), 과실골 유무와 형태(Fruit cork formation and pattern; FCFP), 과피색(fruit skin color; 과실골 유무 및 정도 (creasing of fruit surface; CFS), 과중(fruit weight; FW), 과장(fruit length; FL), 과폭(fruit width; FW), 태좌부위 넓이(placental width; PW), 과실당도(sugar content; SC), 과육색(fruit flesh color; FFC), 과일향 유무(fruit flavor; FF), 흰가루병 저항성(resistance to powdery mildew; RPM), 종피색(seed coat color; SCC), 백립중(one hundred seed weight; OHSW) 등을 포함한 총 19개에 대해 수행하였다(Supplementary data 2). 그리고, 흰가루병 저항성을 판단하기 위해 Edisto 47, Iran H, MR-1, PMR 5, PMR 6, PMR 45, PI 414723, PI124112, Top Mark 의 총9개 계통(cultigen)을 동일한 하우스 내에 재배하였으며, 분리형질이 나타난 경우는 농업유전자원센터의 유전자원의 기초특성 조사 기준에 따라 우점개체의 생육특성을 기록하였다.
Table 1.
List of melon (Cucumis melon L.) accessions used in this study
| Accession no. | Sourcez | Accession no. | Source | Accession no. | Source | Accession no. | Source |
| 173184 | RUS | K018969 | IND | K189241 | IND | K189644 | AFG |
| 190341 | UZB | K018974 | JPN | K189253 | IND | K189652 | AFG |
| 190454 | UZB | K037412 | CHN | K189258 | IND | K189665 | AFG |
| 199217 | RUS | K051416 | CHN | K189265 | IND | K189672 | AFG |
| 805439 | KAZ | K060638 | Unknown | K189274 | IND | K189693 | AFG |
| 805452 | TKM | K126991 | MMR | K189287 | IND | K189702 | AFG |
| 805454 | UZB | K133663 | UZB | K189288 | IND | K189704 | AFG |
| 805462 | IRN | K138764 | Unknown | K189291 | IND | K189719 | AFG |
| 805480 | UZB | K141247 | IND | K189303 | IND | K189722 | IND |
| 805483 | KAZ | K145159 | HUN | K189337 | IND | K189750 | CAN |
| 805484 | TKM | K145164 | BGR | K189338 | IND | K189760 | CAN |
| 805487 | UZB | K145166 | HUN | K189346 | IND | K189761 | CAN |
| 805488 | UZB | K145170 | Unknown | K189348 | IND | K189764 | CAN |
| 805491 | UZB | K145172 | HUN | K189353 | IND | K189765 | CAN |
| 805496 | UZB | K145173 | HUN | K189364 | IND | K189767 | IRN |
| 805499 | UZB | K146753 | BGR | K189368 | IND | K189778 | IRN |
| 805501 | TKM | K146812 | BGR | K189375 | IND | K189780 | IRN |
| 805502 | UZB | K146932 | BGR | K189376 | IND | K189787 | IRN |
| 805505 | UZB | K151072 | UKR | K189377 | IND | K189789 | IRN |
| 805511 | Unknown | K151075 | UKR | K189378 | IND | K189792 | IRN |
| 805513 | Unknown | K151093 | UKR | K189379 | IND | K189793 | IRN |
| 805516 | Unknown | K152719 | ECU | K189382 | IND | K189795 | IRN |
| 805517 | Unknown | K163682 | KOR | K189384 | IND | K189797 | IRN |
| 805520 | Unknown | K174975 | USA | K189396 | IND | K189801 | IRN |
| 805522 | Unknown | K175016 | USA | K189410 | IND | K189802 | IRN |
| 805523 | Unknown | K189080 | IND | K189442 | IND | K189816 | IND |
| 805524 | Unknown | K189096 | IND | K189456 | IND | K189818 | PAK |
| 805526 | Unknown | K189097 | IND | K189460 | IND | K189820 | USA |
| 805528 | TKM | K189109 | FRA | K189462 | IND | K189824 | IND |
| 805532 | UZB | K189111 | FRA | K189463 | IND | K189830 | IND |
| 807688 | UZB | K189112 | FRA | K189471 | IND | K189834 | IND |
| 808289 | THA | K189117 | CHN | K189488 | USA | K189839 | IND |
| 903200 | AFG | K189121 | ALB | K189491 | USA | K189840 | IND |
| 903211 | UZB | K189122 | ALB | K189492 | CHN | K189841 | IND |
| 903225 | UZB | K189123 | ALB | K189498 | IND | K189842 | IND |
| 906951 | KOR | K189125 | FRA | K189501 | IND | K189843 | IND |
| 906954 | Unknown | K189127 | FRA | K189509 | PAK | K189848 | IND |
| 906961 | JPN | K189128 | FRA | K189516 | IND | K189849 | IND |
| 906963 | Unknown | K189148 | USA | K189519 | IND | K189853 | IND |
| 906964 | Unknown | K189163 | ESP | K189520 | IND | K189856 | IND |
| 906965 | Unknown | K189166 | ESP | K189535 | IND | K189857 | IND |
| 906966 | Unknown | K189173 | ESP | K189541 | IND | K189864 | IND |
| 906968 | Unknown | K189175 | ESP | K189546 | IND | K189865 | IND |
| 906975 | Unknown | K189179 | ESP | K189547 | IND | K189878 | TUR |
| 909847 | Unknown | K189184 | ESP | K189553 | IND | K189886 | IND |
| 909849 | Unknown | K189198 | ESP | K189554 | IND | K189900 | TUR |
| K002891 | RUS | K189199 | ESP | K189560 | IND | K189904 | TUR |
| K003003 | TJK | K189200 | ESP | K189561 | IND | K189912 | TUR |
| K014686 | UZB | K189205 | ESP | K189564 | IND | K189919 | TUR |
| K014694 | UZB | K189211 | ESP | K189581 | AFG | K189935 | TUR |
| K014700 | UZB | K189219 | ESP | K189585 | AFG | K189938 | TUR |
| K014703 | UZB | K189220 | ESP | K189599 | AFG | K189943 | TUR |
| K014705 | UZB | K189224 | KAZ | K189600 | AFG | K189949 | TUR |
| K018966 | IND | K189229 | IND | K189603 | AFG | K189951 | TUR |
| K018968 | IND | K189236 | IND | K189612 | AFG |
zAFG, Afghanistan; ALB, Albania; BGR, Bulgaria; CAN, Canada; CHN, China; ECU, Ecuador; ESP, Spain; FRA, France; HUN, Hungary; IND, India; IRN, Iran; JPN, Japan; KAZ, Kazakhstan; KOR, South Korea; MMR, Myanmar; PAK, Pakistan; RUS, Russian Federation; THA, Thailand; TJK, Tajikistan; TKM, Turkmenistan; TUR, Turkey; UKR, Ukraine; USA, United States of America; UZB, Uzbekistan.
통계분석
멜론 219개 자원의 형질을 이용한 주성분 분석 및 유의차 검정은 R 소프트웨어(http://www.r-project.org)에서 제공하는 prcomp 명령어를 이용하였으며, 각 자원에서 형질의 분리가 일어난 경우, 형질 값을 평균으로 대체하여 분석을 수행하였다.
Genomic DNA 추출 및 마커 적용
멜론 유전자원의 잎을 채취한 다음 CTAB 방법(Doyle and Dyole, 1987)을 이용하여 gDNA를 추출하였으며, 추출된 gDNA는 0.8% agarose gel에 전기영동하여 확인한 후, 5 ng/μL로 희석하여 PCR 반응에 사용하였다. PCR 용액은 dNTP (each 10 mM) 0.5 μL, Taq (5 U/ μL) 0.1 μL, 10Xbuffer 2 μL, 각 프라이머(5 μM) 0.5 μL, gDNA 5 μL (5 ng/ μL), 멸균수 10.9 μL 를 혼합하여 20 μL에 맞추었다. 유전자원에 적용한 프라이머는 OrF4 (5’-CTCCTTGGTTTTCTTCATG-3’)와 OrR15 (5’-CAACAAAA CCCATCAAGTC-3’)이다(Tzuri et al., 2015). PCR 반응 조건은 95℃에서 5분간 pre-denaturation, 94℃에서 30초간 denaturation, 58℃에서 30초간 annealing하여 72℃에서 40초간 extension 과정을 35회 반복하고, 72℃에서 10분간 최종 extension을 수행하였다. 증폭된 PCR 산물은 5 μL를 1.5% agarose에서 전기영동하여 확인한 후, HinfI 제한 효소(Enzynomics, Daejeon, Republic of Korea)를 처리하였다. 제한 효소 반응액은 각 PCR 산물에 9 μL에 전용 buffer 1 μL, HinfI 0.05 U를 37℃에서 2시간 처리한 후, 2% agarose에서 전기영동하였다.
염기서열 분석
PCR 산물은 정제 후 ABI PRISM 3730XL Analyzer (96 capillary type, Thermo Fisher Scientific, Ma, USA)를 이용하여 염기서열을 분석한 후, National Center for Biotechnology Information (https://www.ncbi.nlm.nih.gov)의 Nucleotide Basic Local Alignment Search Tool (BLASTN)를 이용하여 확인하였다.
결 과
멜론 유전자원의 생육 특성 조사
본 연구에 사용한 멜론 총 219개 유전자원은 총 24개 국가에서 수집되었으며, 인도에서 수집된 유전자원이 72개로 가장 많았으며, 그 다음으로는 우즈베키스탄 21개였다(Supplementary data 1). 형질 조사는 과실을 중심으로 수행하였으며, 중요한 형질 중 하나인 과형은 총 9개로 구분하여 조사하였다. 그 결과, 타원형(elliptical type)이 65개로 가장 많았으며, 도토리모양(acorn type) 이 7개로 가장 적었으며, 가리비모양(scallop type)은 없었다. 과실 과피색은 총 4개로 구분하였으며, 노란색(yellow) 과피색의 유전자원이 148개(68%)로 대부분을 차지하였으며, 흰색(white)이 10개, 녹회색(green-gray)의 유전자원이 11개였다. 과실 무게는 매우 다양하여 15 g에서 2,000 g이 넘는 유전자원까지 존재하였다. 멜론의 가장 중요한 원예 형질인 당도의 경우, 13 brix 이상의 고당도 유전자원은 5개(807688, 906954, 906965, 906975, K189600) 존재하였으나 모두 과실 1주당 평균 1.5개로 증식이 매우 어려운 자원이었다. 흰가루병 발생 정도도 조사되었으나 완전히 저항성인 유전자원은 없었으며, 43개 유전자원이 중도 저항성을 보였다(Supplementary data 2).
멜론 유전자원의 PCA 분석
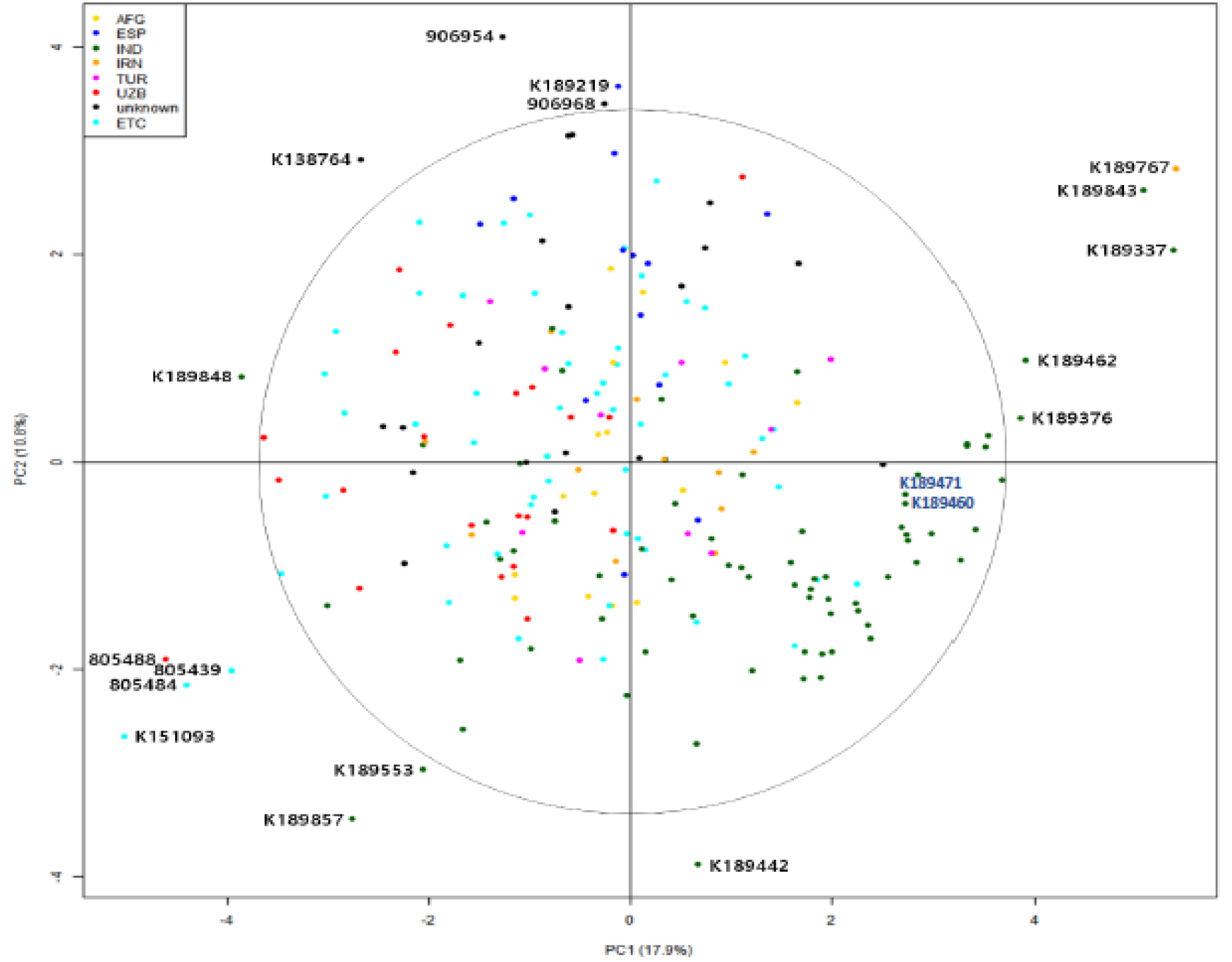
멜론 유전자원 219개의 19개 형질을 이용하여 주성분 분석을 수행한 결과를 Fig. 1 과 Table 2에 나타내었다. Fig. 1을 보면 아프카니스탄(노란색), 이란(주황색), 터키(분홍색) 유전자원은 전체적으로 고르게 분포하였으며, 인도 유전자원(녹색)은 오른쪽 하단에 주로 분포하였다. 그리고, 스페인 유전자원(파란색)은 상단 좌우에 주로 분포하였으며, 우즈베키스탄 유전자원(빨강)은 오른쪽 상하단에 주로 분포하는 것으로 나타났다. 또한, 3.7로 설정된 중심원을 벗어나는 유전자원은 총 17개로 (805439, 805484, 805488, 906968, 906954, K138764, K151093, K189219, K189337, K189376, K189442, K189462, K189553, K189767, K189843, K189848, K189857)였다. 이 중에서, 인도가 8개로 가장 많았으며, 스페인, 이란, 우크라이나, 우즈베키스탄, 카자흐스탄, 투르크메니스탄 등의 유전자원이 각 1개씩 있었으며, 원산지를 알 수 없는 유전자원이 3개 포함되어 있었다. 또한, 주성분 분석을 통하여 얻어진 각 특성들에 대한 고유값(eigenvalue)은 제 1주성분(PC1)의 경우, 1.846으로 전체 분산에 대하여 17.94%의 기여도가 있는 것으로 분석되었으며, 고유값이 1 이상인 제 2주성분(PC2), 제 3주성분(PC3)의 고유값은 각각 1.434와 1.288로 전체 분산에 대하여 각각 28.77%와 37.5%의 기여도를 나타내고 있다. 주성분 분석결과 고유값이 1 이상으로 전체 분산에 대하여 37.5%의 기여도를 나타낸 제 1주성분부터 제 3주성분까지의 특성간 상관관계를 분석한 결과, 제 1주성분은 -0.419(과폭) ~ 0.138(과형)로 상관계수가 분포하였으며, 과폭, 성표현, 잎크기, 과중, 태좌부넓이, 백립중에서 -0.3 이하로 높은 음의 상관관계를 나타내고 있다. 제 2주성분의 경우 -0.318(과형) ~ 0.444(당도)로 상관계수가 분포하고 있으며, 당도, 잎 가장자리 톱니모양, 과피색 등의 요인에 0.3 이상으로 비교적 높게 편중되어 있다. 제 3주성분에서는 -0.416(과형) ~ 0.366(과실 향기)로 상관계수가 분포하고 있으며, 과실향기에서 가장 높은 상관계수를 나타냈다(Fig. 1, Table 2).
Table 2.
Principal component analysis (PCA) of various morphological traits in melon accessions
| Principal Componentsz | PC1 | PC2 | PC3 |
| Eigen value | 1.846 | 1.434 | 1.288 |
| Variance (%) | 17.94 | 10.83 | 8.73 |
| Cumulative (%) | 17.94 | 28.77 | 37.5 |
| SE | -0.340 | 0.292 | -0.032 |
| IL | -0.178 | -0.077 | -0.219 |
| LS | -0.311 | -0.137 | -0.347 |
| LTM | -0.087 | 0.374 | 0.051 |
| FS | 0.138 | -0.318 | -0.416 |
| FCFP | -0.257 | 0.142 | 0.127 |
| FSC | -0.124 | 0.318 | -0.057 |
| DFG | -0.046 | -0.100 | 0.311 |
| CFS | 0.020 | 0.009 | -0.345 |
| FWg | -0.397 | -0.233 | -0.022 |
| FL | -0.060 | -0.124 | 0.084 |
| FW | -0.419 | -0.092 | 0.269 |
| PW | -0.354 | -0.295 | 0.215 |
| SC | -0.051 | 0.444 | 0.153 |
| FFC | 0.050 | -0.093 | 0.100 |
| FF | 0.020 | -0.315 | 0.366 |
| RPM | -0.046 | -0.093 | -0.162 |
| SCC | -0.155 | 0.196 | -0.213 |
| OHSW | -0.389 | -0.019 | -0.235 |
zSE, sex expression; IL, internode length; LS, leaf size; LTM, leaf teeth at the margin; FS, fruit shape; FCFP, fruit cork formation and pattern; FSC, fruit skin color; DFG, depth of fruit grooves; CFS, creasing at the fruit surface; FWg, fruit weight (g); FL, fruit length (cm); FW, fruit width (cm); PW, placenta width (cm); SC, sugar content (Brix); FFC, fruit flesh color; FF, fruit flavor; RPM, resistance to powdery mildew; SCC, seed coat color; OHSW, one-hundred seed weight (g).
과육색 구별 분자 표지 적용과 염기서열 분석
과육색은 멜론의 품종 육성에 매우 중요한 형질 중의 한가지로 이에 대한 표현형과 유전형을 각각 조사하고 그 상관관계를 비교하였다. 과육색은 총 5개의 표현형으로 나누었으며, 오렌지색 87개, 백색 75개, 녹색 21개, 그리고, 카로티노이드가 미미하게 축적된 것으로 보이는 유백색 20개와 황색 2개 순이었다. 또한, 유전형을 조사하기 위해 과육색을 결정하는데 중요한 기능을 한다고 보고된 CmOr유전자를 증폭시키는 프라이머 OrF4와 OrR15를 적용하여 표현형과 유전형을 비교하였다. PCR 반응에 의해 총 219개 유전자원 중 205개에서 예상 사이즈인 478 bp를 확인하였다. 염기서열을 분석하고 BLASTN을 이용하여 상동성을 검색한 결과, 멜론 염색체 9번에 위치한 CmOr 유전자가 정확하게 증폭되었음을 확인하였고(Accession No. LN713263) (Fig. 2, supplementary data 3), 증폭된 PCR산물은 HinfI을 처리하여 유전적 다형성을 조사하였다. 이 효소는 CmOr유전자의 SNP1 위치를 인식하여 HinfI을 처리하면 녹색과 흰색 과육에서는 478 bp, 오렌지 색의 경우는 139 bp와 339 bp로 나뉘어지는 것으로 보고되었다(Tzuri et al., 2015). 흰색 과육 표현형을 가지는 유전자원은 총 75개로 63개 유전자원에서는 예상대로 478 bp만이 관찰되었으며, 2개 유전자원(K126991, K189463)에서는 339 bp와 139 bp의 예상외의 밴드 패턴을 보였다(Fig. 3, Table 3). 그리고, 10개 유전자원에서는 3개 밴드 사이즈가 관찰되어 이것은 유전적으로 heterozygous하거나 불완전하게 digestion된 것으로 추측되었다. 녹색 과육의 경우는 총 21개 유전자원이 모두 478 bp의 1개 밴드만을 나타내었으며, 오렌지 과육색을 보이는 유전자원은 총 87개로 이중 63개 유전자원이 HinfI에 의해 339 bp와 139 bp만을 보였으며, 2개 유전자원(805488, K189912)에서는 478 bp만이 관찰되어 예상 외의 밴드 사이즈를 보였다(Fig. 4 , Table 3). 그 외에 K189460와 K189471의 2개 유전자원에서는 밴드가 증폭되지 않았다. 또한, 20개 유전자원에서는 3개 밴드 사이즈가 나타났는데, 원인으로는 흰색 과육색에서와 같이 유전적으로 heterozygous하거나 불완전하게 digestion 된 것으로 추측되었다. 유백색 표현형을 보이는 유전자원은 총 20개로 HinfI 처리 후, 13개 유전자원에서는 예상대로 478 bp가 보였으나 4개 유전자원(K189258, K189265, K189287, K189442)에서는 339 bp와 139 bp가 관찰되었다(Fig. 5, Table 3).
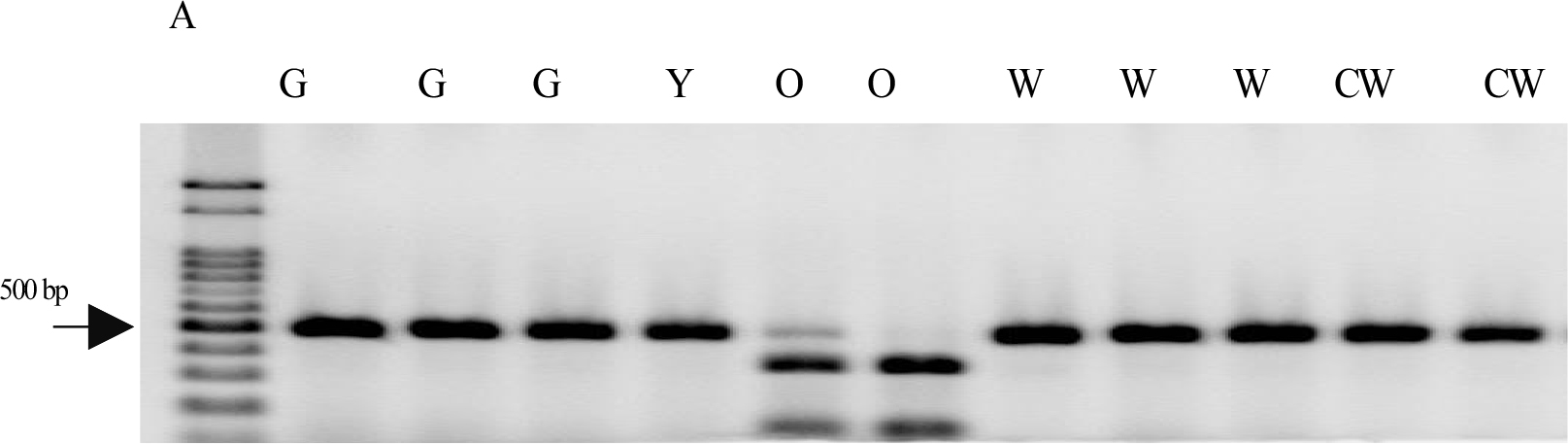
Fig. 2.
Genotyping of CmOr in melon germplasm. Primers OrF4 and OrR15 amplified a 478 bp fragment from the CmOr gene. Digestion of the PCR products with HinfI produced 339bp and 139bp fragment in most of orange-fleshed accessions. G, O, CW, W, and Y indicate accessions with green-, orange-, creamy-white -, white-, and yellow-fleshed fruits, respectively.
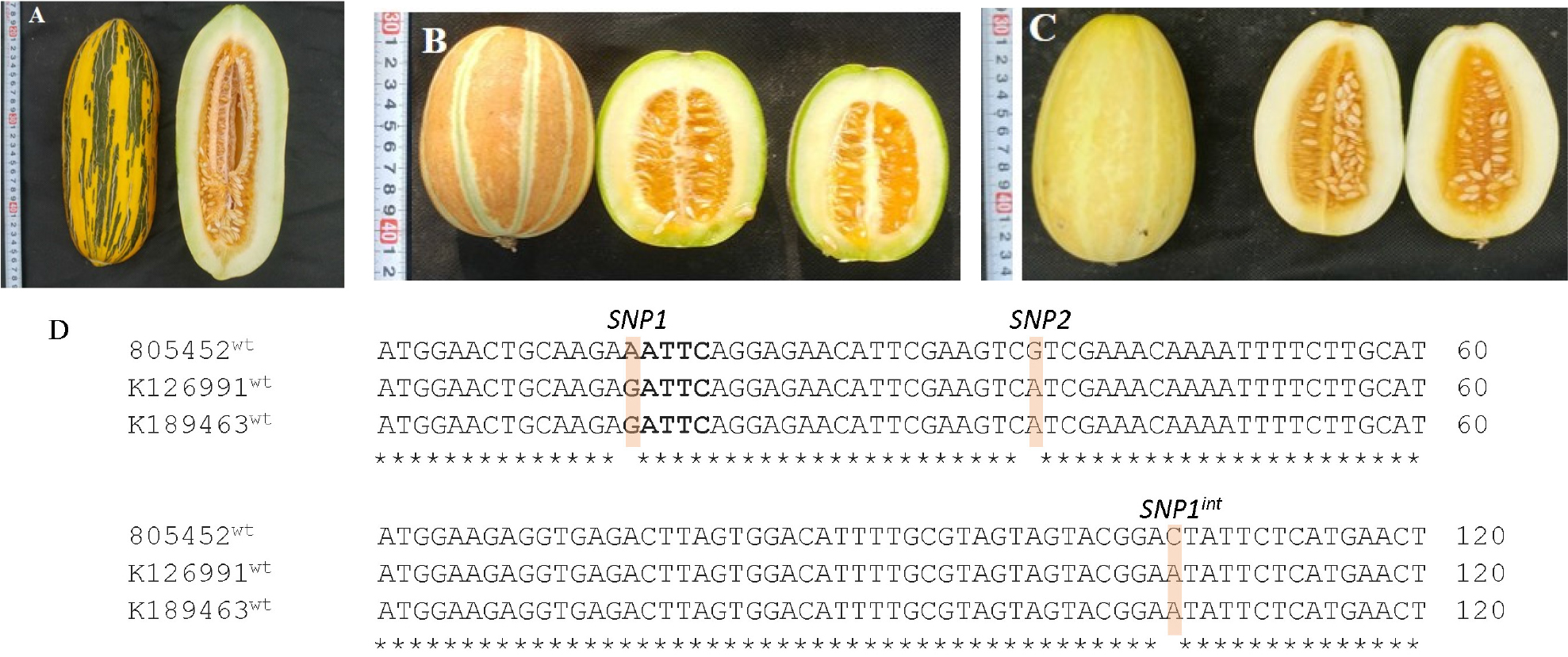
Fig. 3.
Fruit morphology and CmOr sequence analysis of three melon accessions with white flesh color. Photographs of the fruits of melon accessions 805452 (A), K126991 (B), and K189463 (C). (D) Alignment of the partial CmOr CDS amplified using primers OrF4 and OrR15. SNP1, SNP2, and SNP1int (Tzuri et al., 2015) are indicated. wt means white. 5’-GATTC-3’ HinfI restriction site was written in bold letter.
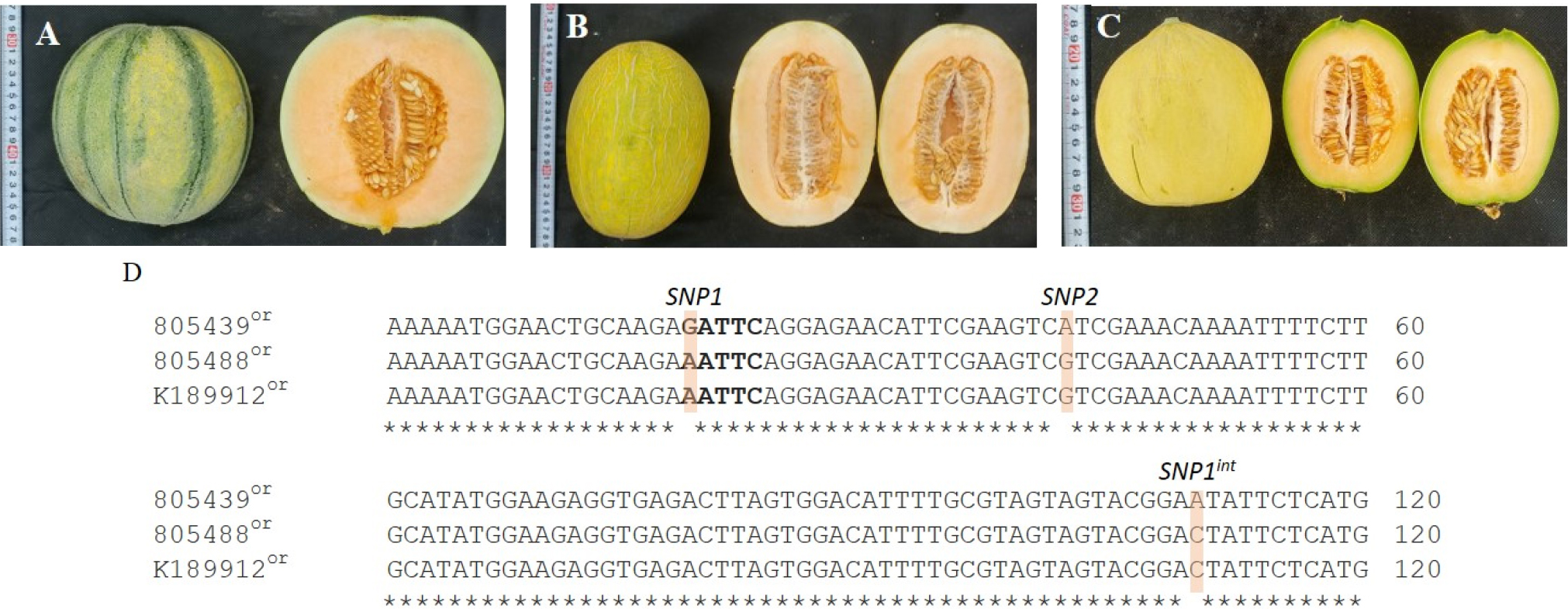
Fig. 4.
Fruit morphology and CmOr sequence analysis of three melon accessions with orange flesh color. Photographs of the fruits of melon accessions 805439 (A), 805488 (B), and K189912 (C) . (D) Alignment of the partial coding sequence (CDS) of the CmOr gene amplified using primers OrF4 and OrR15. Single nucleotide polymorphisms (SNPs), SNP1, SNP2, and SNP1int (Tzuri et al., 2015) are indicated. or means orange. 5’-GATTC-3’ HinfI restriction site was written in bold letter.
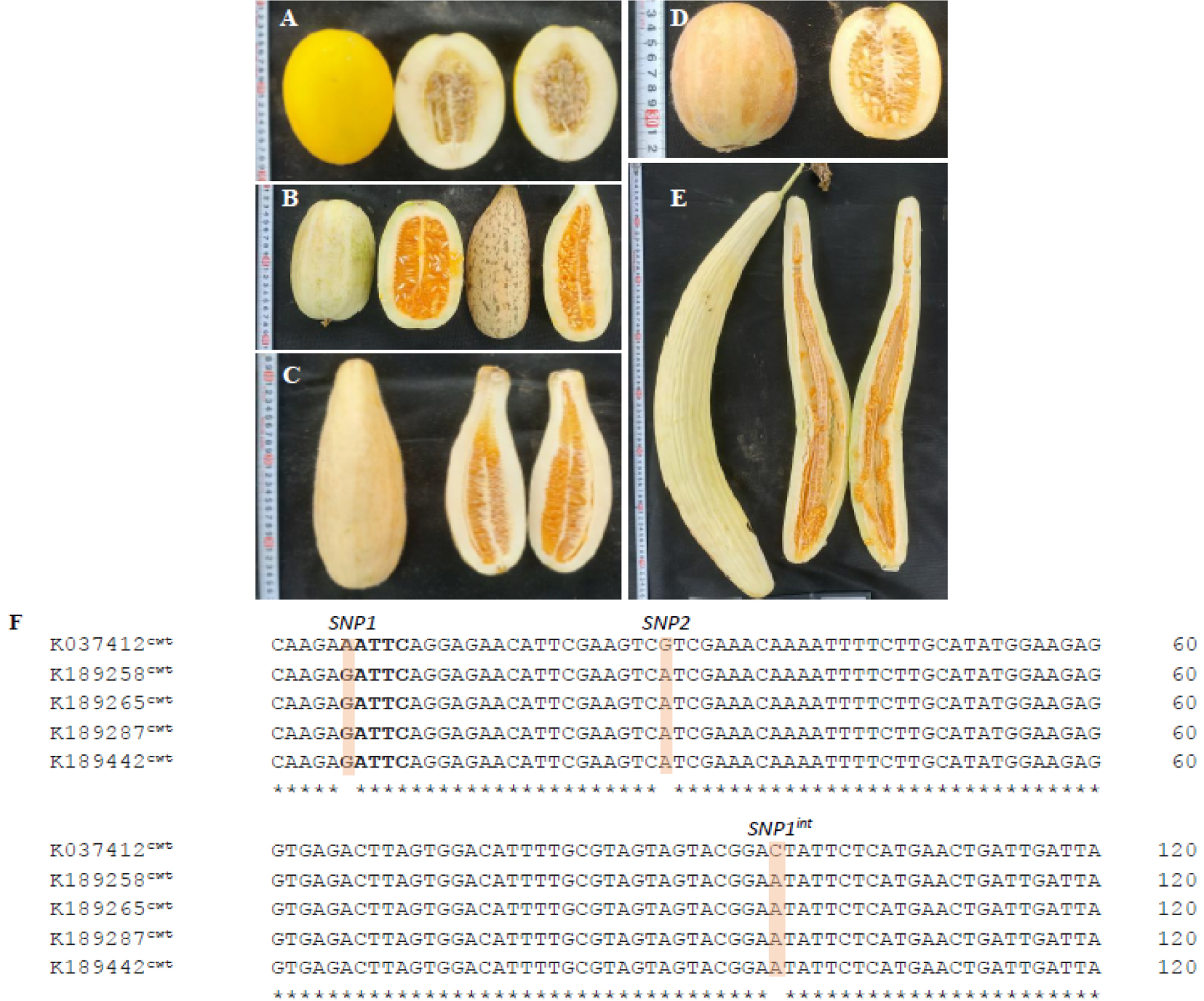
Fig. 5.
Fruit morphology and CmOr sequence analysis of five melon accessions with creamy-white flesh color. Photographs of the fruits of melon accessions K037412 (A), K189258 (B), K189265 (C), K189287 (D), and K189442 (E). (F) Alignment of the partial CmOr CDS amplified using primers OrF4 and OrR15. SNP1, SNP2, and SNP1int (Tzuri et al., 2015) are indicated. cwt means creamy-white. 5’-GATTC-3’ HinfI restriction site was written in bold letter.
Table 3.
Number of melon accessions and polymorphic band patterns with HinfI digestion amplified fragment by OrF4 and OrR15
이들의 염기서열을 분석한 결과, 흰색 과육에서는 엑손2에 위치한 SNP1위치에서는 염기 서열이 ‘A’로 나타나 HinfI에 의한 digestion이 일어나지 않아 478 bp만 관찰되었으나 염기서열이 ‘G’로 나온 유전자원이 2개(K126991, K189463) 존재하여 이 경우는139 bp와 339 bp의 2개 밴드가 관찰되었다. 그리고, 엑손 2에 위치한 SNP2에서는 ‘CGA’ (arginine)인 것이 일반적이나 ‘CAT’ (histidine)로 바뀐 것으로 나타났다. 또 다른 인트론 2에 위치한 SNP1int 위치에서는 ‘C’가 ‘A’로 바뀌는 다형성을 나타내었다(Fig. 3). 오렌지색 과육의 경우 SNP1에서는 염기서열이 ‘G’로 HinfI에 처리에 의해 139 bp, 339 bp가 밴드가 나오는 것이 일반적이나 digestion이 일어나지 않은2개 유전자원(805488, K189912)에서는 염기서열이 ‘A’ 임을 알 수 있었다. 또한, SNP2에서도 ‘CAT’(histidine)가 아닌 ‘CGT’(arginine)의 염기서열을 가진 것으로 분석되었다(Fig. 4). 또한, 카로티노이드가 미미하게 축적된 것이라고 추정되는 유백색의 유전형을 조사한 결과, 백색과 같은 유전형을 나타내었고, HinfI 처리에 의해 2개 밴드로 나뉘어지는 유전자원은 모두 4개(K189258, K189265, K189287, K189442)로 백색 과육의 경우와 동일하게SNP2 위치에서 염기가 ‘G’에서 ‘A’로 되어 변화되어 있었다(Fig. 5).
고 찰
본 연구에 사용된 멜론 219개 유전자원은 24개국에서 수집되었다. 지역별로 보면 140개(64%) 유전자원이 아시아 지역에서 수집된 것이었으며, 아시아와 유럽에 걸쳐 있는 러시아와 터키에서 수집된 자원이 13개였으며, 유럽에서 수집된 자원은 33개였다. 유전자원이 아시아 지역에 편중된 것은 Lee et al. (2020)이 언급한 바와 같이 국가 기관이 유전자원을 보유한 나라가 아시아 원산 유전자원을 많이 갖고 있기 때문일 것이며, 아프리카 원산의 유전자원이 없는 것도 관련 전문 기관이 없기 때문일 것으로 추정된다.
본 연구에서는 멜론의 생육 특성 중 과실을 중심으로 조사하였으며, 당도가 13 brix 이상의 유전자원이 5개(2%) (807688, 906954, 906965, 909847, K189600)가 있었으나 과실이 열리는 개수가 식물체 1개당 1.5개에 불과하여 당도와 수량성이 높은 멜론 유전자원을 획득하는 것은 용이하지 않았다. 이러한 원인으로 명확히 알려진 바는 없으나 이 유전자원들에 대해서는 저당도 유전자원들과 함께 비교 재배 실험을 하는 경우 좋은 연구 재료가 될 것으로 추정된다. 또한, 다른 중요한 형질인 흰가루병 저항성에 대해서도 조사하였는데, 완전히 저항성인 유전자원은 없었으며, 중도저항성인 유전자원이 43개 있었다. 멜론의 흰가루병 저항성에 관한 연구는 매우 활발하여 흰가루병을 발생시키는 균(Podosphaera xanthii)은 race가 다양하여 국내에서는 1, N1, N2, A, S, 그리고 5가 있다고 보고된 바 있다(Kim et al., 2016). 그리고, 각 race에 대한 저항성 유전자(좌)가 존재하는 것으로 알려져 있다(Howlader et al., 2020; Li et al., 2017; Zhang et al., 2013). 흰가루병 저항성과 감수성을 판단하는데 기준이 되는 총 9개 계통을 동일 하우스 내에서 재배하여 저항성을 관찰한 결과, 완전한 저항성을 나타내는 유전자원은 없었다. 이것은 2개 이상의 race가 복합적으로 존재하거나 심겨진 저항성 계통으로는 판단할 수 없는 또 다른 race가 존재하기 때문인 것으로 추측되었다. 하지만, 14개 유전자원(K189520, K189547, K189797, K189802, K189820, K189842, K189853, K189856, K189857, K189864, K189865, K189900, K189912, K189919)은 다소 저항성을 나타내었으므로(supplementary data 2), race 별로 저항성 검정을 한다면 좋은 재료 연구 재료가 될 것으로 추측된다.
PCA 분석 결과에서 3.7로 설정된 원을 벗어나는 유전자원은 총 17개(805439, 805484, 805488, 906968, 906954, K138764, K151093, K189219, K189337, K189376, K189442, K189462, K189553, K189767, K189843, K189848, K189857)였으며, 인도가 8개로 가장 많았는데, 이것은 인도 원산의 멜론 유전자원이 본 연구에서 가장 많이 사용되었기 때문으로 추측되며, 외에, 스페인, 이란, 우크라이나, 우즈베키스탄, 카자흐스탄, 투르크메니스탄 등의 유전자원이 각 1개씩 있었으며, 원산지를 알 수 없는 유전자원이 3개 포함되어 있었다.
멜론의 과육색은 농업유전자원센터의 생육 특성 조사 기준에 따라 총 5개 오렌지색, 백색, 녹색, 유백색, 황색으로 나누어 표현형을 조사하였다. 국내는 녹색의 머스크타입 멜론이 주로 소비되나 유럽, 일본 등은 오렌지색 과육도 많이 소비되는 등 지역에 따라 선호하는 타입이 다양하여 과육색은 육종가들의 중요한 육종 목표 형질 중의 하나이다. 총 219개의 유전자원 중 205개의 유전자원에서 과육색 표현형과 유전형을 조사할 수 있었다. 표현형 별로 유전자원의 개수를 보면 오렌지색과 백색이 대부분을 차지하였으며, 녹색 과실은 21개에 불과하였는데, 이것은 녹색이 오렌지색에 대해 열성이기 때문이라고 추측되었다(Clayberg, 1992). 그리고, CmOr 마커를 적용한 결과 총 15개 유전자원에서는 명확한 밴드를 관찰할 수 없었는데, 이중 13개(805480, K189117, K189121, K189179, K189211, K189501, K189704, K189778, K189792, K189802, K189824, K189856, K189878)에서는 매우 약한 밴드가 검출되었다. 그리고, 2개 유전자원(K189460, K189471)에서는 밴드가 검출되지 않았으며, PCA 분석 결과 이 두 개 유전자원은 매우 가까이 분포하는 것으로 나타났으며, 과육색은 모두 오렌지 색이었고 인도가 원산지였다(Fig. 1). 위 결과에 따라 이 두 가지 유전자원은 생육 특성이 매우 유사한 것으로 보였으며, 소수의 멜론 유전자원에서는 이 프라이머로 CmOr 유전자가 증폭되지 않는 그룹이 있을 것이라 추측되었다. 또한, 제한 효소 처리에 의해서 부분적으로 잘리지 않은 단편이 보이는 유전자원의 경우는 CmOr 유전자형이 heterozygous 한 상태이거나 disgestion이 불완전하기 때문으로 추정되었다. 과육색이 녹색인 유전자원 21개의 경우 표현형과 유전형이 모두 일치하여 Tzuri et al. (2015)이 보고한 바와 같이 CmOr은 녹색 과육 개체를 판별하는데 매우 유용하였다. 하지만, 오렌지색, 백색과 유백색 과육을 가지는 유전자원의 경우 완전히 일치하지 않았는데, 이것은 멜론의 과육색을 결정하는데 있어 아직 밝혀지지 않은 유전자(좌) 또는 유전형식이 있기 때문일 것으로 추측되었다.
동일한 박과 내에서 다양한 과육색을 가지는 작물로는 수박이 있다(Noh et al., 2020). 수박의 과육색은 크게 흰색(white), 밝은 노란색(pale yellow), 진한 노란색(canary yellow), 오렌지색(orange), 분홍색(pink), 적색(red), 주홍색(scarlet) 등으로 구분하며, 이들 과육색의 결정에는 멜론과 동일하게 다양한 카로티노이드 성분과 유전자간 우열 관계, QTL 등이 관여한다고 보고되었다(Bang et al., 2007, 2010; Liu et al., 2015; Yuan et al., 2021). 수박의 카로티노이드 성분은 neoxanthin, violaxanthin, xanthophyll, zeaxanthin 등으로 매우 다양하며, 이들의 함량과 비율에 따라 과육색이 결정된다고 하였다(Bang et al., 2010; Henderson et al., 1998; Liu et al., 2012; Tadmor et al., 2005). 과육색의 유전에 관한 연구도 활발히 이루어져 과육색을 결정하는 중요한 유전자(C)는 흰색(Wf)을 제외한 과육색에 대해 모두 우성임을 보고하였고(Henderson et al., 1998), py 유전자는 수박 과육의 연한 노란색(pale yellow)을 결정하는 중요한 유전자라고 보고하였다(Bang et al., 2010). 또한, 관련 유전자와 QTL에 대한 연구도 이루어져 Bang et al. (2007)은 수박 과육의 카로티노이드 합성에 관여하는 유전자인 lycopene β-cyclase (LCYB)를 이용한 CAPS 마커를 이용하여 적색(red)과 황색(canary yellow)를 구별하는 마커를 개발하였으며, Hashizume et al. (2003)은 적색 과육색에 관련된 2개의 QTL을 보고하였으며, Liu et al. (2015)은 염색체 4번에 적색 과육을 결정하는 중요한 QTL (LCYB 4.1)이 존재한다고 보고하였다. 또한, ClPHT4;2 는 카로티노이드 축적에 관여함으로써 수박의 과육색을 조절한다고 보고하였다(Zhang et al., 2016). Yuan et al. (2021)은 수박의 전사체 분석을 통해 오렌지색 과육 발현에 중요한 유전자인 Cla018406의 존재를 확인하였으며, 이 유전자도 CmOr과 동일한 DnaJ cysteine-rich domain을 가진 chaperone 유전자라고 보고하였다. 또한, CmOr과 homologous하다고 알려진 브로콜리의 BoOr 유전자는 카로티노이드 축적에서 가장 중요한 기능을 하는 phytoene synthase (PSY)의 번역후 과정을 조절함으로써(posttranslational control) 카로티노이드 축적에 관여한다고 보고하였다(Lu et al., 2006). 그리고 염기서열의 다형성을 분석한 결과 멜론과는 달리 염기서열의 결실(deletion)에 의해 기능이 좌우된다고 하였다(Welsch et al., 2020). 파파야(Carica papaya)를 대상으로도 과육색에 관한 연구가 수행되어(Yan et al., 2019) 적색 또는 노란색 과육을 결정에는chromoplast-specific lycopene β-cyclase (CpCYC-b) 유전자가 lycopene을 베타카로틴으로 전환함으로써 과육색을 결정짓는 중요한 기능을 한다고 알려져 있다. 이 유전자의 경우는 2 bp 염기서열의 삽입이 일어나면 premature stop codon이 발생하여 베타카로틴이 축적되지 않는다고 하였다. 저자들은 이를 기반으로 만들어진 마커로 F2 집단을 대상으로 유전형을 조사하여 98% 표현형과 일치율을 보였는데, 100% 일치율이 아닌 이유로는 CpCYC-b 가 염색체의 재조합이 매우 많이 일어나는 부분에 위치하기 때문이라고 하였다. 이와 같이, 과육색의 결정은 식물종에 따라 관여하는 유전자는 다양하고 여러 allele이 존재하여, 멜론의 경우도 이와 같이 다양한 기작으로 멜론의 과육색이 결정되는 것으로 추측된다.
멜론은 국내에서는 1년에 한번 또는 두 번만 재배되어 육종 기간이 오래 걸리는 작물중의 하나이다. 특히, 과육색은 과실이 열려 수확될 때까지 그 표현형을 알기가 어려워 새로운 유전자원을 도입하여 품종 개발을 하는 경우 상당한 시간과 노동력이 소요된다. 그러나, 본 연구에서 사용된 과육색 연관 마커를 적용하는 경우 과육색을 미리 예측할 수 있어 노동력과 비용을 절감할 수 있을 것으로 생각된다. 또한 본 연구에서 얻어진 다양한 유전자원의 생육조사 결과는 육종가들이 멜론 육종 프로그램에 새로운 유전자원을 도입하는 경우 유용한 방법 또는 자료로 이용될 수 있을 것으로 기대된다.
적 요
멜론은 세계 각지에서 재배되는 경제적으로 중요한 작물중의 하나이다. 본 연구는 농업유전자원센터에서 수집 보관중인 멜론 유전자원을 대상으로 다양한 생육 특성을 특성을 조사하고, 멜론의 중요한 육종 형질중의 하나인 과육색의 유전형과 표현형을 조사하여 멜론 육종에 필요한 육종 재료 확보를 위한 기초 자료를 마련하고자 수행되었다. 총 219개의 멜론 유전자원을 대상으로 19개의 생육 특성과 PCA분석을 수행하고, 멜론의 중요한 육종 형질중의 하나인 과육색의 유전형을 조사하여 표현형과 비교하였다. 과육색은 오렌지색, 백색, 녹색, 유백색, 황색의 5가지로 분류하였으며, 이중 오렌지색이 87개로 가장 많았으며, 그 다음으로 백색이 75개였다. 그리고, 오렌지색과 녹색 과육 구별용 마커를 적용한 결과, 녹색 과육 21개의 경우는 표현형과 유전형 일치율이 100%였으며, 오렌지색의 경우는 98%, 백색은 97%, 유백색의 경우는 80%의 일치율을 보였다. 표현형과 유전형이 일치하는 않는 총 8개 유전자원의 염기서열을 분석한 결과, 3곳의 위치에서 단일염기다형성(SNP; single nucleotide polymorphism)이 있었다. 이러한 결과는 멜론의 과육색을 결정하는 아직 알려지지 않은 유전기작이 존재한다는 것을 제시하였으며, 본 연구에서 얻어진 다양한 유전자원의 생육조사 결과는 멜론 육종에 유용하게 쓰일 것으로 생각된다.