서 언
재료 및 방법
두류 농업유전자원 현황
엽록체 유전자 분석 및 군집화
결 과
두류의 형태적 형질
엽록체 유전자의 이종상동성 분석
엽록체 유전자를 이용한 두류형질 유전력 추정
고 찰
적 요
서 언
두류(legumes or pulse)는 탄수화물, 단백질, 식이섬유, 비타민 등을 포함하는 복합 영양원으로서 식량자원으로 많이 이용되고 있다(Choi et al., 2018). 특히 단백질 함량이 높고 지방함량은 적어서 향료와 부 재료 첨가를 통해 풍미와 조직감을 육제품과 유사하게 제조할 수 있어서 육류를 대체하는 식물성 대체육인 ‘콩고기’ 식품의 소재로 활용되고 있다(Jeong and Jo, 2018; Seo et al., 2021). 또한 두류는 혈중 콜레스테롤 감소, 혈당과 인슐린 분비의 정상화, 항암, 항산화, 면역력 강화에 영향을 주는 2차 대사산물을 다양하게 함유하고 있어 건강관련 보조식품으로도 이용되고 있다(Lee, 2011). 이처럼 먹거리 및 건강식으로서 두류의 활용도는 높지만 수입 콩과의 가격경쟁으로 인한 수익률의 감소로 국내에서의 콩 자급률은 매년 점차적으로 낮아지고 있기 때문에 이에 대하여 높은 수량성과 고기능, 고품질의 품종개발과 관련 형질에 대한 연구와 조사가 요구되고 있다(Kim et al., 2019).
두류를 포함한 식물은 종에 따라 환경적 영향, 유전적 영향으로 인하여 다양한 형질로 표현되며, 그 중에서 유용한 형질은 산업적으로 이용되고 있다. 특히 농업분야에서는 작물의 종 간 형질의 차이를 비교하여 유용한 형질을 발굴함으로써 생산성과 품질이 우수한 유전자원을 선발하고 관련 신품종을 개발 등과 같이 형질의 다양성을 활용하고 있다. 여타 작물과 마찬가지로 두류도 종 간의 형질 차이를 분석하여 우수자원 발굴 및 품종 개발을 위한 유용한 연구결과를 제공하고 있는데, 예를 들어, 최 외(Choi et al., 2018)는 열대 및 아열대 원산지인 렌즈(렌틸)콩(Lens culinaris), 병아리콩(Cicer arietinum), 송이콩(Cyamopsis tetragonoloba) 유전자원에 대한 개화일수, 생육일수의 차이와 조단백질, 조지방, 식이섬유의 평균 함량에 대한 종에 따른 차이를 검정함으로써 유용자원 선발을 한 두류의 형태적 형질의 차이를 정리하여 연구자, 육종가 등 다양한 수요자에게 정보를 제공하고 있다. 선택된 우수형질은 환경 등의 외부 요인에 의해 쉽게 변하지 않고 다음 세대로 유전될 수록 유용하다고 할 수 있고, 일부 형질, 특히 양적형질은 유전적인 영향보다 환경 요인에 민감하게 반응하여 작물의 형질이 변화하기도 한다(Jung et al., 1999). 그러므로 이용가능한 형질의 선택과 이용에 있어서 형질을 고정(fixation)하거나 혹은 동일한 형질발현을 위하여 재배조건을 유사하게 설정하는 등과 같이 증식 및 재배계획을 면밀히 수립해야 한다(Beavis et al., 2021). 그러기 위해서는 형질의 변화에 작용하는 환경적 혹은 유전적 영향 관계를 설명할 수 있는 정보가 요구되며, 이는 유전력(heritability)를 통해서 추정할 수 있다.
유전력은 유전이 가능한 변이의 수준과 세대로 유전되는 정도를 말하며, 높은 유전력의 형질은 변이 발생의 요인이 환경보다는 유전적 영향이 더 크게 작용함을 의미한다(Lee et al., 2021), 유전력은 광의의 유전력(broad sense heritability)과 협의의 유전력(narrow sense heritability)으로 세분화되는데 광의의 유전력은 조사된 분산(환경분산과 유전분산)에서 유전분산이 차지하는 비율, 협의의 유전력은 상가적 유전자 효과(additive gene effects)에 대한 유전분산의 비율로 평가한다. 일반적으로 육종분야에서는 유전력이 높은 형질, 특히 상가적 유전자 효과에 의한 높은 형질의 보존으로 협의의 유전력이 높은 형질을 기반한 육종소재 발굴을 선호하는 것으로 알려져 있다(Lee et al., 2021). 그러므로 우수형질을 발굴하기 위해서는 요구되는 형질에 대한 유전력의 추정이 요구되고 부모세대와 자식세대 간 형질의 유전력을 분석하기 위해서는 복잡한 교배조합을 위한 실험 설계, 작물을 증식하는데 필요한 인력, 비용, 시간 등이 크게 요구되므로 보다 효율적으로 분석하기 위한 대안이 요구된다(Lee et al., 2021).
직접조사나 실험을 제외하고 우회적으로 유전력을 추정하기 위한 방법으로 계통발생 신호(phylogenetic signal) - 서로 유사한 종의 특징, 경향, 혹은 패턴(pattern) –을 이용할 수 있다(Blomberg et al., 2003). 기공의 길이, 면적과 밀도, 엽맥의 밀도, 잎 두께 등과 같은 식물의 구조적 특징은 계통발생 신호와 관련이 있는 것으로 알려져 있으며, 유전력 또한 높은 값을 보인다는 점에서 계통발생 신호와 유전력은 서로 성관성이 있음을 알 수 있다(Ávila-Lovera et al., 2023; Yassin et al., 2022). 계통발생 신호는 식물 유전자의 계통분류 및 진화와 관련된 유전자 정보(gene information)를 이용하여 추정할 수 있는데 식물의 엽록체 유전체는 계통발생 신호를 추정하기에 적절하다(Yaradua and Alzahrani, 2019). 왜냐하면 식물의 엽록체 유전체는 크기가 비교적 짧은(110~150 kb) 반면, 식물생장에 필요한 에너지를 생산하는 필수 유전자(essential genes)를 포함하고, 유전체 재조합(DNA recombination), 수평적 유전자 이동(lateral/horizontal gene transfer)이 거의 발생하지 않으며, 단친성으로 유전되는 특징 때문에 식물, 미생물 등 엽록체 유전체를 가지는 생물의 계통분류, 진화, 미기록종 발굴연구에 이용되고 있기 때문이다(Rivas et al., 2002; Sim et al., 2022; Sousa et al., 2020). 따라서 두류의 특정 형질 차이에 대한 환경적 요인과 유전적 영향의 관계는 두류의 각 종에 대한 엽록체 유전자의 차이와의 상관성 분석을 통해서 형질에 대한 유전력을 예상하고 형질변이에 대한 환경적, 유전적 요인관계를 추정할 수 있을 것이다.
본 연구에서는 국립농업과학원 농업유전자원센터에서 제공하고 있는 두류 6종(렌즈콩, 리마콩(Phaseolus lunatus), 병아리콩, 비둘기콩(Cajanus cajan), 송이콩, 제비콩(Lablab purpureus))을 대상으로 종에 따른 형태적 형질의 차이와 NCBI (Dobrogojski et al., 2020)에서 제공하는 6종에 대한 엽록체내 유전자 서열을 이용한 유전적 차이를 분석하였다. 그리고 유전적 영향관계를 분석하기 위하여 서열 간의 차이와 형태적 형질의 차이에 대한 상관관계를 분석하여 형질의 유전적 관계와 유전력을 추정하였다. 본 연구를 통해서 두류의 형질에 대한 유전적 영향을 염색체 유전자 서열과의 상관관계를 통해 두류 형질을 추정함으로써 두류 형질의 유전적 영향 관계를 적용한 육종기술의 개발과 형질 유지 및 관리를 위한 정보를 제공하고자 한다. 또한 두류 뿐만 아니라 다른 유용작물의 형질에 대한 유전적, 환경적 요인의 발굴 및 증식 설계에 본 연구의 방법을 활용함으로써 다양하고 심도있는 농업연구와 농산업발전에 기여할 수 있기를 기대한다.
재료 및 방법
두류 농업유전자원 현황
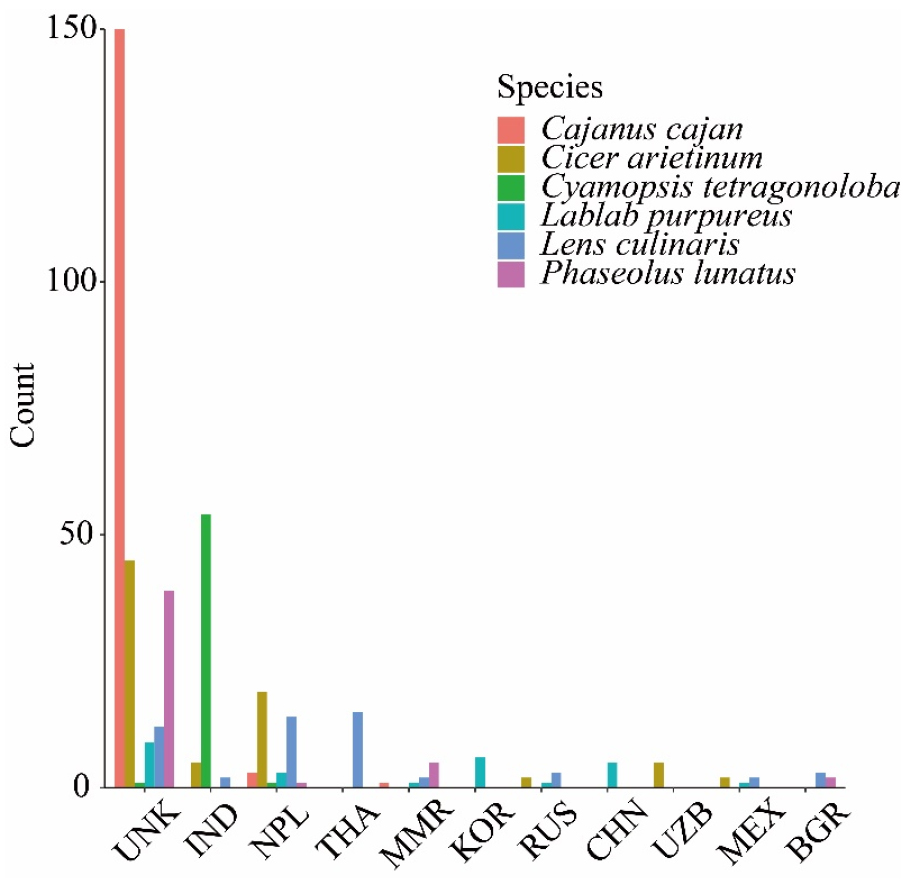
본 연구에서 사용한 두류는 농업유전자원센터에서 수집 및 보존하고 있는 비둘기콩(156자원), 병아리콩(80자원), 송이콩(62자원), 제비콩(36자원), 렌즈콩(69자원), 리마콩(49자원)으로 총 6종, 452자원을 대상으로 연구를 수행하였다. 이 들 자원의 원산지를 분류했을 때, 256자원(56.64%)의 원산지가 불명(UNK, Unknown origin)으로 특히 비둘기콩이 150자원에 해당하였다. 주요 원산지를 살펴봤을 때, 병아리콩은 네팔(NPL)에서 19자원, 송이콩은 인도(IND)에서 54자원, 렌즈콩은 태국(THA)과 네팔에서 각각 15자원 및 14자원으로 주로 인도, 네팔, 태국의 자원이 수집되었다. 이 밖에 미얀마(MMR), 한국(KOR), 러시아(RUS), 우즈베키스탄(UZB) 등 다양한 국가에서도 소수 수집되었으며, 한국에서는 제비콩이 6자원 수집되었다(Fig. 1).
두류의 유전자원 관리 및 유용자원 선발 등을 위하여 전주 농업유전자원센터(전라북도 전주시)에서 2017년부터 2019년까지 두류 6종을 증식하면서 형태적 형질을 조사하였고(Choi et al., 2018), 조사 및 분석결과는 농업유전자원관리시스템(Germplasm Management System, GMS) 및 씨앗은행(https://genebank.rda.go.kr)을 통해서 제공하고 있다(Park et al., 2020b). 본 연구에서 사용된 두류의 형태적 형질은 6종에서 공통으로 조사된 파종기(seeding date), 개화기(flowering date), 성숙기(maturing date), 협당 립수(the number of seeds per pod), 100립중(100-seeds weight), 조단백질(crude protein), 조지방(crude oil), 조섬유(crude fiber) 및 식이섬유(dietary fiber) 조사결과를 사용하였다(Table 1). 파종기, 개화기, 성숙기는 기후조건에 따른 시기가 다르기 때문에 파종기에서 개화기까지의 개화일수(days to flowering), 파종기에서 성숙기까지의 생육일수(days to maturity), 개화기에서 성숙기까지의 성숙일수(period from flowering and maturity)를 줄리안 일수(Julian date)로 변환하여 계산하였는데, 이 들은 작물의 생장기간과 관련이 있는 것으로 알려져 있다. 그리고 협당 립수와 100립중은 콩의 수량 구성을 판별할 수 있는 기준으로 활용되는데 수량의 증가를 위한 요인들을 선발할 때 유용하다고 알려져 있다(Cho et al., 2006; Kim et al., 2013; Younis et al., 2008). 그리고 식품으로서 중요한 지표 중의 하나인 영양성분의 함량은 식량으로서 활용성과 고품질을 위한 육종 및 신품종 개발, 관련 산업의 소재로 사용할 수 있는 척도로 이용할 수 있기 때문에 고기능성 식품생산을 위한 자원 발굴에 적용할 수 있는 형질이라 선택하였다(Seo et al., 2017).
Table 1.
The number of germplasms surveyed genetic traits in six species
엽록체 유전자 분석 및 군집화
식물의 엽록체 유전체는 종 분류, 계통분석, 진화연구 등과 같이 식물의 유전적 관계를 이해하고 근연종 검색을 통한 유전적 특징을 추정하기 위한 연구재료로 많이 활용되고 있다(Palmer, 1985; Tian et al., 2021). 두류 6종에 대한 종 수준에서 형질에 대한 유전력을 추정하고자 엽록체 내 유전자를 선발하기 위하여 NCBI의 Genbank와 refseq (O'Leary et al., 2015)에 등록되어 있는 엽록체 유전체 정보를 Artemis 프로그램(Carver et al., 2011)을 이용하여 종간의 계통발생 신호를 유전자 서열의 차이로 분석하였다(Table 2). 엽록체 유전체의 구조를 분석했을 때, 병아리콩과 렌틸콩은 특이하게도 식물의 엽록체에서 나타나는 중복된 유전자집단 영역인 IR (inversed repeat)영역이 확인되지 않았는데 이는 아마도 IR영역의 손실(loss)이 발생했을 것으로 추정된다(Jansen et al., 2008; Sveinsson and Cronk, 2016). 엽록체 유전자에 대한 계통을 분석함에 있어서 비록 계통발생 신호의 부정확성에 대한 문제가 있지만(Gonçalves et al., 2019; Park et al., 2020a), 일반적으로 종분류를 위한 기초분석으로 일부 엽록체 유전자를 이용하는 것을 확대하여 본 연구에서는 엑손-인트론으로 분리된 유전자를 제외하고 총 472개 유전자 서열을 분석에 사용함으로써 계통발생 신호의 부정확성을 보완하고자 하였다.
Table 2.
Genetic features of six species using Artemis
| Features | Cajanus cajan | Cicer arietinum | Cyamopsis tetragonoloba | Lablab purpureus | Lens culinaris | Phaseolus lunatus |
| Accession ID | NC_031429 | NC_011163 | NC_037714 | MW169030 | KJ850239 | MW423611 |
| Size | 153,841 | 125,319 | 152,530 | 151,916 | 122,967 | 150,902 |
| Available CDSs (totalz) | 80 (80) | 73 (75) | 86 (88) | 81 (83) | 72 (73) | 80 (82) |
| tRNAs | 37 | 29 | 39 | 38 | 30 | 37 |
| rRNAs | 8 | 4 | 8 | 8 | 4 | 8 |
콩의 형태적 형질 정보와 엽록체 유전체 정보의 관계를 분석하기 위하여 거리행렬을 이용한 상관분석을 수행하였다. 형태적 형질 정보()는 수치형의 연속형 자료이기 때문에 거리행렬()은 0에서 1사이의 거리 값을 가지는 유클리드 거리(Euclidean distance) 계산식(식 (1))을 이용하였다(Sharifi et al., 2018).
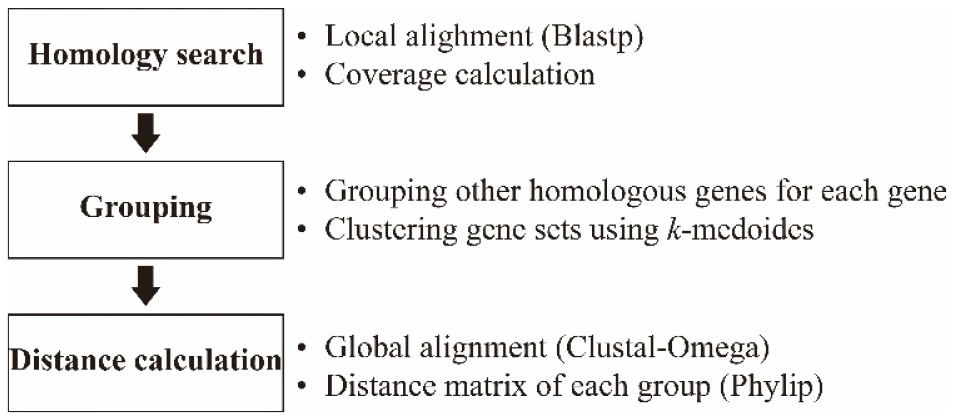
각 종의 엽록체 유전자에 대하여 계통발생 신호 추정을 위한 계통분류적 차이는 472개 유전자 서열을 Fig. 2와 같은 흐름도를 통해서 계산하였다.
유전자 유사도 검색(homology search) 단계에서는 모든 유전자서열을 대상으로 지역정렬 알고리즘기반의 blastp 프로그램(Altschul et al., 1990)을 이용하여 서열 간의 유사한 정도(유사도, )와 차이()를 식2와 식3으로 계산하였다. 그리고 그룹화 단계(grouping)에서는 각 유전자에 대하여 종별로 가장 유사한 유전자를 선별하여 그룹화하였다. 각 종의 유전체는 중복된 단백질 유전자가 존재하고 유사성의 차이가 있기 때문에, 6종에서 공통적으로 유사성이 높지만 다른 그룹에 이미 포함된 중복된 유전자도 새로운 유전자 그룹에 할당하여 총 455개의 유전자 그룹을 선발하였다. 그리고 각 유전자와 유전자 집단이 동일한 기능으로 유전된 유전자(이종상동성 유전자, orthologous gene)에 해당하는지를 확인하기 위하여 NCBI의 COG (Clusters of Orthologous Groups) 데이터베이스(Galperin et al., 2020)에 대하여 Blastp best hit 검색을 수행하였다. 추가적으로 단백질 유전자 가운데 중복된 유전자 혹은 서열이 유사한 유전자가 존재하기 때문에 개별적인 유전자 그룹과 더불어 대표객제기반 알고리즘인 K-medoids (Park and Jun, 2009)을 적용하여 식 3을 통한 를 바탕으로 유사한 유전자들을 군집화(clustering)화하여 군집단위에서 추정된 유전력도 함께 확인하였다.
455개의 유전자 그룹은 각각 거리계산(distance calculation) 단계에서 전역정렬 알고리즘(global alignment algorithm)기반의 Clustal Omega 프로그램(Sievers and Higgins, 2018)과 Phylip의 PROTDIST 프로그램을 사용하여 상관분석 및 계통적 거리행렬(phylogenetic distance matrix)을 계산하였다(Guillén-Escribà et al., 2021).
특성평가 정보에 대한 종 간의 거리행렬과 유전자간 거리와의 상관관계 분석은 R의 psych 라이브러리에 포함된 cor.test 함수에 피어슨 상관계수(Pearson’s coefficient)를 적용하여(Revelle and Revelle, 2022), 종 간의 유전자에 따른 계층적 차이와 형질의 차이에 대한 상관관계를 통해서 유전력을 추정하였다.
결 과
두류의 형태적 형질
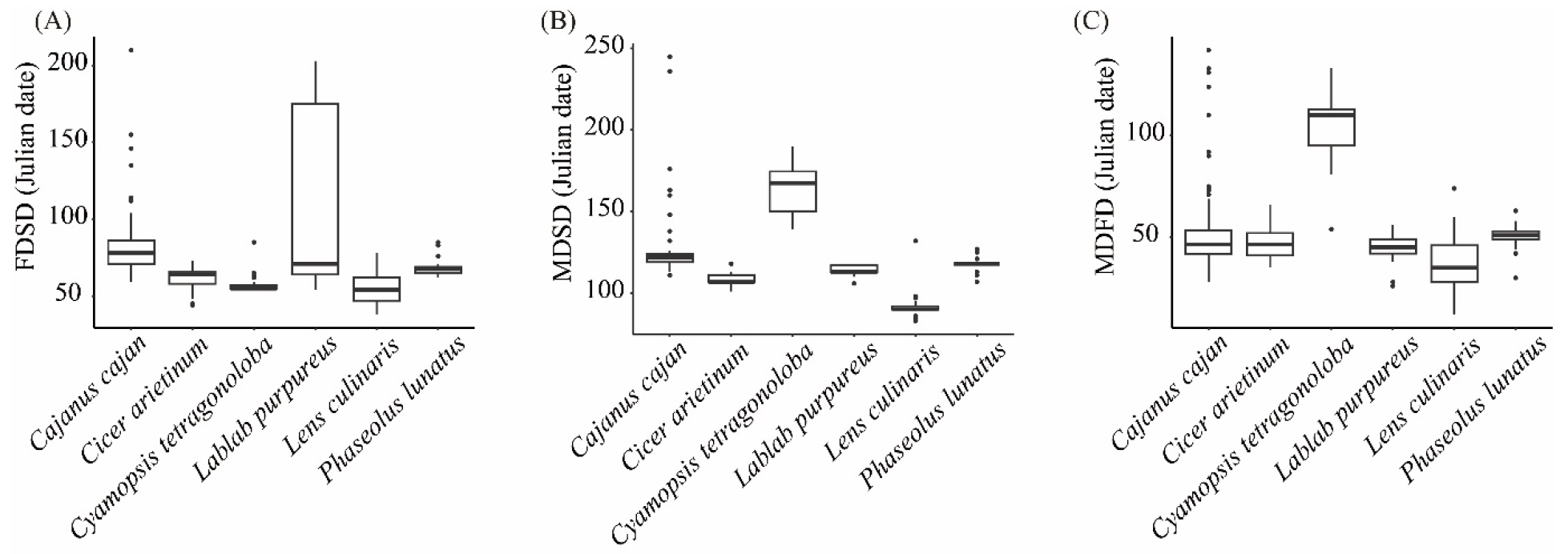
두류 6종에 대한 개화일수, 생육일수, 성숙일수를 분석했을 때, 렌즈콩은 개화일수(Fig. 3A), 생육일수(Fig. 3B), 성숙일수(Fig. 3C)에서 각각 평균 81.5일, 91.1일, 35.9일로 가장 짧은 기간을 보였다. 송이콩은 생육일수와 성숙일수에서 각각 평균 163.7일, 106.5일로 다른 종에 비해 가장 긴 기간으로 조사되었는데 다른 종에 비해 비교적 차이가 많은 것으로 나타났다. 제비콩은 개화일수에서 다른 종과는 달리 개화일수가 넓게 분포하였다(1사분위 64일~3사분위 175일). 각각의 개화일수, 생육일수, 성숙일수에 대하여 종에 따른 크루스칼-왈리스 분산분석(Kruskal-Wallis test)을 수행했을 때 유의적 수준(P-value <0.05)에서 종에 따라 차이가 있을 것으로 예상하지만 성숙일수의 경우 최고일수를 보인 송이콩을 제외하면 종에 따른 차이(표준편차)가 6.2일로 개화일수(10.7일, 최고일수 제외)와 생육일수(14.9일, 최고일수 제외)에 비해 큰 차이가 나타나지 않아서 실제 조사된 성숙일수를 바탕으로 종을 구분하기는 어려울 것으로 판단된다.
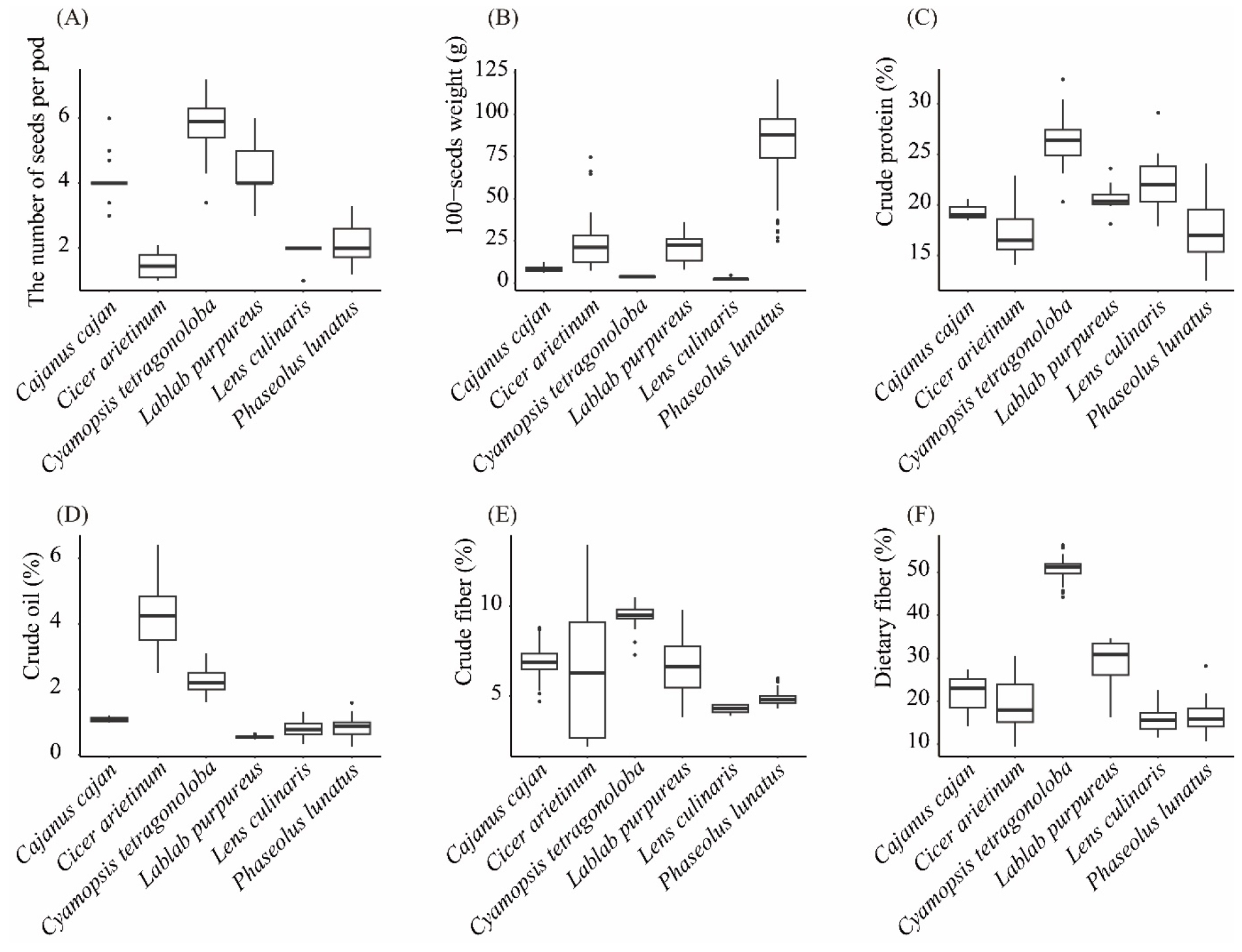
종에 대하여 각 형질특성을 분석했을 때, 협당 립수(Fig. 4A)는 송이콩이 평균 6.0개로 가장 많았으며, 100립중(Fig. 4B)은 리마콩이 79.5g으로 가장 높았다. 유용성분에 있어서 조단백질(Fig. 4C), 조섬유(Fig. 4E) 및 식이섬유(Fig. 4F)는 송이콩이 평균 26.4%, 9.5%, 50.7%로 가장 높았고 조지방(Fig. 4D)은 병아리콩이 평균 4.3%로 높은 함량비를 보임으로써 종에 대하여 유의적 차이(P-value <0.05)가 나타났다. 하지만 본 연구에 사용된 유전자원에 대한 형질 정보는 통계적 유의수준에 따른 종 간의 차이를 검정할 수는 있지만 동일한 지역에서 짧은 기간동안(2~3년) 세대교배를 하지 않은 유전자원을 증식, 재배하였기 때문에 순계(pure crop)와 자식세대 정보를 알 수가 없어서 종의 따른 형질의 유전력을 실험결과를 통해 추정하기가 어렵다. 따라서 엽록체 유전체 정보를 이용하여 종 수준의에서의 계통진화적 방법을 적용하여 형질의 유전력을 추정하고자 하였다.
엽록체 유전자의 이종상동성 분석
형질에 대한 유전적인 영향 관계를 추정하기에 앞서 각 종의 엽록체 유전자가 진화적으로 조상으로부터 동일한 기능이 유전된 이종상동성 유전자(orthologous gene)에 해당하는지를 분석하였다. NCBI의 COG 데이터베이스를 대상으로 Blastp best hit 검색을 수행했을 때, 두류 6종의 엽록체 내의 유전자 서열을 대상으로 확인된 이종상동성 유전자는 전체 472개 중에서 445개(93.3%), K-medoides기반의 유전자 군집을 기준으로는 총 83개중에서 76개(91.7%)으로 분류되어 두류 6종의 대부분의 유전자가 해당되었고, 기능에 있어서는 에너지 생산 및 보존(Energy production and conversion)과 단백질 합성(Translation, ribosomal structure and biogenesis) 관련 기능의 유전자 군집이 다수 확인되었다(Table 3). 따라서 식물생장과 유지를 위해 필요한 에너지원인 당(C6H12O6)을 합성하는 엽록체의 역할과 엽록체 유지에 필요한 유전자 집단 구성으로써 필요한 단백질 합성 기능으로 유전자가 구성되어 있고, 이 들은 이종상동성 유전자로서 계통적으로 동일한 기능이 유전되었을 것으로 추정할 수 있다. 따라서 이러한 엽록체 유전자의 특성을 고려하여 472개 유전자 가운데, 두류 6종에 모두 유사성이 있고 중복을 허용한 455개 유전자 그룹으로 구분하여 형질과의 상관관계를 분석하였다.
Table 3.
COG functions of the cluster of homologous genes
엽록체 유전자를 이용한 두류형질 유전력 추정
형질이 유전될 수 있는지를 추정하기 위하여 6종이 공통으로 포함된 455개 유전자 그룹을 대상으로 형태적 형질에 대한 종 수준에서의 차이와 유전적 차이의 상관관계(Pearson’s correlation)를 분석하였다. 그런데 각 형질의 상관관계 분석에 있어서 유의수준이 0.05 이하(P-value ≤0.05)일 때와 0.1 이하(P-value ≤0.1)일 때의 평균상관계수의 다른 유의수준과 비교했을 때 상대적으로 차이가 크지 않았다. 이는 엽록체 유전체 자체의 정보부족과 노지에서 증식한 작물의 불특정한 환경요인에 의한 형질차이로 인한 결과로 판단되어 본 연구에서는 유의수준 0.05 이하(p-value ≤0.05, 신뢰구간 95%)와 0.1 이하(p-value ≤0.1, 신뢰구간 90%)를 함께 고려하였다. 총 9가지 형질에서 유의수준이 0.05 이하일 때는 상관계수가 0.43 이상(평균 0.49)이었고, 유의수준이 0.1 이하인 경우 0.38 이상(평균 0.44)으로 상관관계가 다소 높은 것으로 확인되었다. 반면에 유의수준 0.05와 0.1을 초과하는 경우에는 각각 0.23, 0.18 이하로 낮은 상관계수를 보였다(Table 4). 따라서 유전자 및 유전자 그룹에서 좁게는 신뢰수준 95% 범위, 넓게는 90% 범위에서 상관관계가 있음을 추정할 수 있었고 유전자 군집에서도 동일한 결과를 확인할 수 있었다.
Table 4.
Average correlation coefficient for genes and clusters at each P-value
유의수준에 따른 유전자 수를 분석했을 때(Table 5), 개화일수에 대하여 상관성이 있는 유전자 수가 유의적 수준이 0.05 이하인 경우에 62.86%를 보여 유전적 분류관계와 높은 것으로 확인되었으며, 기 연구결과와 유사하였다(Bekele et al., 2012; Seo et al., 2017; Toker, 2004). 그런데 생육일수는 0.05 이하 유의 수준에서 34.95%를 보였지만 0.1 이하(p-value ≤0.1)에서는 62.42%를 보였다. 일반적으로 유전적 영향에 따른 유전력의 수준을 상(high, 0.6>), 중(intermediate, 0.3~0.6), 하(low, 0.3<)로 분류하고 있는 점을 고려할 때 생육일수는 환경과 유전적 영향이 함께 작용할 것으로 추정할 수 있다(Bista et al., 2022; Khan et al., 2020). 성숙일수는 다른 누에콩 연구의 경우 비교적 높은 유전력으로 알려져 있지만(Mesfin et al., 2021; Tola et al., 2023) 본 연구에서는 유의수준 0.05와 0.1에서 각각 5.71%와 8.57%로 상관성을 추정할 수 없었다. 이는 Fig. 3(C)에서와 같이 종 간 성숙일수의 차이가 크지 않는 점과 비록 다수의 엽록체 유전자를 대상으로 분석했지만 모계유전, 낮은 유전변이 수준 등과 같은 엽록체 유전자의 특성으로 나타나는 낮은 계통발생 신호(Ackerly, 2009), 유전자 트리(gene tree)와 종 트리(species tree)간의 불일치(Nge et al., 2021), 모호한 형질의 차이, 그리고 성숙일수와 같이 다소 높은 유전력과 낮은 유전적 진전(genetic advance)때문에 나타나는 유전자의 우점(dominance) 혹은 상위적 효과(epistatic effects)의 영향(Kumar et al., 2017) 등의 복잡한 요인이 작용했을 것으로 판단된다. 형태적 형질에서는 협당 립수와 조지방이 유의적으로 상관성이 있는 유전자 그룹이 유의수준 0.05 이하일 때, 각각 69.45%, 57.14%, 0.1 이하에서는 76.04%, 72.53%로 상관성이 높은 반면, 100립중, 조단백질, 조섬유 및 식이섬유는 76% 이상이 낮은 유의성(P-value >0.05)을 보였다. 이는 강낭콩(common bean)에서 조단백질, 조섬유, 식이섬유에 대한 비교적 낮은 유전력이 보고된 연구결과와 유사하였다(Bekele et al., 2012; Escribano et al., 1997; Jiang et al., 2018a; Jibril, 2019; Leleji et al., 1972). 그런데 또 다른 연구에서는 콩(soybean)의 조단백질, 조섬유의 유전력은 높은 것으로 보고되었는데(Jiang et al., 2018b), 성숙일수의 차이와 마찬가지로 형질의 유전력이 높다고 하더라도 엽록체 유전자에 대한 계통분석의 문제, 지리적 위치, 재배환경, 작물재배에 대한 농약 및 비료의 사용과 같은 인간활동 등에 의해 유전력이 차이가 있었을 것으로 보여진다. 특히 100립중은 선택압(selection pressure)과 상가적 유전자에 의한 유전적 영향이 높은 것으로 알려져 있는데(Chandrawat et al., 2017; Neelima et al., 2018; Rimlinger et al., 2020; Rosbakh, 2014), 아마도 다수의 유전자의 복잡한 과정에 의해 결정되는 형질은 추정이 더욱 어려울 것으로 판단된다.
Table 5.
No. (%) of genes and clusters at each P-value
고 찰
두류는 식량으로서 주요한 자원 중의 하나로 형태적, 유전적인 요인에 따라 나타나는 다양한 형질을 이용하여 장기적인 먹거리의 소재로 활용하고 있다. 그런데 형태적 형질은 유전적 혹은 환경적 요인에 따라 형질의 차이 혹은 변화할 수 있기 때문에 환경과 유전적 요인에 따른 영향을 추정하는 유전력은 식량자원으로서 장기적으로 안정적인 공급을 위한 식물육종 및 품종 개발, 형질 보존을 위한 중요한 지수로 활용되고 있다(Holland et al., 2003). 하지만 형질에 대한 유전력을 검정하기 위해서는 복잡한 세대증식의 설계와 재배시간, 농업노동력 공급 등으로 인하여 많은 시간과 비용이 소요되기 때문에 유전력을 추정할 수 있는 대안이 요구된다. 본 연구에서는 두류 6종에 대한 형태적 형질의 차이와 엽록체 유전자 정보의 계통분류적 차이를 상호 비교함으로써 형질에 대한 유전력을 대략적으로 추정할 수 있는 방법을 수행하였다. 그 결과 성숙일수와 100립중, 조단백질, 조섬유, 식이섬유를 제외하고 개화일수, 생육일수, 협당 립수, 조지방이 유전자의 계층적 차이와 높은 상관성을 보여 형질에 대한 유전력이 높을 것으로 추정하였다. 기 연구된 결과와 비교했을 때 성숙일수와 100립중이 불일치한 결과를 보였는데, 이는 엽록체 유전체 정보의 낮은 계통발생 신호, 재배환경 및 인간에 의한 영향과 함께 형질에 대한 유전자의 우점, 상위적 유전자 및 상가적 유전자 영향 등으로 인하여 일부 형질은 예측이 어려울 것으로 예상된다. 그러므로 이를 보완하기 위해서는 엽록체 유전체와 함께 계통분류에 많이 활용되고 있는 미토콘드리아 유전체 정보(Yue et al., 2023)를 함께 활용하여 계통발생 신호정보의 보완을 통해 계통정보의 신뢰를 높여야 할 것이다. 또한 단일염기다형성(single nucleotide polymorphism)과 같은 핵DNA에 대한 유전적 다양성 정보를 활용도 추가로 고려해야 할 것이다. 이미 GWAS (genome wide association study) 연구에서 유용하게 사용되고 있는 GAPIT은 SNP를 이용한 유전력 정보를 제공하고 있기 때문에 충분한 자원과 SNP 정보가 수집된다면 본 연구와 더불어 형질에 대한 신뢰높은 유전력을 추정할 수 있을 것이다(Lipka et al., 2012). 그래서 여러 환경에서의 교차재배를 통한 장기적인 형질 조사정보와의 비교, 검정으로 유전력 추정의 정확성과 신뢰도를 보완해 나간다면, 항 후 신품종 개발 및 우수자원 선발과 육종기술 등과 같이 농업유전자원의 보존, 관리, 재배, 증식 등 광범위한 농업분야의 발전에 크게 기여할 수 있을 것으로 기대한다.
적 요
두류는 식량으로서 중요한 자원 중의 하나로 유용한 형질을 이용하여 장기적인 먹거리의 소재로 개발 및 활용하고 있다. 그런데 형질은 작물의 다양성과 관련하여 유전적 혹은 환경적 요인에 따라 형질 변이가 발생하기 때문에 유용한 형질의 장기적인 활용을 위해서는 유전적으로 장기적인 유지가 가능한 유전력이 높은 형질을 선발하는 것이 중요하다. 그러나 유전력의 추정은 복잡한 세대증식의 설계와 재배시간, 농업노동력 공급 등에 필요한 많은 시간과 비용이 소요되기 때문에 이를 해결할 수 있는 대안이 요구된다. 본 연구에서는 두류 6종에 대한 형태적 형질과 엽록체의 총 455개 유전자의 계통 분류학적 차이를 통계적으로 상호 비교하여 형질에 대한 유전력을 추정하였다. 그 결과로 개화일수, 협당 립수, 조지방이 유의수준 0.05 이하(P-value ≤0.05)에서 상관관계가 있는 유전자가 각각 62.86%, 69.45%, 57.14%로 높은 상관성을 보였고, 생육일수는 유의수준 0.1 이하에서 62.42%로 상관성을 보임으로써 환경적 영향보다 유전적 영향에 의한 변이가 더 크게 작용할 것으로 예상된다. 반면 성숙일수, 100립중, 조단백질, 조섬유, 식이섬유 함량에 대한 변이는 유의수준 0.05이하에서 상관성이 있는 유전자가 평균 11.82%로 환경적 변화영향이 더 클 것으로 추정되었다. 이 가운데 성숙일수와 100립중은 기존의 연구와는 다른 결과를 보였는데 이는 재배환경, 인간활동(농약, 비료 사용 등)과 같은 환경적 영향 뿐만 아니라 유전력과 유전적 진전과의 관계에 따른 상위적, 상가적 유전자의 복잡한 영향으로 인하여 다른 연구결과가 나타났을 것으로 판단된다. 비록 엽록체 유전자 정보를 이용한 유전력 추정이 다소 미흡하지만 향 후에 단일염기다형성의 활용, 낮은 계통발생 신호의 보완을 위한 미토콘드리아 유전체 정보, 핵DNA 유전자 정보 등을 활용하고, 실제 조사결과와의 비교검정을 통한 보완이 이루어진다면 유전력 추정을 통한 신품종 개발 및 우수자원 선발과 육종기술 등과 같이 농업유전자원의 보존, 관리, 재배, 증식 등 광범위한 농업분야의 발전에 크게 기여할 수 있을 것이다.