서 언
재료 및 방법
샘플 수집
Genomic DNA추출
NGS 분석 및 InDel 구간 탐색
프라이머 제작
Polymerase chain reaction (PCR)
Genotyping
통계분석 및 군집분석
결 과
NGS 분석을 통한 InDel 구간 탐색
개발된 InDel 마커를 이용한 닥나무 속 식물의 genotyping
InDel 마커를 이용한 닥나무 속 식물들의 군집 분석
고 찰
적 요
서 언
국내에서 자생하고 있는 닥나무 속(Broussonetia)에는 닥나무(Broussonetia kazinoki Siebold), 애기닥나무(Broussonetia monoica Hance), 꾸지나무(Broussonetia papyrifera (L.) L'Hér. ex Vent.)가 있다. 이 중 닥나무는 애기닥나무와 꾸지나무의 중간 형태를 가지고 있어 잡종으로 인식되어 왔으며, 최근 연구에서도 닥나무는 유전적으로 애기닥나무와 꾸지나무의 자연 교잡종인 것으로 보고되었다(Kim et al., 1992; Yun and Kim, 2009; Ohba and Akiyama, 2014; Chung et al., 2017; Won, 2019; Kim et al., 2022a). 따라서 닥나무, 애기닥나무, 꾸지나무는 서로 형태적 특성이 유사하고, 속 내 교잡이 가능할 것으로 유추할 수 있다.
닥나무는 자웅이주이며, 잎의 길이와 폭은 각각 14~20 ㎝, 10~14 ㎝로 끝은 날카롭고 간혹 깊이 갈라진 것도 있으며, 가장자리에 날카로운 톱니가 있고, 줄기는 회갈색이다. 애기닥나무는 자웅동주이며, 잎의 길이와 폭은 각각 8~11 ㎝, 4~6 ㎝로 끝은 갈라짐이 있고, 가장자리에 톱니가 있다. 꾸지나무는 자웅이주이며, 잎의 길이와 폭은 각각 20~29 ㎝, 14~18 ㎝로 끝이 갈라지지 않거나 불규칙하게 3~5개로 갈라지며, 가장자리에는 톱니가 있고, 줄기는 암회색이다(Kim et al., 1992; Kwon, 2020). 이러한 특징에 따라 일반적으로 닥나무 속은 잎의 특징, 줄기의 색과 무늬 유무로 구분한다(Go et al., 2019). 그러나 닥나무와 꾸지나무의 형태적 특성이 유사할 뿐 아니라 자연교잡종의 존재로 인해 형태학적 형질에 따른 종의 식별은 오동정의 우려가 발생할 수 있다.
닥나무 속 식물들의 인피섬유는 오래 전부터 한지의 주된 원료로 사용되어왔다. 닥나무 속 식물의 섬유는 길고 질기며, 엉키는 성질이 있어 창호지, 우산지, 장판지 등을 만드는 데에도 많이 사용되었다(Kwon, 2020). 또한, 최근에는 닥 섬유와 전통한지가 일회용 속옷, 양말, 의상, 가방 등 여러 분야에서 활용되고 있다(Park, 2021). 그러나 국내 한지 산업에서 원료의 종은 명확하게 구분되고 있지 않다. 한지의 품질은 수종에 따른 섬유 특성의 영향을 많이 받기 때문에(Go and Jeong, 2018) 국내 한지의 균일한 품질관리를 위해서는 닥나무 속 식물들에 대한 명확한 분류체계를 확립할 필요가 있다.
Insertion or Deletion (InDel) 마커는 표준이 되는 유전체와 비교하여 염기서열의 삽입 또는 결실로 인한 차이를 이용하여 생물을 식별하는데 활용하는 분자마커를 말한다. 전체 genome에 분포하는 공우성 마커로 유의미한 다형성을 효율적으로 분석할 수 있다는 장점이 있다(Chen et al., 2021; Kim et al., 2022b). InDel 마커를 사용한 연구로는 국내 콩(Glycine max (L.) Merrill) 품종을 대상으로 한 연구와(Chun et al., 2019) 오미자과(Schisandraceae) 식물의 종 판별을 위해 엽록체 기반 InDel 마커를 개발한 연구가 있으며(Jeong et al., 2021), 국내 대추(Ziziphus jujuba var. inermis) 품종을 대상으로 국내에서 유통, 재배되는 대추의 품종을 구분하기 위하여 InDel 마커를 개발한 연구(Kim et al., 2021) 등이 있다.
식물체의 엽록체 유전체는 안정적인 구조를 가지고 있으며 핵 DNA보다 진화 속도가 느리고, 모계유전을 하는 특성 때문에 여러 종류의 분자마커가 개발되어 다양한 종의 판별 연구에 사용되고 있다(Wolfe et al., 1987; Pan et al., 2012; Park et al., 2017).
최근 닥나무 속 식물을 대상으로 한 연구로는 꾸지나무의 유전적 다양성에 관한 연구(Peñailillo et al., 2017), 지리적 기원(Chang et al., 2015), 성 판별 마커 개발 연구(Peñailillo et al., 2016) 등과 닥나무 속 식물들의 유연관계분석 연구사례(Won, 2019), 계통발생학적 분석(Pratiwi et al., 2021)이 있으며, 이들 연구에서 simple sequence repeat (SSR), internal transcribed spacer (ITS), intergenic spacer (IGS) 마커 등이 개발 및 활용되었으나, 닥나무 속 식물의 엽록체 기반 InDel 마커 개발은 이루어진 바 없다.
본 연구에서는 닥나무 속 식물에서 엽록체 유전체 서열을 토대로 InDel 마커를 개발하여 닥나무 속 유전자원의 유전적 특성 파악에 활용하고자 하였다.
재료 및 방법
샘플 수집
본 연구에서는 대한민국 전역(강원, 경기, 경북, 전남, 전북, 제주, 충북)에서 자생 및 재배되고 있는 닥나무 속 식물들을 수집하여 충북 청주시에 위치한 충북대학교 내 묘포장에 식재된 22개체를 사용하였다(Table 1). 분석을 위한 재료는 어린잎을 수집하여 –70℃의 초저온 냉동고에 보관하였다.
Table 1.
The twenty-two samples used in this study
Genomic DNA추출
Next-generation sequencing (NGS) 분석을 위한 genomic DNA (gDNA)는 형태적으로 차이가 있는 6개체의 잎 시료를 액체질소를 이용하여 급랭시킨 후, DNeasy Plant Mini Kit (Qiagen GmbH, Hilden, Germany)를 사용하여 제조사가 제시한 방법에 따라 추출하였다. PCR 분석을 위한 gDNA는 cetyltrimethylammonium bromide (CTAB)를 사용하여 Doyle and Doyle (1990)가 제시한 방법에 따라 추출하였다. 추출한 gDNA는 1% agarose gel에서 전기영동분석을 통해 확인한 후, DeNovix® DS-11+ spectrophotometer (DeNovix Inc., Wilmington, DE, USA) 기기를 사용하여 농도를 측정하고 최종농도가 5 ng/μL 가 되도록 멸균된 증류수를 이용하여 희석하였다.
NGS 분석 및 InDel 구간 탐색
NGS 분석을 위하여 Macrogen사(Seoul, Korea)에 의뢰하여 Illumina HiSeq 2500 platform (Illumina, San Diego, CA, USA)의 151 bp paired-end sequencing을 수행하였다.
닥나무 속 식물의 InDel 구간 탐색을 위하여 CLC Genomics Workbench (ver. 20.0, Qiagen, Aarhus, Denmark)를 이용하였다. NGS 분석을 통하여 얻은 reads는 trimming을 하여 sequencing data의 품질을 높인 뒤, contig 작성을 위해 de novo assembly 과정을 진행했다. 조립된 염기서열로부터 Basic Local Alignment Search Tool (BLAST) 분석을 통하여 엽록체 유전체를 선발한 뒤, 각 개체들의 contig들을 비교하여 InDel이 예상되는 구간을 탐색하였다.
프라이머 제작
프라이머는 CLC Main Workbench (ver. 20. Qiagen, Aarhus, Denmark)를 사용하여InDel 구간을 포함하는 정방향 프라이머와 역방향 프라이머를 각각 디자인하였다. 프라이머 길이는 16 bp~22 bp, G+C 함량은 40~60%, annealing 온도는 47~55℃, 프라이머 세트 사이의 annealing 온도 차이는 4℃ 이하, 그리고 증폭 산물 길이는 550 bp 이하로 하였다.
Polymerase chain reaction (PCR)
PCR 반응은 5 ng/μL 농도의 gDNA 2 μL, DSBio 사의 Taq mix (0.25 U/μL Taq DNA polymerase, 2 × PCR buffer, 0.4 mM dNTPs, 3.2 mM MgCl2, 0.02% bromophenol blue) 5 μL, 정방향 프라이머(5 μM) 0.3 μL, 역방향 프라이머(5 μM) 0.3 μL, 3차 증류수 2.4 μL로 총 용량 10 μL으로 하였고, T100™ Thermal Cycler (Bio-Rad Laboratories Inc. Hercules, CA, USA)를 이용하여 PCR 증폭을 수행하였다.
PCR 조건은 95℃에서 pre-denaturation 3분, 95℃에서 denaturation 30초, 44~51℃에서 annealing 30초, 72℃에서 extension 1분으로 총 35회 반복하였으며, 72℃에서 final extension 10분을 실시하였다. 최종 PCR 산물은 –20℃의 냉동고에 보관하였다.
Genotyping
본 연구에서는 PCR 증폭 확인 및 다형성 분석을 위하여 Fragment Analyzer (Agilent, Santa Clara, CA, USA)를 이용하였다. Fragment Analyzer 분석에는 Agilent 사의 protocol에 따라 PCR product, Dilution buffer, 930 dsDNA Inlet buffer, capillary conditioning solution, dsDNA 905 gel, 35~400 bp DNA ladder, 1 bp lower and 500 bp upper marker, intercalating dye가 사용되었다.
통계분석 및 군집분석
PowerMarker version 3.25 (Liu and Muse, 2005)를 이용하여 개발된 마커에 대한 major alleles frequency (MAF), genotype number (GN), allele number (AN), gene diversity (GD), heterozygosity (H), polymorphism information content (PIC)를 분석하였고, genotyping을 통해 산출된 allele 값은 PowerMarker를 이용하여 Unweighted Pair Group Method with Arithmetic mean (UPGMA) 방식으로 분기도 작성을 하였다.
결 과
NGS 분석을 통한 InDel 구간 탐색
수집된 자원 중 D-01, D-02, D-03, D-04, D-05, D-06번 개체를 NGS 기법을 이용하여 분석하였다(Table 2).
Table 2.
Summary of next-generation sequencing data using six Broussonetia plants
CLC Genomics Workbench 프로그램을 이용하여 NGS 데이터로부터 얻은 개체 별로 조립된 contig들 중 엽록체 서열을 비교하여 다형성이 보이는 InDel 구간을 탐색하였다. 크기가 8 bp 이상인 InDel 구간 중 마커 개발이 용이한 8개의 locus를 선정하였다. 후보 마커를 증폭하기 위해 8개의 InDel 구간에 대해 정방향 프라이머와 역방향 프라이머를 각각 디자인하였다.
제작된 프라이머가 정상적으로 증폭하는지 확인하기 위해 PCR 분석을 수행하였고, gel 전기영동을 이용하여 결과를 분석하였다. 그 결과 8개의 프라이머 세트 중 7개의 프라이머 세트에서 특이적인 증폭이 이루어졌다.
성공적으로 증폭한 7개의 InDel 구간이 in silico에서 발견한 InDel 구간과 실제 실험결과가 일치하는지 확인하기 위해 fragment analyzer를 이용하여 다형성을 분석하였다. 그 결과 5개의 프라이머 세트에서 다형성이 확인되었으며, 증폭 산물의 염기서열 분석을 통하여 증폭된 염기서열의 차이를 확인하였다. 최종적으로 2개의 유전자형을 나타내는 마커 4개와 3개의 유전자형을 나타내는 마커 1개로 총 5개의 마커를 개발하였다(Table 3).
Table 3.
Primer information for Broussonetia spp. InDel markers
개발된 InDel 마커를 이용한 닥나무 속 식물의 genotyping
개발된 마커들을 이용하여 22개의 닥나무 속 식물을 대상으로 genotyping 분석을 실시하였다. Fragment Analyzer를 이용한 genotyping 분석 결과, Br-cp-InDel-1 마커에서는 D-01, D-03, D-04, D-11, D-12, D-15, D-17, D-19이 270 bp 크기의 밴드를 형성하였고, D-02, D-05, D-06, D-07, D-08, D-09, D-10, D-13, D-14, D-16, D-18, D-20, D-21, D-22는 291 bp 크기의 밴드를 형성하였다.
Br-cp-InDel-2 마커에서는 D-01, D-03, D-04, D-11, D-12, D-15, D-17, D-19이 248 bp 크기의 밴드를 형성하였고 D-02, D-05, D-06, D-07, D-08, D-09, D-10, D-13, D-14, D-16, D-18, D-20, D-21, D-22는 264 bp 크기의 밴드를 형성하였다.
Br-cp-InDel-3 마커에서는 D-01, D-02, D-04, D-05, D-06, D-07, D-08, D-09, D-10, D-11, D-12, D-13, D-14, D-16, D-17, D-18, D-20, D-21, D-22이 236 bp 크기의 밴드를 형성하였고 D-03, D-15, D-19는 244 bp 크기의 밴드를 형성하였다.
Br-cp-InDel-5 마커에서는 D-11, D-12이 74 bp 크기의 밴드를 형성하였고 D-01, D-03, D-04, D-15, D-17, D-18, D-19는 94 bp 크기의 밴드를 형성하였다. 그리고 D-02, D-05, D-06, D-07, D-08, D-09, D-10, D-13, D-14, D-16, D-20, D-21, D-22는 115 bp 크기의 밴드를 형성하였다.
Br-cp-InDel-7 마커에서는 D-02, D-05, D-06, D-07, D-08, D-09, D-10, D-13, D-14, D-16, D-20, D-21, D-22이 243 bp 크기의 밴드를 형성하였고 D-01, D-03, D-04, D-11, D-12, D-15, D-17, D-18, D-19는 262 bp 크기의 밴드를 형성하였다(Fig. 1).
Br-cp-InDel-1, Br-cp-InDel-2, Br-cp-InDel-3, Br-cp-InDel-7은 2가지의 유전자형이 발견되었고, Br-cp-InDel-5는 3가지의 유전자형이 발견되었다.
본 연구에서 개발한 마커들의 통계학적 특성을 22개의 닥나무 속 식물을 분석한 결과를 토대로 분석하였다. MAF 값은 Br-cp-InDel-1에서 0.6364, Br-cp-InDel-2에서 0.6364, Br-cp-InDel-3에서 0.8636, Br-cp-InDel-5에서 0.5909, Br-cp-InDel-7에서 0.5909로 평균 0.6636의 값을 나타냈다. GN 값과 AN 값은 Br-cp-InDel-1, Br-cp-InDel-2, Br-cp-InDel-3, Br-cp-InDel-7은 2개, Br-cp-InDel-5는 3개를 나타내었다. GD는 Br-cp-InDel-1에서 0.4628, Br-cp-InDel-2에서 0.4628, Br-cp-InDel-3에서 0.2355, Br-cp-InDel-5에서 0.5413, Br-cp-InDel-7에서 0.4835로 평균 0.4372의 값을 나타냈다. H 값은 모두 0이었으며, PIC는 Br-cp-InDel-1에서 0.3557, Br-cp-InDel-2에서 0.3557, Br-cp-InDel-3에서 0.2078, Br-cp-InDel-5에서 0.4632, Br-cp-InDel-7에서 0.3666으로 평균 0.3498을 나타냈다(Table 4).
Table 4.
Diversity statistics of twenty-two Broussonetia genus genetic resources analyzed using five InDel markers
| Marker | MAFz | GNy | SSx | ANw | GDv | Hu | PICt |
| Br-cp-Indel-1 | 0.6364 | 2 | 22 | 2 | 0.4628 | 0 | 0.3557 |
| Br-cp-Indel-2 | 0.6364 | 2 | 22 | 2 | 0.4628 | 0 | 0.3557 |
| Br-cp-Indel-3 | 0.8636 | 2 | 22 | 2 | 0.2355 | 0 | 0.2078 |
| Br-cp-Indel-5 | 0.5909 | 3 | 22 | 3 | 0.5413 | 0 | 0.4632 |
| Br-cp-Indel-7 | 0.5909 | 2 | 22 | 2 | 0.4835 | 0 | 0.3666 |
InDel 마커를 이용한 닥나무 속 식물들의 군집 분석
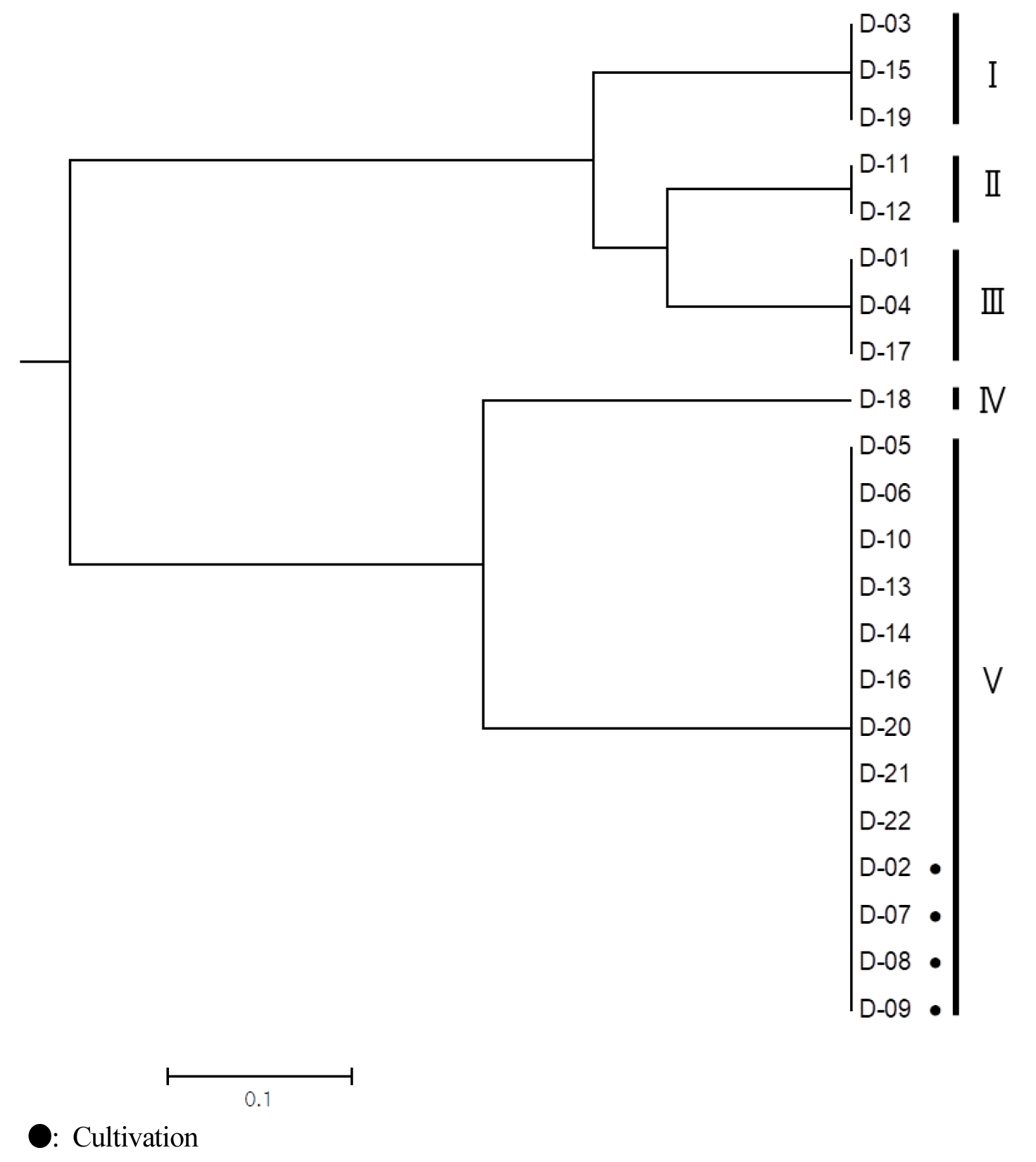
본 연구에서 개발된 마커들을 이용한 22개의 닥나무 속 식물의 genotyping 결과를 이용하여 군집분석을 수행한 결과, 총 5개의 그룹을 형성하였다(Fig. 2).
첫 번째 그룹(Group I)은 자웅이주이며, 잎의 길이가 15.7 ㎝, 잎자루의 길이가 5.9 ㎝인 D-03와 자웅동주이며, 잎의 길이가 15.4 ㎝, 잎자루의 길이가 15.5 ㎝인 D-15, 그리고 자웅동주이며, 잎의 길이가 13.4 ㎝, 잎자루의 길이가 1.4 ㎝인 D-19로 이루어졌다.
두 번째 그룹(Group II)은 자웅이주이며, 잎의 길이가 20 ㎝, 잎자루의 길이가 5.1 ㎝인 D-11, 자웅이주이며, 잎의 길이가 20.4 ㎝, 잎자루의 길이가 5.9 ㎝인 D-12로 이루어졌다.
세 번째 그룹(Group III)은 자웅이주이며, 잎의 길이가 15.0 ㎝, 잎자루의 길이가 1.1 ㎝이며 적색 수피를 가진 D-01과 자웅동주이며, 잎의 길이가 12.1 ㎝, 잎자루의 길이가 1.5 ㎝인 D-4, 그리고 자웅이주이며, 잎의 길이가 12.9 ㎝, 잎자루의 길이가 1.8 ㎝인 D-17로 이루어졌다.
네 번째 그룹(Group IV)에는 자웅동주이며, 잎의 길이가 11 ㎝, 잎자루의 길이가 0.9 ㎝인 D-18이 있었다.
가장 많은 개체들이 이룬 다섯 번째 그룹(Group V)은 자웅이주이며, 잎의 길이가 16.9 ㎝, 잎자루의 길이가 5.2 ㎝이며 회백색 무늬의 수피를 가진 D-02, 자웅이주이며, 잎의 길이가 15.6 ㎝, 잎자루의 길이가 5.6 ㎝이며 회백색 무늬의 수피를 가진 D-05, 자웅이주이며, 잎의 길이가 20.3 ㎝, 잎자루의 길이가 5.3 ㎝이며 녹색 무늬의 수피를 가진 D-06, 자웅이주이며, 잎의 길이가 18.6 ㎝, 잎자루의 길이가 8.0 ㎝이며 회백색 무늬의 수피를 가진 D-07, 자웅이주이며, 잎의 길이가 11.5 ㎝, 잎자루의 길이가 2.3 ㎝이고 녹색 무늬의 수피를 가진 D-08, 자웅이주이며, 잎의 길이가 14 ㎝, 잎자루의 길이가 8.0 ㎝이며 적색 수피를 가진 D-09, 자웅이주이며, 잎의 길이가 14.5 ㎝, 잎자루의 길이가 3.9 ㎝인 D-10, 자웅동주이며, 잎의 길이가 13.8 ㎝, 잎자루의 길이가 1.4 ㎝인 D-13, 자웅동주이며, 잎의 길이가 13.9 ㎝, 잎자루의 길이가 1.1 ㎝인 D-14, 자웅이주이며, 잎의 길이가 20.2 ㎝, 잎자루의 길이가 5.0 ㎝인 D-16, 자웅이주이며, 잎의 길이가 12.6 ㎝, 잎자루의 길이가 4.5 ㎝인 D-20, 자웅동주이며 잎의 길이가 13.5 ㎝, 잎자루의 길이가 1.1 ㎝인 D-21, 그리고 자웅이주이며, 잎의 길이가 20.3 ㎝, 잎자루의 길이가 5.4 ㎝인 D-22로 이루어졌다.
고 찰
최근에 발표된 연구 결과를 토대로(Won, 2019) 닥나무 속은 애기닥나무와 닥나무 계열이 모계가 같아 한 그룹을 이루고, 꾸지나무 계열이 한 그룹을 이루어 2 개의 그룹을 이룰 것으로 예상하였으나 본 연구에서 분석한 자원들이 예상보다 많은 5 개의 그룹으로 나뉘었다. 그리고 농가에서 재배되고 있는 자원들인 D-02, D-07, D-08, D-09는 그 중 다섯 번째 그룹에 포함되었다. 이러한 결과는 우리나라에 자생하는 닥나무 속 식물의 유전적 조성이 기존의 생각보다 다양하다는 것을 보여준다.
D-18 개체만으로 구성된 Ⅳ 번 그룹을 제외한 4 개 그룹의 자생 자원들의 형태적 특성을 보았을 때, II 번 그룹은 형태적 특성이 유사성을 보였다. 그러나 Ⅰ, III, V 번 그룹에서는 공통적인 특성을 찾기 어려웠다. 이는 닥나무 속 식물들은 속 내 교잡이 쉽게 일어나기 때문에 엽록체를 기준으로 나뉜 그룹에서는 하나의 그룹에서 다양한 형태적 특성이 나타날 수 있기 때문인 것으로 보인다(Fig. 2). 엽록체 DNA는 모계유전을 하기 때문에 엽록체 기반 마커에서는 잡종 개체가 모계의 종과 같이 구분될 수 있기 때문에 유전학적 분석에 한계가 발생할 수 있다(Kim et al., 2022b). 따라서 보다 뚜렷한 구분을 위해서는 염색체 기반 마커의 추가적인 개발이 필요할 것으로 보인다.
본 연구에서 닥나무 속 식물 엽록체 유전체 기반의 InDel 마커들을 개발함으로써 닥나무 속 식물들의 유전적 다양성 분석에 활용할 수 있었다. 최근 발표된 매실과 살구의 잡종 판별에 관련된 연구에서(Kim et al., 2022b) 엽록체 유전체 기반의 InDel 마커와 염색체 유전체 기반의 InDel 마커를 함께 분석하였을 때, 잡종의 유무와 함께 잡종을 이룬 모계를 구분해낼 수 있음을 알 수 있다. 따라서 본 연구에서 개발된 엽록체 유전체 기반의 마커와 더불어 염색체 유전체 기반의 마커를 추가적으로 개발하여 함께 분석한다면 국내에 자생하고 있는 닥나무 속 식물들의 종간의 유연관계를 보다 자세하게 알 수 있고, 닥나무 속 식물의 육종이나 종의 판별에 활용할 수 있을 것이라 기대한다.
적 요
본 연구에서는 닥나무 속 식물에 대한 InDel 마커를 개발하였다. 전국의 닥나무 속 식물 22개체를 수집하였고, 수집한 닥나무 속 식물 중 6개체를 차세대염기서열 분석(NGS)을 실시하였다. NGS를 통하여 얻은 염기서열 정보를 기존에 발표되었던 닥나무 엽록체 서열과 비교하여 InDel 마커 후보를 선발하였다. 선발한 마커 후보를 수집된 닥나무 속 식물에 적용하여 마커의 특성 검정을 통해 총 5개의 엽록체 기반 마커를 개발하였다. 개발된 InDel 마커를 22개의 유전자원에 적용한 후 군집 분석을 실시한 결과, 총 5개의 그룹으로 나뉘었다. 본 연구에서 개발된 마커들은 닥나무 속의 육종이나 종 판별에 활용할 수 있을 것이라 판단된다.