서 언
재료 및 방법
식물재료
기내배양과 기관분화 단계별 재분화체 작성
기관분화 관련 유전자 Reverse transcriptase-polymerase chain reaction (RT-PCR)
Genomic DNA 추출 및 ISSR 분석
Data 분석
결과 및 고찰
기내 기관분화 단계별 재분화체의 기관분화 관련 유전자의 발현
ISSR분석에 따른 기관분화 단계별 및 재분화체의 DNA 밴드 양상
기관분화 단계별 재분화체의 유전적 유사도 지수
기관분화 단계별 재분화체의 군집분석
적 요
서 언
기내배양을 통한 식물체 재생과정에서는 기관분화에 관련된 유전자의 발현 등 분자생물학적 과정을 통하여 기능이 작동함으로써 식물체가 재발생된다(Hans et al., 2014). 식물의 기내배양에 따른 기관분화는 직접 재분화와 탈분화를 거쳐 재분화에 이르는 경로가 알려져 있다. 직접 재분화는 치상조직으로부터 지상부가 출현하고 지하부가 출현하는 과정을 통해 개체가 완성된다. 또한 탈분화세포인 캘러스를 통한 경로에는 식물체가 발생하는 체세포배발생 경로와 캘러스에서 지상부(신초)와 지하부(부정근)가 생성되는 경로로 알려져 있다(Cho and Byeon, 2011; Lee et al., 2023c; Murashige and Skoog, 1962; Rebouillat et al., 2009; Seo, 2018). 이처럼 일련의 기관분화의 단계에 따라서 기관분화와 관련된 유전자가 활성화되어 기관분화 관련 유전자가 작동하므로 이 과정에서의 관련 유전자 발현에 대한 동태파악으로 기관분화에 대한 이해를 높일 수 있다. 한편 기내 재생식물체의 경우 생장조절물질처리 및 기내 특수환경에 기인한 체세포영양계변이 등 변이가 보고되어 있으므로(Bednarek and Renata, 2020; Fatiha et al., 2019; Ferreira et al., 2023; Goh et al., 2011; Jin et al., 2008; Kang et al., 2021; Lee et al., 2023b; Mo et al., 2009; Ramakrishnan et al., 2014) 각 단계별 재분화체의 유전적 안정성에 대한 검토를 통하여 기내 대량식물체 증식에 활용하거나 식물의 유전적 개량(Ferreira et al., 2023)에 이용된다.
기관분화와 관련된 다양한 식물종 및 유전자에 대한 자료가 보고되고 있다. 탈분화 관련 유전자로 애기장대 캘러스 발달(Bouyer et al., 2011; Iwase et al., 2013a), 뿌리 특이 발현 유전자로 단자엽식물(Rebouillat et al., 2009; Wu et al., 2014) 및 애기장대(Liu et al., 2018) 뿌리 발달, 신초 특이 발현 유전자로 애기장대(Lopes et al., 2021), 체세포배발생 관련 유전자로 LEC1, BBM1 (Lewis et al., 2010; Nic-Can et al., 2013), 식물종 별로는 유채(Ouakfaoui et al., 2010), 애기장대(Lewis et al., 2010)등에서 다양한 기관분화 관련 유전자들의 기능이 보고되고 있다. 또한 Gallois et al. (2004)과 Lopes et al. (2021)은 신초형성과 체세포배발생에 공동 관여하는 유전자임을 보고하고 있다.
기내배양을 통한 기내식물체 대량번식시 안정적인 개체 생산이 요건이므로 기내배양에 따른 재분화 식물체의 유전적 변이와 안정성 관련 연구(Dewir et al., 2018; Garcia et al., 2019)는 화본과 식물에서도 ISSR 분석 등 다양한 방법(Goh et al., 2011; Jin et al., 2008; Kang et al., 2021; Lee et al., 2023b; Ramakrishnan et al., 2014)으로 진행되고 있다.
화본과 띠(Imperata cylindrica L.) 속의 원산지는 북아메리카, 아시아, 아프리카의 온대 및 열대 지방이며(Doopedio, 2008; Holm et al., 1977), 띠 속의 다년생 식물인 홍띠(Imperata cylindrica 'Rubra')는 잎의 상단부가 붉은색을 띤 관상 가치가 있는 조경용 식물자원으로 기내배양을 통한 대량생산으로 효율적인 활용이 가능하다(Kang et al., 2021). 본 연구는 홍띠(Imperata cylindrica ‘Rubra’)의 기내배양시 식물체 재생과정에서 기관분화 단계에 따른 재분화체의 기관분화 관련 유전자의 발현과 단계별 재분화체의 유전적 안정성에 대한 자료를 제공할 목적으로 기관분화 단계별 재분화체를 작성하여 기관분화 관련 유전자 발현과 ISSR 표지를 이용한 재생 단계별 재분화체의 유전적 안정성을 분석하였다.
재료 및 방법
식물재료
공시재료는 재배식물로 개발된 홍띠(Imperata cylindrica ‘Rubra’)로 ‘하늘정원야생화(대표 이동국)’에서 분양받았다. 기관분화 단계별 기관분화 관련 유전자 분석 시료는 5가지로 각 시료당 3개체씩 총 15개체를 공시하였다. 즉 대조구 모식물체 3개체, 캘러스 3개체, 뿌리가 발생한 캘러스 3개체, 신초가 발생한 캘러스 3개체, 재분화식물체 3개체를 공시하였다(Fig. 1, Lee et al., 2023a). 또한, ISSR 분석용 시료로는 대조구 모식물체(mother plant) 3개체, 캘러스 3개체, 녹색 신초가 발생한 캘러스 3개체, 적색 신초가 발생한 캘러스 3개체, 기내배양으로 재분화된 홍띠(적색) 식물체 3개체, 실내포트에서 재배시킨 재분화 녹색 식물체 3개체, 1년간 노지에서 재배시킨 재분화 녹색 식물체 3개체로 7종류, 총 21개체를 공시하였다(Fig. 2).

Fig. 1.
Fifteen individual plant-materials used in reverse transcriptase-polymerase chain reaction (RT-PCR) of organogenesis-related genes in Imperata cylindrica ‘Rubra’. zA: Mother plants; B: Calli; C: Calli with adventitious roots; D: Calli with shoots; E: Regenerants with green. *Bars represent 10 ㎜, respectively.

Fig. 2.
Twenty one individual plant-materials used in ISSR analysis of Imperata cylindrica ‘Rubra’. zA: Mother plants (control); B: Green-regenerants cultivated at culture room; C: Green-regenerants cultivated at field condition; D: In vitro regenerants with red shoots; E: Calli with green shoots; F: Calli with red shoots; G: Calli only. *Bars represent 10 ㎜, respectively.
기내배양과 기관분화 단계별 재분화체 작성
홍띠의 기부생장점 부위를 포함한 식물체를 70% 에탄올에서 30초간, 2% sodium hypochlorite에서 15분, 멸균증류수에서 1분간 침지한 다음 3회 수세 후 멸균된 여과지로 수분을 제거하여 표면 살균하였다. 기본배지로 MS 배지(Murashige and Skoog, 1962)를 사용하였고, pH는 멸균 전 5.80 ± 0.05로 적정하여 121℃, 1.2기압 하에서 15분간 멸균하였다. 재분화 녹색 식물체 재생은 Umami et al. (2012)의 띠(congongrass)의 기내 배양사례를 일부 수정하여 MS 배지 + 0.1 ㎎/L 2,4-D + 2.0 ㎎/L BA 첨가 배지에 생장점을 포함한 부위를 치상하여 캘러스를 유도하고, 캘러스 증식은 0.1 ㎎/L 2,4-D를 포함한 MS배지에서, 신초 재분화는 0.05 ㎎/L 2,4-D + 0.1 ㎎/L BA를 포함한 MS배지에서, 뿌리 재분화는 재분화된 신초를 MS에 이식하여 부정근을 유도하였다. 이들 기관분화 각 단계별 시료는 분석용 시료로 사용하였다. 신초 분화후 뿌리를 유도하고 재생된 유식물체는 인공상토에 이식하여 단계별로 순화, 활착시켰다(Kang et al., 2021). 활착된 식물체의 일부는 노지에 이식하여 2년간 재배하여 식물재료로 사용하였다. 배양은 26 ± 2℃, 25 μ㏖/s/㎡, 14h/10h (day/night)의 조건에서 실시하였다.
기관분화 관련 유전자 Reverse transcriptase-polymerase chain reaction (RT-PCR)
기관분화 관련 유전자는 NCBI (National Center for Biotechnology Information; NCBI)에서 탐색한 후 Macrogen사(Korea)에 의뢰하여 primer를 합성하였다.
RNA 추출: RNA를 추출하기 위하여 식물체 시료를 4℃에서 유지한 1 × PBS로 2회 세척한 후 RNeasy Mini Kit (QIAGEN GmbH., Hilden, Germany)를 이용하여 total RNA를 추출하고, DNaseⅠ (ThermoFisher)을 처리하였다.
cDNA 합성 및 확인: 추출한 1 ㎍의 total RNA를 사용하여 SuperScriptTM Ⅳ First-Strand Synthesis System (Thermo Fishrer) 매뉴얼에 따라 cDNA를 합성하였다. 추출한 cDNA는 Nanodrop Spectrophotometer (Thermo Fisher Scientific, Netherlands)로 농도를 측정하여 50 ng/μL로 희석하였고, 1.5%의 agarose gel에서 전기영동하여 밴드를 확인하였다.
RT-PCR 및 증폭산물 확인: PCR은 PCR Master Mix Kit (Promega Co., Madison, WI, USA)를 이용하여 수행하였고, 사용된 primer는 Table 1에서와 같이 기관분화 관련 유전자를 디자인하여 사용하였다. cDNA 50 ng/μL, 2X PCR Premix (LugenTM), 10 pmol 프라이머로 총 20 μL되게 혼합하여 사용하였다. RAP2-4 (WID1), FIE, BBM1, SERK1 4개 프라이머에 대한 PCR 반응은 Gene Amp 2700 (Applied Biosystem)을 사용하여 94℃에서 5분간 초기 변성시킨 후 94℃에서 20초간 denaturation, 56℃에서 30초간 annealing, 72℃에서 60초 extension 과정을 40회 수행한 후 최종적으로 72℃에서 7분간 반응시킨 후 종료하였다. OsSCR, WOX11, WUS, LEC1B 4개의 프라이머의 경우 annealing 온도 60℃를 제외한 나머지 조건은 앞의 PCR 반응 조건과 같게 진행하였다. RT-PCR이 완료된 후 증폭산물 15 μL를 RedSafe Nucleic Acid Staining Solution 20,000X (Intron Biotechnology, Inc.)를 첨가한 1.2% agarose gel에 loading하여 전기영동(50V, 60분)한 다음 Digital GelDoc System (GDS200D, Korea)을 사용하여 주요 밴드를 확인하였다.
Table 1.
Sequences of oligonucleotide primers used for RT-PCR analysis of regeneration and dedifferentiation related genes in the Imperata cylindrica ‘Rubra’
Genomic DNA 추출 및 ISSR 분석
gDNA는 식물체의 잎 부위를 CTAB 방법(Fang et al., 1992)에 따라 추출하여 0.8%의 agarose gel에서 전기영동하여 밴드를 확인하고, Nanodrop Spectrophotometer (Thermo Fisher Scientific, Netherlands)로 농도를 측정하여 최종농도는 50 ng/μL로 희석하였다.
ISSR 분석은 총18개의 ISSR 프라이머(UBC primer Set No. 9, University of British Columbia, Canada)로 예비실험 후 밴드가 명확한 14개의 프라이머를 선발하여 ISSR 분석에 이용하였다(Table 1). PCR 반응액은 주형 DNA 50 ng/μL, 2X PCR Premix (LugenTM), 10 pmol 프라이머로 총 20 μL로 혼합하여 사용하였다. PCR 반응은 Gene Amp 2700 (Applied Biosystem)을 사용하여 94℃에서 5분간 초기 변성시킨 후 94℃에서 45초간 denaturation, 52℃에서 45초간 annealing, 72℃에서 90초간 extension 과정을 40회 수행한 후 최종적으로 72℃에서 7분간 반응시킨 후 종료하였다. PCR이 완료된 후의 증폭산물 15 μL를 RedSafe Nucleic Acid Staining Solution 20,000X (Intron Biotechnology, Inc.)를 첨가한 1.2% agarose gel에 loading하여 전기영동(50V, 60분)한 다음 GelDoc (GDS200D, Korea)을 사용하여 주요 밴드를 확인하였다(Lee et al., 2023b).
Data 분석
ISSR로 분석된 DNA 밴드 양상은 이진수를 이용하여 밴드 유·무에 따라 1과 0으로 data를 표시하여 Jaccard 계수 (Jaccard coefficient)로 유전적 유사도지수를 산출하고 평균연결법(Average linkage method)으로 군집분석을 하였다. 모든 통계처리는 SPSS V.12 (SPSS Inc., USA)를 이용하여 실시하였다.
결과 및 고찰
기내 기관분화 단계별 재분화체의 기관분화 관련 유전자의 발현
공시한 15개체에 대하여 기관분화 관련 유전자(Table 1)의 발현을 확인한 결과는 Fig. 3과 같다. 캘러스 발생 유전자인 FIE는 모식물체 1번을 제외한 14개의 식물체에서 모두 관찰되었으며, 100~300 bp에 존재하였다. 또한, 뿌리 발생 관련 유전자인 WOX11은 15개의 모든 식물체에서 발현되었으며, 600~700 bp에 존재하였다. 신초 재분화 및 체세포 발생 관련 유전자인 WUS에서도 15개의 식물체에서 모두 발현하였으며, 각 처리구별로 비슷한 양상의 밴드를 형성하는 것을 확인하였다. 체세포 발생 유전자인 LEC1B는 15개 식물체에서 모두 발현하였고, 각 처리구별 비슷한 양상으로 비교적 약하게 밴드가 형성되었고, 100~300 bp에 존재하였다.
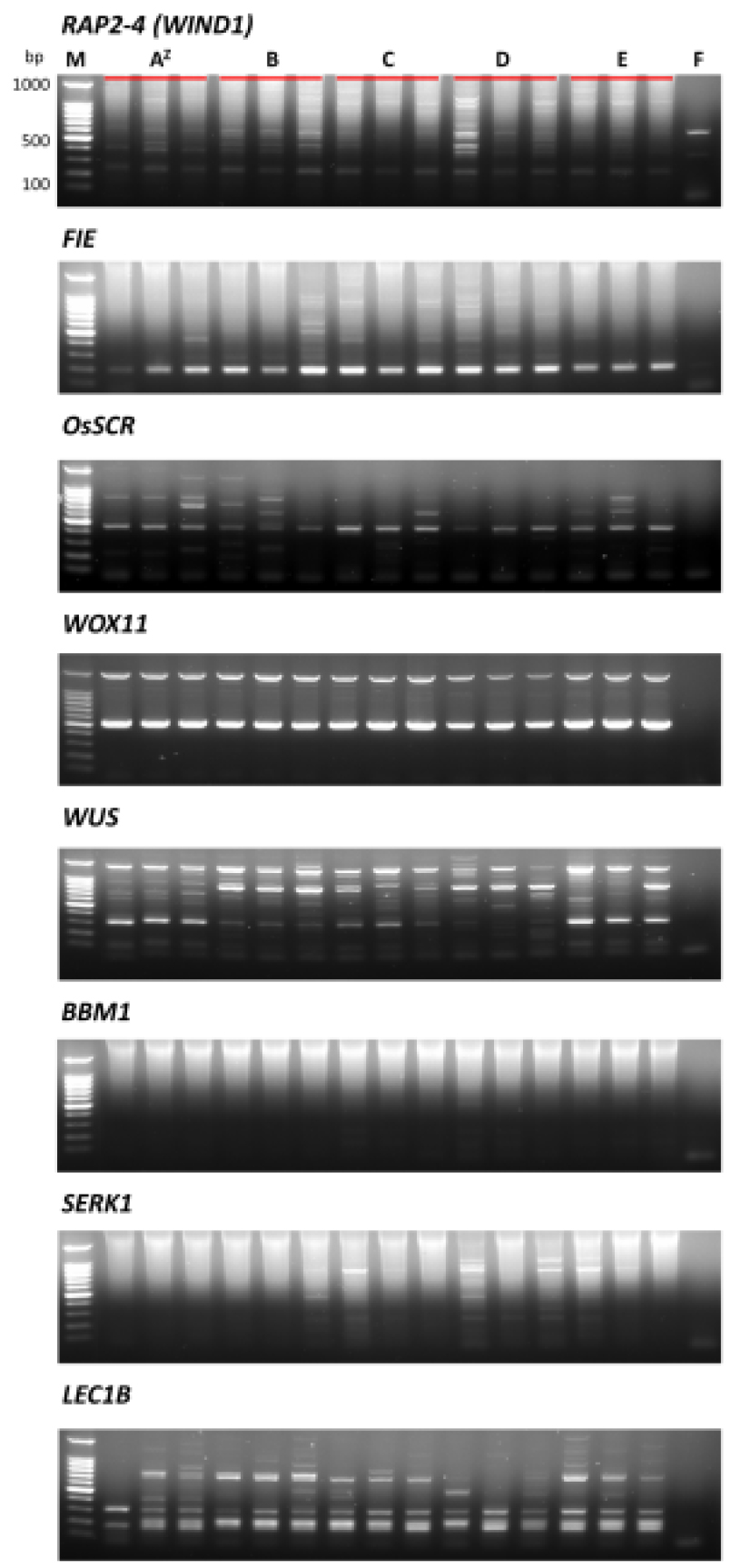
Fig. 3.
Electrophoresis profiles of reverse transcriptase- polymerase chain reaction (RT-PCR) products produced by organogenesis-related genes in plan-materials of the Imperata cylindrica ‘Rubra’. Sequences of 8 oligonucleotide primers were shown in Table 1 and plant-materials were in Fig. 1 (zLane M: 100 DNA marker, Lanes A: Mother plants; B: Calli; C: Calli with adventitious roots; D: Calli with shoots; E: Regenerants with green; F: Negative control).
기관분화와 관련된 유전자와 관련하여 체세포배발생 관련 유전자의 발현은 BBM1, SERK1, LEC1B, MEA 유전자에서 보고되었고(Hu et al., 2005; Lee et al., 2003; Lewis et al., 2010; Luo et al., 2000; Nic-Can et al., 2013; Ouakfaoui et al., 2010), SERK (Lewis et al., 2010; Nic-Can et al., 2013), WUS (Gallois et al., 2004; Nic-Can et al., 2013)는 신초분화에도 관여하는 것으로 보고되었다. 탈분화 세포인 캘러스에서는 FIE, RAP2-4 (WIND1) 유전자가 관여한 것으로 보고되었으며(Iwase et al., 2013b), WUS (Gallois et al., 2004; Hans et al., 2014)는 재생식물체와 캘러스+신초유도체, OsSCR (Kamiya et al., 2003; Rebouillat et al., 2009), WOX11 (Cheng et al., 2018; Zhao et al., 2009)은 캘러스+부정근체에서 보고되고 있다. 그러나 본 연구에서는 탈분화, 신초분화 및 부정근 재분화와 관련하여 기관분화 특이적으로 발현하지 않고 단계별 재분화체 전체에 대하여 발현하였으므로 향후 각 기관분화 관련 유전자별 실시간 발현량에 대한 검토를 통하여 관련 기관분화 관련 유전자 발현에 대한 상세한 동태 파악이 필요할 것으로 판단된다. 한편 Zhao et al. (2009)은 WUS와 WOX 11의 homeobox 유전자로서 기능을 보고하고 있어서 생체내에서 부정근/신초 재분화시 유전자 기능이 연결되어 작동함을 보고하고 있다.
ISSR분석에 따른 기관분화 단계별 및 재분화체의 DNA 밴드 양상
재분화 녹색 식물체를 실내에서 재배한 3개체와 녹색 재분화 식물체를 포장에서 재배한 3개체, 기내배양으로 재분화된 홍띠(적색) 식물체 3개체, 녹색 신초가 발생한 캘러스 3개체, 적색 신초가 발생한 캘러스 3개체, 캘러스 3개체, 그리고 대조구로 모식물체 3개체, 총 21개체를 시료로 하여 재현성이 높은 14개의 ISSR 프라이머를 선발하여 분석하였다(Table 2, Fig. 4).
ISSR 분석 결과, 대조구 3개체에서 밴드 수는 최저 3개(UBC 824, UBC 853, UBC 866)에서 최고 7개(UBC 810, UBC 843, UBC 847)로 한 프라이머 조합당 평균 밴드 수는 5.14개였다. 1개 프라이머(UBC 854)에서 1개의 다형성 밴드로 프라이머당 0.07개가 검출되었다(Table 2, Fig. 4).
실내재배 재분화 녹색 식믈체에서 밴드 수는 최저 3개(UBC 824, UBC853, UBC 866)에서 최고 8개(UBC 843)로 한 프라이머 조합당 5.28개였으며, 다형성 밴드 수는 최저 0개(UBC 810, UBC 813, UBC 815, UBC 824, UBC 826, UBC 847, UBC 848, UBC 853, UBC 857, UBC 862, UBC 866, UBC 889)에서 최고 2개(UBC 843)였다. 2개 프라이머(UBC 843, UBC 854)에서 3개의 다형성 밴드로 프라이머당 0.21개가 검출되었다(Table 2, Fig. 4).
노지재배 재분화 녹색 식물체에서 밴드 수는 최저 2개(UBC 824)에서 최고 7개(UBC 843, UBC 847)로 한 프라이머 조합당 4.93개였으며, 다형성 밴드 수는 최저 0개(UBC 810, UBC 813, UBC 826, UBC 843, UBC 848, UBC 853, UBC 854, UBC 857, UBC 862, UBC 866, UBC 889)에서 최고 1개(UBC 815, UBC 824, UBC 847)였다. 3개 프라이머(UBC 815, UBC 824, UBC 847)에서 3개의 다형성 밴드로 프라이머당 0.21개가 검출되었다(Table 2, Fig. 4).
기내배양 홍띠(적색) 식물체에서 밴드 수는 최저 2개(UBC 824)에서 최고 8개(UBC 847)로 한 프라이머 조합당 5.07개였으며, 다형성 밴드 수는 최저 0개(UBC 810, UBC 813, UBC 815, UBC 824, UBC 826, UBC 843, UBC 847, UBC 848, UBC 854, UBC 857, UBC 866, UBC 889)에서 최고 2개(UBC 862)였다. 2개 프라이머(UBC 853, UBC 862)에서 3개의 다형성 밴드로 프라이머당 0.21개가 검출되었다(Table 2, Fig. 4).
녹색 신초가 발생한 캘러스에서는 밴드 수는 최저 2개(UBC 824, UBC 853)에서 최고 8개(UBC 810, UBC 857)로 한 프라이머 조합당 5.14개였으며, 다형성 밴드 수는 최저 0개(UBC 810, UBC 813, UBC 824, UBC 826, UBC 843, UBC 847, UBC 848, UBC 853, UBC 854, UBC 862, UBC 866)에서 최고 2개(UBC 857)였다. 3개 프라이머(UBC 815, UBC 857, UBC 889)에서 4개의 다형성 밴드로 프라이머당 0.29개가 검출되었다(Table 2, Fig. 4).
적색 신초가 발생한 캘러스에서는 밴드 수는 최저 2개(UBC 824, UBC 853)에서 최고 8개(UBC 810, UBC 847)로 한 프라이머 조합당 5.00개였으며, 다형성 밴드는 검출되지 않았다(Table 2, Fig. 4).
캘러스에서는 밴드 수는 최저 2개(UBC 824, UBC 853)에서 최고 8개(UBC 847)로 한 프라이머 조합당 4.93개였으며, 다형성 밴드 수는 최저 0개(UBC 810, UBC 813, UBC 815, UBC 824, UBC 843, UBC 847, UBC 848, UBC 853, UBC 854, UBC 857, UBC 862, UBC 866, UBC 889)에서 최고 1개(UBC 826)였다. 1개 프라이머(UBC 826)에서 1개의 다형성 밴드로 프라이머당 0.07개가 검출되었다(Table 2, Fig. 4).
이상과 같이 유전적 다형성 비율은 재분화 발생 부위 별 재분화체 및 순화 재분화체[실내포트 재배식물체 4.1%, 노지 재배식물체 4.3%, 기내배양 홍띠(적색)식물체 4.2%, 녹색신초 발생 캘러스 5.6%, 캘러스 1.4%]에서 대조구인 모식물체 (1.4%)보다 같거나 높게 나타났으며, 적색신초 발생 캘러스는 0.0%로 대조구보다 낮게 나타났다.
이러한 결과는 옥수수(Ramakrishnan et al., 2014), 바나나 재분화체(Ray et al., 2006), 3.1%~4.1%의 홍띠 재분화체(Kang et al., 2021)에서와 유사한 경향을 보였다. 재분화체에서 모식물체보다 변이성이 높은 이유로는 생장조절물질 처리(Garcia et al., 2019)나 노지와는 다른 기내환경(Bednarek and Renata, 2020; Goh et al., 2011; Ramakrishnan et al., 2014) 등에 기인한 것으로 사료된다. 한편 종자유래 갈대의 재분화체 17% (Ryu et al., 2013)보다는 낮은 결과로서 종자의 다양한 유전적 배경 때문으로 사료된다.
Table 2.
Sequences of the 14 ISSR primers, numbers of total bands and polymorphic bands amplified by each primers across all 21 individuals of Imperata cylindrica ‘Rubra’
| Primer | Sequencez | No. of total bands | No. of polymorphic bands | Polymorphism rate (%) | ||||||||||||||||||
| Conty | ReR | ReF | ReRP | CG | CR | C | Cont | ReR | ReF | ReRP | CG | CR | C | Cont | ReR | ReF | ReRP | CG | CR | C | ||
| UBC 810 | (GA)8T | 7 | 7 | 6 | 6 | 8 | 8 | 7 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 813 | (CT)8T | 5 | 6 | 5 | 5 | 5 | 6 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 815 | (CT)8G | 4 | 4 | 4 | 3 | 3 | 3 | 3 | 0 | 0 | 1 | 0 | 1 | 0 | 0 | 0.0 | 0.0 | 25.0 | 0.0 | 33.3 | 0.0 | 0.0 |
| UBC 824 | (TC)8G | 3 | 3 | 2 | 2 | 2 | 2 | 2 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 50.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 826 | (AC)8C | 4 | 4 | 4 | 4 | 4 | 3 | 4 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 25.0 |
| UBC 843 | (CT)8RA | 7 | 8 | 7 | 7 | 5 | 5 | 5 | 0 | 2 | 0 | 0 | 0 | 0 | 0 | 0.0 | 25.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 847 | (CA)8RC | 7 | 7 | 7 | 8 | 7 | 8 | 8 | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 14.3 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 848 | (CA)8RG | 6 | 6 | 6 | 6 | 6 | 6 | 6 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 853 | (CT)8RT | 3 | 3 | 3 | 3 | 2 | 2 | 2 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 33.3 | 0.0 | 0.0 | 0.0 |
| UBC 854 | (CT)8RG | 6 | 6 | 6 | 5 | 5 | 5 | 5 | 1 | 1 | 0 | 0 | 0 | 0 | 0 | 16.7 | 16.7 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 857 | (AC)8YG | 5 | 5 | 5 | 6 | 8 | 6 | 6 | 0 | 0 | 0 | 0 | 2 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 25.0 | 0.0 | 0.0 |
| UBC 862 | (AGC)6 | 6 | 6 | 6 | 7 | 7 | 6 | 6 | 0 | 0 | 0 | 2 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 28.6 | 0.0 | 0.0 | 0.0 |
| UBC 866 | (CTC)6 | 3 | 3 | 3 | 4 | 5 | 5 | 5 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| UBC 889 | DBD(AC)7 | 6 | 6 | 5 | 5 | 5 | 5 | 5 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0.0 | 0.0 | 0.0 | 0.0 | 20.0 | 0.0 | 0.0 |
| Total | 72 | 74 | 69 | 71 | 72 | 70 | 69 | 1 | 3 | 3 | 3 | 4 | 0 | 1 | 1.4 | 4.1 | 4.3 | 4.2 | 5.6 | 0.0 | 1.4 | |

Fig. 4.
ISSR electrophoresis profiles of 21 individual accessions of Imperata cylindrica ‘Rubra’. Amplification products were separated on 1.2% agarose gels in 1×TAE buffer and photographed using gel documentation system (Bio-rad, USA). Lane M: 1kb ladder marker, Lanes A: Mother plants, B: Regenerants cultivated at culture room, C: Regenerants cultivated at field condition, D: In vitro regenerants with red plants, E: Calli with green shoots, F: Calli with red shoots, G: Calli. All the primer names were Included in Table 1.
기관분화 단계별 재분화체의 유전적 유사도 지수
총 21개체 간의 유전적 유사도 지수는 최소 0.747(5번과 13번/실내재배 재분화 녹색 식물체 2번과 녹색신초 발생 캘러스 1번)에서 최대 1.000 (1번과 2번, 7번과 9번 외) 사이로 21개체간 유전적 유사도 지수의 평균은 0.868이었다. 모식물체 간 유전적 유사도 지수는 0.991, 실내재배 재분화 녹색 식물체 간 0.973, 노지재배 재분화 녹색 식물체 간 0.981, 기내배양 홍띠 식물체 간 0.972, 녹색 신초 발생 캘러스 간 0.972, 적색 신초 발생 캘러스 간 1.000, 캘러스 간 0.990으로 나타났다(Table 3).
이는 다양한 단계별 재분화체에서 얻은 것으로 Kang et al. (2021)이 보고한 같은 식물에서 유래한 녹색 재분화 식물체에서보다는 다소 낮게 나타난 결과이다. 또한 재분화식물체 초본(Aversano et al., 2009; Liu et al., 2011) 및 목본의 결과(Vijayan et al., 2015)보다는 낮은 결과이다.
Table 3.
Genetic similarity matrix of 21 accessions derived from in vitro organogenesis in Imperata cylindrica 'Rubra' revealed by ISSR analysisz
| No.Z | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 1 | 1.000 | ||||||||||||||||||||
| 2 | 1.000 | 1.000 | |||||||||||||||||||
| 3 | 0.986 | 0.986 | 1.000 | ||||||||||||||||||
| 4 | 0.986 | 0.986 | 0.973 | 1.000 | |||||||||||||||||
| 5 | 0.946 | 0.946 | 0.959 | 0.959 | 1.000 | ||||||||||||||||
| 6 | 0.973 | 0.973 | 0.986 | 0.986 | 0.973 | 1.000 | |||||||||||||||
| 7 | 0.944 | 0.944 | 0.958 | 0.932 | 0.919 | 0.945 | 1.000 | ||||||||||||||
| 8 | 0.917 | 0.917 | 0.931 | 0.904 | 0.892 | 0.918 | 0.971 | 1.000 | |||||||||||||
| 9 | 0.944 | 0.944 | 0.958 | 0.932 | 0.919 | 0.945 | 1.000 | 0.971 | 1.000 | ||||||||||||
| 10 | 0.880 | 0.880 | 0.868 | 0.868 | 0.833 | 0.857 | 0.904 | 0.903 | 0.904 | 1.000 | |||||||||||
| 11 | 0.855 | 0.855 | 0.844 | 0.844 | 0.810 | 0.833 | 0.878 | 0.877 | 0.878 | 0.972 | 1.000 | ||||||||||
| 12 | 0.842 | 0.842 | 0.831 | 0.831 | 0.797 | 0.821 | 0.865 | 0.863 | 0.865 | 0.958 | 0.986 | 1.000 | |||||||||
| 13 | 0.788 | 0.788 | 0.778 | 0.778 | 0.747 | 0.768 | 0.785 | 0.782 | 0.785 | 0.844 | 0.868 | 0.880 | 1.000 | ||||||||
| 14 | 0.795 | 0.795 | 0.785 | 0.785 | 0.753 | 0.775 | 0.792 | 0.789 | 0.792 | 0.829 | 0.853 | 0.865 | 0.958 | 1.000 | |||||||
| 15 | 0.795 | 0.795 | 0.785 | 0.785 | 0.753 | 0.775 | 0.792 | 0.789 | 0.792 | 0.829 | 0.853 | 0.865 | 0.958 | 1.000 | 1.000 | ||||||
| 16 | 0.808 | 0.808 | 0.797 | 0.797 | 0.765 | 0.788 | 0.805 | 0.803 | 0.805 | 0.842 | 0.867 | 0.878 | 0.919 | 0.931 | 0.931 | 1.000 | |||||
| 17 | 0.808 | 0.808 | 0.797 | 0.797 | 0.765 | 0.788 | 0.805 | 0.803 | 0.805 | 0.842 | 0.867 | 0.878 | 0.919 | 0.931 | 0.931 | 1.000 | 1.000 | ||||
| 18 | 0.808 | 0.808 | 0.797 | 0.797 | 0.765 | 0.788 | 0.805 | 0.803 | 0.805 | 0.842 | 0.867 | 0.878 | 0.919 | 0.931 | 0.931 | 1.000 | 1.000 | 1.000 | |||
| 19 | 0.805 | 0.805 | 0.795 | 0.795 | 0.763 | 0.785 | 0.803 | 0.800 | 0.803 | 0.865 | 0.890 | 0.903 | 0.918 | 0.930 | 0.930 | 0.971 | 0.971 | 0.971 | 1.000 | ||
| 20 | 0.818 | 0.818 | 0.808 | 0.808 | 0.775 | 0.797 | 0.816 | 0.813 | 0.816 | 0.878 | 0.904 | 0.917 | 0.932 | 0.944 | 0.944 | 0.958 | 0.958 | 0.958 | 0.986 | 1.000 | |
| 21 | 0.805 | 0.805 | 0.795 | 0.795 | 0.763 | 0.785 | 0.803 | 0.800 | 0.803 | 0.865 | 0.890 | 0.903 | 0.918 | 0.930 | 0.930 | 0.971 | 0.971 | 0.971 | 1.000 | 0.986 | 1.000 |
zGenetic similarity was calculated on the basis of Jaccard coefficients. 1~21: Accessions numbers shown in Table 1.
기관분화 단계별 재분화체의 군집분석
ISSR 마커 밴드 양상을 기반으로 평균연결법(Average linkage method)으로 군집 분석한 결과(Fig. 5), 모든 개체는 유사도 지수 0.809~1.000 내에 분포하였다. 유전적 유사도 지수 0.809에서 2개 그룹으로 유집되었으며, 모식물체(1~3번)와 실내재배 (4~6번), 노지재배 재분화 녹색 식물체(7~9번)가 함께 그룹화되었고, 기내배양 중인 홍띠 식물체(10~12번), 녹색 신초(13~15번), 적색신초 발생 캘러스(16~18번), 캘러스(19~21번)가 속한 그룹은 유전적 유사도 지수 0.869에서 두 개의 그룹으로 세분화되었다.
이상에서와 같이 재분화체 중 기내식물체가 독립적으로 유집된 것은 생장조절물질 처리나 특수한 기내환경(Bednarek and Renata, 2020; Garcia et al., 2019; Goh et al., 2011; Ramakrishnan et al., 2014) 등에 따른 변이의 축적에 기인한 것으로 사료된다. 또한 가지과(Aversano et al., 2009), 옥수수(Ramakrishnan et al., 2014), Innula 식물체(Amin et al., 2018) 기내배양체의 ISSR분석 시 모식물체와 재분화체가 같은 그룹으로 유집된 결과와는 다른 결과이다.
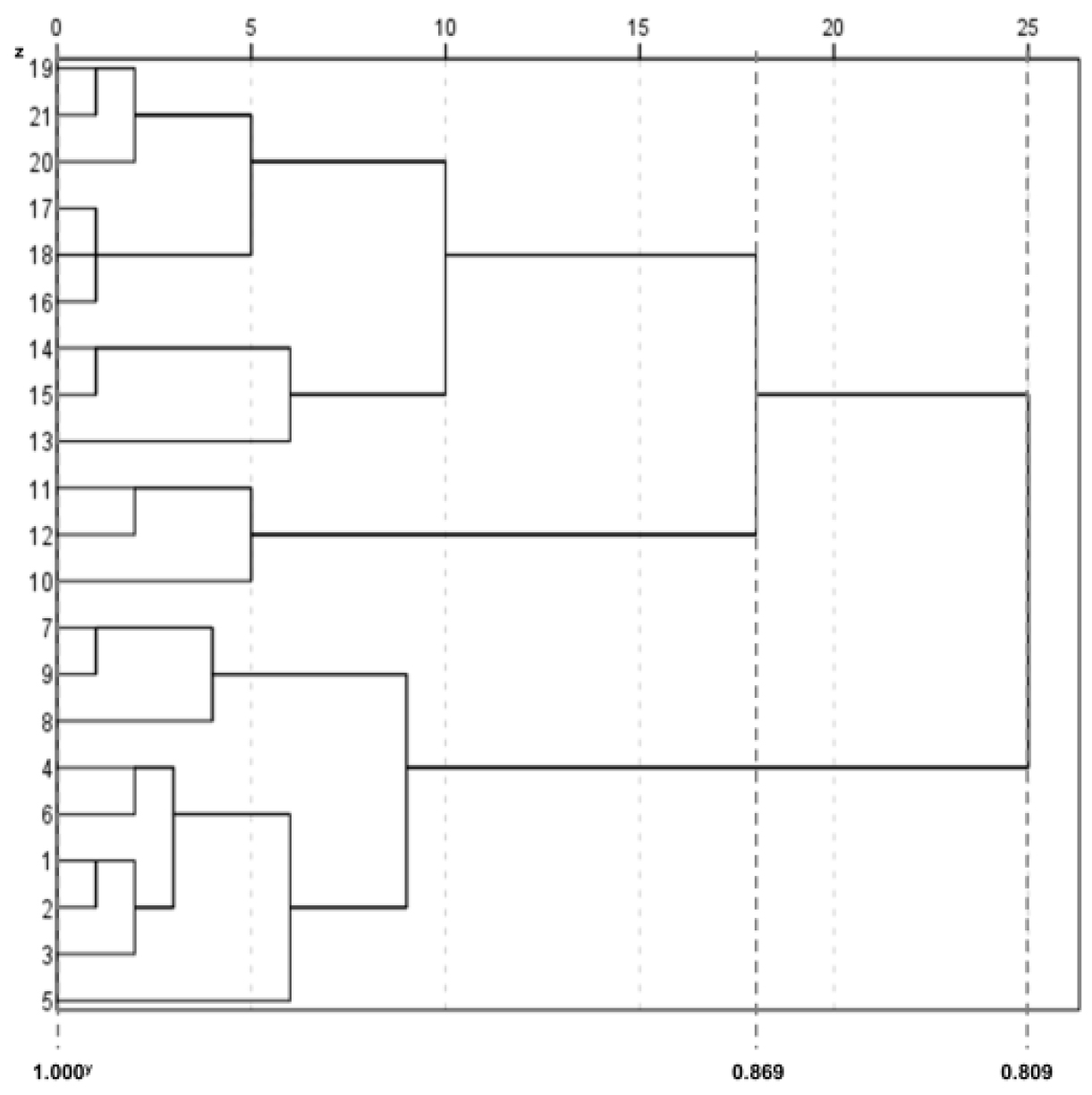
Fig. 5.
Dendrogram illustrating genetic relationships among 21 accessions of in vitro cultured Imperata cylindrica ‘Rubra’ generated by cluster analysis with average linkage method. z1-3: Mother plants, 4-6: Regenerants cultivated at culture room, 7-9: Regenerants cultivated at field condition, 10-12: in vitro regenerants of red plant, 13-15: Calli with green shoots, 16-18: Calli with red shoots, 19-21: Calli, yGenetic distance.
적 요
화본과 식물 홍띠(Imperata cylindrica ‘Rubra’)의 기관분화 관련 유전자의 동태와 기내재생체의 유전적 안정성을 조사하고자 기관분화 단계별 재분화체를 작성하여 기관분화 관련 유전자 발현과 ISSR 마커 기반 변이성을 조사하였다. 5종류 총 15개체의 기관분화 단계별 재분화체에서 캘러스 발생 유전자인 FIE는 모식물체 1번을 제외한 14개의 식물체에서 모두 발현되었으며, 뿌리 발생 유전자인 WOX11도 15개의 모든 단계별 재분화체에서 발현하였다. 체세포 발생 유전자인 LEC1B는 15개 식물체에서 모두 발현하였으나 비교적 약하게 발현하였다. 7종류 총 21개체의 기관분화 단계별 재분화체 및 재분화식물체에 대하여 ISSR 분석한 결과, 유전적 다형성은 기관분화 단계별 재분화체 및 순화 재분화체(실내포트 재배식물체 4.1%, 노지 재배식물체 4.3%, 기내배양 홍띠(적색)식물체 4.2%, 녹색신초 발생 캘러스 5.6%, 캘러스 1.4%)에서 대조구인 모식물체(1.4%)와 같거나 높게 나타났다. 또한, 유전적 유사도 지수는 0.747~1.0 사이에 분포하며, 평균 0.868로 나타났다. 군집분석 결과 유전적 유사도 지수 0.809에서 기외 식물체(모식물체, 실내재배 및 노지재배 재분화 녹색 식물체)와 기내식물체 및 재분화 과정상의 분화체(기내배양 중인 홍띠 식물체, 녹색 신초, 적색신초 발생 캘러스, 캘러스)는 독립적인 2개 그룹으로 유집되었다. 이상의 결과는 화본과 식물의 기내배양 시 재분화 과정에서 일어나는 일련의 유전학적 기초자료를 제공해 준다.




